+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1ho6 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
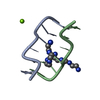
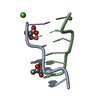
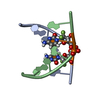
| タイトル | CHIMERIC ARABINONUCLEIC ACID (ANA) HAIRPIN WITH ANA/RNA HYBRID STEM | ||||||||||||||||||
 要素 要素 | DNA/RNA (5'-R(* キーワード キーワードDNA / RNA / Arabinonucleic acid / Hairpin | 機能・相同性 | DNA/RNA hybrid / DNA/RNA hybrid (> 10) |  機能・相同性情報 機能・相同性情報手法 | 溶液NMR / simulated annealing |  データ登録者 データ登録者Denisov, A.Y. / Gehring, K. |  引用 引用 ジャーナル: Nucleic Acids Res. / 年: 2001 ジャーナル: Nucleic Acids Res. / 年: 2001タイトル: Solution structure of an arabinonucleic acid (ANA)/RNA duplex in a chimeric hairpin: comparison with 2'-fluoro-ANA/RNA and DNA/RNA hybrids. 著者: Denisov, A.Y. / Noronha, A.M. / Wilds, C.J. / Trempe, J.F. / Pon, R.T. / Gehring, K. / Damha, M.J. #1:  ジャーナル: Nucleosides and Nucleotides / 年: 2001 ジャーナル: Nucleosides and Nucleotides / 年: 2001タイトル: Properties of Arabinonucleic Acids (ANA & 2'F-ANA): Implications for the Design of Antisense Therapeutics that Invoke RNase H Cleavage of RNA 著者: Damha, M.J. / Noronha, A.M. / Wilds, C.J. / Trempe, J.F. / Denisov, A.Y. / Pon, R.T. / Gehring, K. 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1ho6.cif.gz 1ho6.cif.gz | 83.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1ho6.ent.gz pdb1ho6.ent.gz | 51.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1ho6.json.gz 1ho6.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1ho6_validation.pdf.gz 1ho6_validation.pdf.gz | 312.9 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1ho6_full_validation.pdf.gz 1ho6_full_validation.pdf.gz | 361.4 KB | 表示 | |
| XML形式データ |  1ho6_validation.xml.gz 1ho6_validation.xml.gz | 6.7 KB | 表示 | |
| CIF形式データ |  1ho6_validation.cif.gz 1ho6_validation.cif.gz | 8.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ho/1ho6 https://data.pdbj.org/pub/pdb/validation_reports/ho/1ho6 ftp://data.pdbj.org/pub/pdb/validation_reports/ho/1ho6 ftp://data.pdbj.org/pub/pdb/validation_reports/ho/1ho6 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 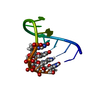
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: DNA/RNAハイブリッド | 分子量: 3768.350 Da / 分子数: 1 / 由来タイプ: 合成 / 詳細: Solid phase synthesis, phosphoramidite chemistry |
|---|
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
| ||||||||||||
| NMR実験の詳細 | Text: This structure was determined using standard 2D homo- and heteronuclear techniques: NOESY, DQF-COSY, TOCSY, H,C-HMQC and H,P-HetCOSY |
- 試料調製
試料調製
| 詳細 |
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 試料状態 | pH: 7 / 圧: 1 atm / 温度: 288 K | |||||||||
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
-NMR測定
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M |
|---|---|
| 放射波長 | 相対比: 1 |
| NMRスペクトロメーター | タイプ: Bruker DRX / 製造業者: Bruker / モデル: DRX / 磁場強度: 500 MHz |
- 解析
解析
| NMR software |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: simulated annealing / ソフトェア番号: 1 詳細: Starting A- and B-type stem hairpin structures with randomly organized loops were refined by 14 ps restrained molecular dynamics (300-1000 K) with final enegry minimization | ||||||||||||
| 代表構造 | 選択基準: the hairpin stem structure closest to the average | ||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: all calculated structures submitted 計算したコンフォーマーの数: 10 / 登録したコンフォーマーの数: 10 |
 ムービー
ムービー コントローラー
コントローラー







 PDBj
PDBj


 X-PLOR
X-PLOR