+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1gmm | ||||||
|---|---|---|---|---|---|---|---|
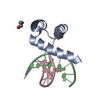
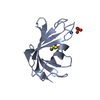
| タイトル | Carbohydrate binding module CBM6 from xylanase U Clostridium thermocellum | ||||||
 要素 要素 | CBM6 | ||||||
 キーワード キーワード | XYLANASE / CARBOHYDRATE BINDING MODULE / CBM FAMILY 6 / XYLAN BINDING | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報hydrolase activity, acting on carbon-nitrogen (but not peptide) bonds / endo-1,4-beta-xylanase activity / endo-1,4-beta-xylanase / xylan catabolic process / carbohydrate binding / metal ion binding 類似検索 - 分子機能 | ||||||
| 生物種 |  CLOSTRIDIUM THERMOCELLUM (バクテリア) CLOSTRIDIUM THERMOCELLUM (バクテリア) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  多波長異常分散 / 解像度: 2 Å 多波長異常分散 / 解像度: 2 Å | ||||||
 データ登録者 データ登録者 | Czjzek, M. / Mosbah, A. / Bolam, D. / Allouch, J. / Zamboni, V. / Henrissat, B. / Gilbert, H.J. | ||||||
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2001 ジャーナル: J.Biol.Chem. / 年: 2001タイトル: The Location of the Ligand-Binding Site of Carbohydrate-Binding Modules that Have Evolved from a Common Sequence is not Conserved. 著者: Czjzek, M. / Bolam, D. / Mosbah, A. / Allouch, J. / Fontes, C.M. / Ferreira, L.M. / Bornet, O. / Zamboni, V. / Darbon, H. / Smith, N.L. / Black, G.W. / Henrissat, B. / Gilbert, H.J. #1: ジャーナル: Biochem.J. / 年: 1999 タイトル: Homologous Xylanases from Clostridium Thermocellum: Evidence for Bi-Functional Activity, Synergism between Xylanase Catalytic Modules and the Presence of Xylan-Binding Domains in Enzyme Complexes 著者: Fernandes, A. / Fontes, C.M.G.A. / Gilbert, H.J. / Hazelwood, G.P. / Fernandes, T.H. / Ferreira, L.M.A. / Gilbert, H.J. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1gmm.cif.gz 1gmm.cif.gz | 42.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1gmm.ent.gz pdb1gmm.ent.gz | 29.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1gmm.json.gz 1gmm.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1gmm_validation.pdf.gz 1gmm_validation.pdf.gz | 433.5 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1gmm_full_validation.pdf.gz 1gmm_full_validation.pdf.gz | 435.1 KB | 表示 | |
| XML形式データ |  1gmm_validation.xml.gz 1gmm_validation.xml.gz | 9.7 KB | 表示 | |
| CIF形式データ |  1gmm_validation.cif.gz 1gmm_validation.cif.gz | 13.3 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/gm/1gmm https://data.pdbj.org/pub/pdb/validation_reports/gm/1gmm ftp://data.pdbj.org/pub/pdb/validation_reports/gm/1gmm ftp://data.pdbj.org/pub/pdb/validation_reports/gm/1gmm | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 14115.347 Da / 分子数: 1 / 断片: XYLAN BINDING MODULE (DOMAIN), RESIDUE 248-380 / 由来タイプ: 組換発現 由来: (組換発現)  CLOSTRIDIUM THERMOCELLUM (バクテリア) CLOSTRIDIUM THERMOCELLUM (バクテリア)株: F1 / 発現宿主:  |
|---|---|
| #2: 化合物 | ChemComp-SO4 / |
| #3: 化合物 | ChemComp-NA / |
| #4: 化合物 | ChemComp-CA / |
| #5: 水 | ChemComp-HOH / |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.73 Å3/Da / 溶媒含有率: 55 % | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | pH: 5.5 詳細: 30% PEG 4000, 100MM NA-CITRATE PH 5.5, 100MM (NH4)2SO4 | ||||||||||||||||||||||||||||||||||||
| 結晶化 | *PLUS 温度: 4 ℃ / pH: 5 / 手法: 蒸気拡散法, ハンギングドロップ法 | ||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 100 K | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ESRF ESRF  / ビームライン: ID14-4 / 波長: 0.9793, 0.9795, 0.9465, 0.933 / ビームライン: ID14-4 / 波長: 0.9793, 0.9795, 0.9465, 0.933 | |||||||||||||||
| 検出器 | 検出器: CCD / 日付: 2000年12月15日 / 詳細: MIRRORS | |||||||||||||||
| 放射 | プロトコル: MAD / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | |||||||||||||||
| 放射波長 |
| |||||||||||||||
| 反射 | 解像度: 2→39.22 Å / Num. obs: 11981 / % possible obs: 99.3 % / Observed criterion σ(I): 0.01 / 冗長度: 6.6 % / Rmerge(I) obs: 0.05 / Net I/σ(I): 9.3 | |||||||||||||||
| 反射 シェル | 解像度: 2→2.11 Å / 冗長度: 6.2 % / Rmerge(I) obs: 0.204 / Mean I/σ(I) obs: 1.2 / % possible all: 98.2 | |||||||||||||||
| 反射 | *PLUS Rmerge(I) obs: 0.055 | |||||||||||||||
| 反射 シェル | *PLUS % possible obs: 98.2 % |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  多波長異常分散 / 解像度: 2→39 Å / SU B: 3.345 / SU ML: 0.095 / 交差検証法: THROUGHOUT / σ(F): 0.01 / ESU R Free: 0.142 多波長異常分散 / 解像度: 2→39 Å / SU B: 3.345 / SU ML: 0.095 / 交差検証法: THROUGHOUT / σ(F): 0.01 / ESU R Free: 0.142 詳細: TLS REFINEMENT WAS PERFORMED ON 6 GROUPS. FOUR RESIDUES, THR A 65, THR A 67, SER A 99 AND THR A 103 HAVE BEEN MODELED WITH MULTIPLE ALTERNATE CONFORMATIONS.
| ||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2→39 Å
| ||||||||||||||||||||
| 精密化 | *PLUS 最高解像度: 2.1 Å / 最低解像度: 32 Å / Rfactor obs: 0.2 / Rfactor Rfree: 0.216 / Rfactor Rwork: 0.2 | ||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||
| 原子変位パラメータ | *PLUS | ||||||||||||||||||||
| 拘束条件 | *PLUS
|
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj








