+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1ffj | ||||||
|---|---|---|---|---|---|---|---|
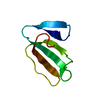
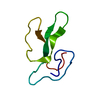
| タイトル | NMR STRUCTURE OF CARDIOTOXIN IN DPC-MICELLE | ||||||
 要素 要素 | CYTOTOXIN 2 | ||||||
 キーワード キーワード | TOXIN / all-beta sheet protein / membrane perturbation | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報other organism cell membrane / toxin activity / killing of cells of another organism / extracellular region / membrane 類似検索 - 分子機能 | ||||||
| 生物種 |  Naja oxiana (コブラ) Naja oxiana (コブラ) | ||||||
| 手法 | 溶液NMR / torsion angle dynamics | ||||||
 データ登録者 データ登録者 | Dubovskii, P.V. / Dementieva, D.V. / Bocharov, E.V. / Utkin, Y.N. / Arseniev, A.S. | ||||||
 引用 引用 |  ジャーナル: J.Mol.Biol. / 年: 2001 ジャーナル: J.Mol.Biol. / 年: 2001タイトル: Membrane binding motif of the P-type cardiotoxin. 著者: Dubovskii, P.V. / Dementieva, D.V. / Bocharov, E.V. / Utkin, Y.N. / Arseniev, A.S. #1:  ジャーナル: Eur.J.Biochem. / 年: 1999 ジャーナル: Eur.J.Biochem. / 年: 1999タイトル: Two forms of Cytotoxin II (cardiotoxin) from Naja naja oxiana in Aqueous Solution. Spatial Structures with Tightly Bound Water Molecules 著者: Dementieva, D.V. / Bocharov, E.V. / Arseniev, A.S. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1ffj.cif.gz 1ffj.cif.gz | 353.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1ffj.ent.gz pdb1ffj.ent.gz | 294.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1ffj.json.gz 1ffj.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ff/1ffj https://data.pdbj.org/pub/pdb/validation_reports/ff/1ffj ftp://data.pdbj.org/pub/pdb/validation_reports/ff/1ffj ftp://data.pdbj.org/pub/pdb/validation_reports/ff/1ffj | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ | |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 | 分子量: 6648.238 Da / 分子数: 1 / 断片: 1 / 由来タイプ: 天然 / 由来: (天然)  Naja oxiana (コブラ) / Secretion: VENOM / 参照: UniProt: P01441 Naja oxiana (コブラ) / Secretion: VENOM / 参照: UniProt: P01441 |
|---|---|
| #2: 水 | ChemComp-HOH / |
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
| ||||||||||||||||||||||||||||||||||||||||||||
| NMR実験の詳細 | Text: This structure was determined using standard 2D homonuclear techniques. Sites of tightly bound water molecules were determined as in Dementieva et al., Eur.J.Biochem.1999,263,152-162. |
- 試料調製
試料調製
| 詳細 |
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料状態 |
| |||||||||||||||
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
-NMR測定
| NMRスペクトロメーター | タイプ: Varian UNITY / 製造業者: Varian / モデル: UNITY / 磁場強度: 600 MHz |
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: torsion angle dynamics / ソフトェア番号: 1 詳細: structures are based on a total of 368 NOE-derived constraints, 154 dihedral angle restraints, 248 distance restraints from hydrogen bonds and disulfides | ||||||||||||||||||||
| 代表構造 | 選択基準: fewest violations | ||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: target function / 計算したコンフォーマーの数: 220 / 登録したコンフォーマーの数: 20 |
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj


