+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1e0g | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
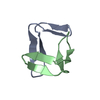
| タイトル | LYSM Domain from E.coli MLTD | |||||||||
 要素 要素 | Membrane-bound lytic murein transglycosylase D | |||||||||
 キーワード キーワード | HYDROLASE / CELL WALL / GLYCOSIDASE / LIPOPROTEIN / OUTER MEMBRANE / MULTIGENE FAMILY | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報lytic endotransglycosylase activity / : / peptidoglycan lytic transglycosylase activity / peptidoglycan metabolic process / cell wall organization / lyase activity / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 溶液NMR / SA | |||||||||
 データ登録者 データ登録者 | Bateman, A. / Bycroft, M. | |||||||||
 引用 引用 |  ジャーナル: J.Mol.Biol. / 年: 2000 ジャーナル: J.Mol.Biol. / 年: 2000タイトル: The Structure of a Lysm Domain from E.Coli Membrane Bound Lytic Murein Transglycosylase D (Mltd) 著者: Bateman, A. / Bycroft, M. #1: ジャーナル: Can.J.Microbiol. / 年: 1994 タイトル: Cloning, Molecular Characterization, and Expression of the Genes Encoding the Lytic Functions of Lactococcal Bacteriophage-Phi-Lc3; a Dual Lysis System of Modular Design 著者: Birkeland, N.K. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1e0g.cif.gz 1e0g.cif.gz | 304.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1e0g.ent.gz pdb1e0g.ent.gz | 252.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1e0g.json.gz 1e0g.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1e0g_validation.pdf.gz 1e0g_validation.pdf.gz | 350.6 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1e0g_full_validation.pdf.gz 1e0g_full_validation.pdf.gz | 479.2 KB | 表示 | |
| XML形式データ |  1e0g_validation.xml.gz 1e0g_validation.xml.gz | 30.8 KB | 表示 | |
| CIF形式データ |  1e0g_validation.cif.gz 1e0g_validation.cif.gz | 46.7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/e0/1e0g https://data.pdbj.org/pub/pdb/validation_reports/e0/1e0g ftp://data.pdbj.org/pub/pdb/validation_reports/e0/1e0g ftp://data.pdbj.org/pub/pdb/validation_reports/e0/1e0g | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質・ペプチド | 分子量: 5442.219 Da / 分子数: 1 / 断片: LYSM DOMAIN / 由来タイプ: 合成 / 由来: (合成)  参照: UniProt: A0A2Z2J5F1, UniProt: P0AEZ7*PLUS, 付加脱離酵素(リアーゼ); 炭素-酸素リアーゼ類; 多糖に作用する |
|---|
-実験情報
-実験
| 実験 | 手法: 溶液NMR |
|---|---|
| NMR実験の詳細 | Text: THE STRUCTURE WAS DETERMINED USING TRIPLE-RESONANCE NMR SPECTROSCOPY ON 13C, 15N-LABELED MTLD LYSM DOMAIN |
- 試料調製
試料調製
| 試料状態 | pH: 6.8 / 温度: 310 K |
|---|---|
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
-NMR測定
| NMRスペクトロメーター | タイプ: Bruker AMX / 製造業者: Bruker / モデル: AMX / 磁場強度: 500 MHz |
|---|
- 解析
解析
| NMR software |
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: SA / ソフトェア番号: 1 / 詳細: SA FROM RANDOM COIL STARTING STRUCTURES | |||||||||
| NMRアンサンブル | コンフォーマー選択の基準: LEAST RESTRAINT VIOLATION 計算したコンフォーマーの数: 100 / 登録したコンフォーマーの数: 20 |
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj





 X-PLOR
X-PLOR