+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1dcd | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | DESULFOREDOXIN COMPLEXED WITH CD2+ | ||||||
 要素 要素 | PROTEIN (DESULFOREDOXIN) | ||||||
 キーワード キーワード | ELECTRON TRANSPORT / RUBREDOXIN TYPE PROTEIN | ||||||
| 機能・相同性 | Desulforedoxin / Desulfoferrodoxin, N-terminal domain / Desulfoferrodoxin, N-terminal domain superfamily / Desulfoferrodoxin, N-terminal domain / iron ion binding / : / Desulforedoxin 機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  Desulfovibrio gigas (バクテリア) Desulfovibrio gigas (バクテリア) | ||||||
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 2 Å 分子置換 / 解像度: 2 Å | ||||||
 データ登録者 データ登録者 | Archer, M. / Carvalho, A.L. / Teixeira, S. / Romao, M.J. | ||||||
 引用 引用 |  ジャーナル: Protein Sci. / 年: 1999 ジャーナル: Protein Sci. / 年: 1999タイトル: Structural studies by X-ray diffraction on metal substituted desulforedoxin, a rubredoxin-type protein. 著者: Archer, M. / Carvalho, A.L. / Teixeira, S. / Moura, I. / Moura, J.J. / Rusnak, F. / Romao, M.J. #1:  ジャーナル: J.Mol.Biol. / 年: 1995 ジャーナル: J.Mol.Biol. / 年: 1995タイトル: Crystal structure of desulforedoxin from Desulfovibrio gigas determined at 1.8 A resolution: a novel non-heme iron protein structure. 著者: Archer, M. / Huber, R. / Tavares, P. / Moura, I. / Moura, J.J. / Carrondo, M.A. / Sieker, L.C. / LeGall, J. / Romao, M.J. | ||||||
| 履歴 |
|
- 構造の表示
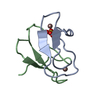
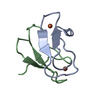
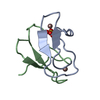
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1dcd.cif.gz 1dcd.cif.gz | 25.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1dcd.ent.gz pdb1dcd.ent.gz | 16.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1dcd.json.gz 1dcd.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1dcd_validation.pdf.gz 1dcd_validation.pdf.gz | 371.6 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1dcd_full_validation.pdf.gz 1dcd_full_validation.pdf.gz | 371 KB | 表示 | |
| XML形式データ |  1dcd_validation.xml.gz 1dcd_validation.xml.gz | 2.8 KB | 表示 | |
| CIF形式データ |  1dcd_validation.cif.gz 1dcd_validation.cif.gz | 4 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/dc/1dcd https://data.pdbj.org/pub/pdb/validation_reports/dc/1dcd ftp://data.pdbj.org/pub/pdb/validation_reports/dc/1dcd ftp://data.pdbj.org/pub/pdb/validation_reports/dc/1dcd | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||
| 単位格子 |
| ||||||||||
| Components on special symmetry positions |
| ||||||||||
| 非結晶学的対称性 (NCS) | NCS oper: (Code: given Matrix: (-0.939027, -0.226955, -0.258303), ベクター: |
- 要素
要素
| #1: タンパク質・ペプチド | 分子量: 3807.350 Da / 分子数: 2 / 由来タイプ: 組換発現 由来: (組換発現)  Desulfovibrio gigas (バクテリア) Desulfovibrio gigas (バクテリア)細胞内の位置: CYTOPLASM / 細胞内の位置 (発現宿主): CYTOPLASM / 遺伝子 (発現宿主): DSR / 発現宿主:  #2: 化合物 | #3: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 1.9 Å3/Da / 溶媒含有率: 35 % | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | pH: 5 詳細: 30% ETHANOL, 0.1 M SODIUM ACETATE PH 5, 0.2 M CACL2, pH 5.0 | ||||||||||||||||||||||||||||||||||||
| 結晶化 | *PLUS pH: 7.6 / 手法: 蒸気拡散法 | ||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 277 K |
|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: RIGAKU / 波長: 1.5418 回転陽極 / タイプ: RIGAKU / 波長: 1.5418 |
| 検出器 | タイプ: MARRESEARCH / 検出器: IMAGE PLATE |
| 放射 | モノクロメーター: NI FILTER / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.5418 Å / 相対比: 1 |
| 反射 | 解像度: 1.98→14.8 Å / Num. all: 4403 / Num. obs: 4403 / % possible obs: 97.5 % / Observed criterion σ(I): 0 / 冗長度: 6.4 % / Rsym value: 0.115 / Net I/σ(I): 8 |
| 反射 シェル | 解像度: 1.98→2.05 Å / Mean I/σ(I) obs: 2.5 / Rsym value: 0.432 / % possible all: 96.4 |
| 反射 | *PLUS Num. measured all: 28142 / Rmerge(I) obs: 0.115 |
| 反射 シェル | *PLUS % possible obs: 96.4 % / Rmerge(I) obs: 0.359 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ENTRY 1DXG 解像度: 2→14.8 Å / Num. parameters: 2325 / Num. restraintsaints: 2656 / σ(F): 0 / 立体化学のターゲット値: ENGH & HUBER 詳細: GEOMETRIC RESTRAINTS WERE APPLIED TO THE METAL CENTER (DFIX CD S 2.55 A)
| |||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: MOEWS & KRETSINGER, J.MOL.BIOL.91(1973)201-228 | |||||||||||||||||||||||||||||||||
| Refine analyze | Num. disordered residues: 2 / Occupancy sum hydrogen: 0 / Occupancy sum non hydrogen: 576 | |||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2→14.8 Å
| |||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj


