+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1cxr | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | AUTOMATED 2D NOESY ASSIGNMENT AND STRUCTURE CALCULATION OF CRAMBIN(S22/I25) WITH SELF-CORRECTING DISTANCE GEOMETRY BASED NOAH/DIAMOD PROGRAMS | ||||||
 要素 要素 | CRAMBIN | ||||||
 キーワード キーワード | PLANT PROTEIN / CRAMBIN / CRAMBE ABYSSINICA / PLANT SEED PROTEIN FEB. 20 / 1997 | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  Crambe hispanica subsp. abyssinica (植物) Crambe hispanica subsp. abyssinica (植物) | ||||||
| 手法 | 溶液NMR / SELF-CORRECTING DISTANCE GEOMETRY | ||||||
 データ登録者 データ登録者 | Xu, Y. / Wu, J. / Gorenstein, D. / Braun, W. | ||||||
 引用 引用 |  ジャーナル: J.Magn.Reson. / 年: 1999 ジャーナル: J.Magn.Reson. / 年: 1999タイトル: Automated 2D NOESY assignment and structure calculation of Crambin(S22/I25) with the self-correcting distance geometry based NOAH/DIAMOD programs. 著者: Xu, Y. / Wu, J. / Gorenstein, D. / Braun, W. #1:  ジャーナル: J.Mol.Biol. / 年: 1995 ジャーナル: J.Mol.Biol. / 年: 1995タイトル: Automated Assignment of Simulated, Experimental Noesy Spectra of Proteins by Feedback Filtering and Self-Correcting Distance Geometry 著者: Mumenthaler, C. / Braun, W. #2:  ジャーナル: J.Biomol.NMR / 年: 1997 ジャーナル: J.Biomol.NMR / 年: 1997タイトル: Automated Combined Assignment of Noesy Spectra and Three-Dimensional Protein Structure Determination 著者: Mumenthaler, C. / Guntert, P. / Braun, W. / Wuthrich, K. #3:  ジャーナル: Biological Magnetic Resonance / 年: 1999 ジャーナル: Biological Magnetic Resonance / 年: 1999タイトル: Combined Automated Assignment of NMR Spectra and Calculation of Three- Dimensional Protein Structures 著者: Xu, Y. / Schein, C.H. / Braun, W. #4:  ジャーナル: Biopolymers / 年: 1990 ジャーナル: Biopolymers / 年: 1990タイトル: A Program, Fantom, for Energy Refinement of Polypeptides and Proteins Using a Newton-Raphson Minimizer in the Torsion Angle Space 著者: Schaumann, T.H. / Braun, W. / Wuthrich, K. #5:  ジャーナル: J.Comput.Chem. / 年: 1998 ジャーナル: J.Comput.Chem. / 年: 1998タイトル: Exact and Efficient Analytical Calculation of the Accessible Surface Areas and Their Gradients Macromolecules 著者: Fraczkiewicz, R. / Braun, W. | ||||||
| 履歴 |
|
- 構造の表示
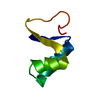
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1cxr.cif.gz 1cxr.cif.gz | 131.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1cxr.ent.gz pdb1cxr.ent.gz | 107.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1cxr.json.gz 1cxr.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1cxr_validation.pdf.gz 1cxr_validation.pdf.gz | 333.1 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1cxr_full_validation.pdf.gz 1cxr_full_validation.pdf.gz | 381.9 KB | 表示 | |
| XML形式データ |  1cxr_validation.xml.gz 1cxr_validation.xml.gz | 8 KB | 表示 | |
| CIF形式データ |  1cxr_validation.cif.gz 1cxr_validation.cif.gz | 12.3 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/cx/1cxr https://data.pdbj.org/pub/pdb/validation_reports/cx/1cxr ftp://data.pdbj.org/pub/pdb/validation_reports/cx/1cxr ftp://data.pdbj.org/pub/pdb/validation_reports/cx/1cxr | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質・ペプチド | 分子量: 4728.410 Da / 分子数: 1 / 断片: ISOFORM OF CRAMBIN (46 RESIDUES) / 変異: S22P, I25L / 由来タイプ: 天然 由来: (天然)  Crambe hispanica subsp. abyssinica (植物) Crambe hispanica subsp. abyssinica (植物)器官: SEED / 生物種: Crambe hispanica / 株: subsp. abyssinica / 参照: UniProt: P01542 |
|---|---|
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
| ||||||||||||||||
| NMR実験の詳細 | Text: THIS STRUCTURE WAS DETERMINED USING STANDARD 2D HOMONUCLEAR TECHNIQUES. |
- 試料調製
試料調製
| 詳細 | 内容: 2.5 MM CRAMBIN(SER/ILE) 0.7 ML OF 75% D6-ACETONE, 20%H2O,5%D2O BUFFER |
|---|---|
| 試料状態 | pH: 6.5 / 圧: 1 atm / 温度: 298 K |
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
-NMR測定
| NMRスペクトロメーター | タイプ: Varian VXRS / 製造業者: Varian / モデル: VXRS / 磁場強度: 600 MHz |
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: SELF-CORRECTING DISTANCE GEOMETRY / ソフトェア番号: 1 / 詳細: SEE REFERENCE: J. MAGN. RESON. 136, P76-85(1999) | ||||||||||||||||
| 代表構造 | 選択基準: closest to the average | ||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: STRUCTURES WITH THE LEAST RESTRAINT VIOLATIONS,STRUCTURES WITH THE LOWEST ENERGY,TARGET FUNCTION 計算したコンフォーマーの数: 40 / 登録したコンフォーマーの数: 10 |
 ムービー
ムービー コントローラー
コントローラー









 PDBj
PDBj
