+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1c0o | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | SOLUTION STRUCTURE OF THE P5 HAIRPIN FROM A GROUP I INTRON COMPLEXED WITH COBALT (III) HEXAMMINE, NMR, 19 CONVERGED STRUCTURES | ||||||||||||||||||||
 要素 要素 | RNA (5'-R(* キーワード キーワードRNA / RIBONUCLEIC ACID / COBALT (III) HEXAMMINE / METAL BINDING / RNA STRUCTURE | 機能・相同性 | COBALT HEXAMMINE(III) / RNA / RNA (> 10) |  機能・相同性情報 機能・相同性情報生物種 |  手法 | 溶液NMR / RESTRAINED MOLECULAR DYNAMICS |  データ登録者 データ登録者Colmenarejo, G. / Tinoco Jr., I. |  引用 引用 ジャーナル: J.Mol.Biol. / 年: 1999 ジャーナル: J.Mol.Biol. / 年: 1999タイトル: Structure and thermodynamics of metal binding in the P5 helix of a group I intron ribozyme. 著者: Colmenarejo, G. / Tinoco Jr., I. 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1c0o.cif.gz 1c0o.cif.gz | 176.2 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1c0o.ent.gz pdb1c0o.ent.gz | 146.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1c0o.json.gz 1c0o.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1c0o_validation.pdf.gz 1c0o_validation.pdf.gz | 335.9 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1c0o_full_validation.pdf.gz 1c0o_full_validation.pdf.gz | 419.1 KB | 表示 | |
| XML形式データ |  1c0o_validation.xml.gz 1c0o_validation.xml.gz | 5 KB | 表示 | |
| CIF形式データ |  1c0o_validation.cif.gz 1c0o_validation.cif.gz | 8.5 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/c0/1c0o https://data.pdbj.org/pub/pdb/validation_reports/c0/1c0o ftp://data.pdbj.org/pub/pdb/validation_reports/c0/1c0o ftp://data.pdbj.org/pub/pdb/validation_reports/c0/1c0o | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 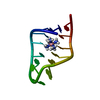
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: RNA鎖 | 分子量: 4471.667 Da / 分子数: 1 / 由来タイプ: 組換発現 詳細: CLOSED BY A UUCG TETRALOOP; COBALT (III) HEXAMMINE BINDS AT THE MAJOR GROOVE OF TWO TANDEM G-U WOBBLE PAIRS IN 5'-GU-3' ORIENTATION 由来: (組換発現)  解説: SYNTHESIZED ENZYMATICALLY IN-VITRO USING T7 RNA POLYMERASE |
|---|---|
| #2: 化合物 | ChemComp-NCO / |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
| ||||||||||||||||||||||||||||||||||||
| NMR実験の詳細 | Text: THE STRUCTURE WAS DETERMINED USING A NON-ISOTOPICALLY LABELLED RNA SAMPLE. ALL 13C EXPERIMENTS WERE DONE USING NATURAL ABUNDANCE. COBALT (III) HEXAMMINE WAS LOCATED USING DOCKING WITH 7 ...Text: THE STRUCTURE WAS DETERMINED USING A NON-ISOTOPICALLY LABELLED RNA SAMPLE. ALL 13C EXPERIMENTS WERE DONE USING NATURAL ABUNDANCE. COBALT (III) HEXAMMINE WAS LOCATED USING DOCKING WITH 7 INTERMOLECULAR NOES OBSERVED IN 2D WATER NOESY EXPERIMENTS. |
- 試料調製
試料調製
| 詳細 | 内容: PHOSPHATE BUFFER, EDTA |
|---|---|
| 試料状態 | イオン強度: 100 mM NACL / pH: 6.4 / 圧: 1 atm / 温度: 298 K |
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
-NMR測定
| NMRスペクトロメーター |
|
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: RESTRAINED MOLECULAR DYNAMICS / ソフトェア番号: 1 詳細: REFINEMENT DETAILS CAN BE FOUND IN THE JRNL CITATION ABOVE | ||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: NOE ENERGY (LEAST RESTRAINT VIOLATIONS), DIHEDRAL ENERGY (LEAST RESTRAINT VIOLATIONS), TOTAL ENERGY 計算したコンフォーマーの数: 40 / 登録したコンフォーマーの数: 19 |
 ムービー
ムービー コントローラー
コントローラー







 PDBj
PDBj






























 X-PLOR
X-PLOR