+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1bar | ||||||
|---|---|---|---|---|---|---|---|
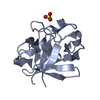
| タイトル | THREE-DIMENSIONAL STRUCTURES OF ACIDIC AND BASIC FIBROBLAST GROWTH FACTORS | ||||||
 要素 要素 | ACIDIC FIBROBLAST GROWTH FACTOR | ||||||
 キーワード キーワード | GROWTH FACTOR | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報FGFR4 ligand binding and activation / FGFR1b ligand binding and activation / FGFR3b ligand binding and activation / FGFR3c ligand binding and activation / FGFR1c ligand binding and activation / FGFR2c ligand binding and activation / FGFR2b ligand binding and activation / Phospholipase C-mediated cascade: FGFR1 / Phospholipase C-mediated cascade; FGFR2 / Phospholipase C-mediated cascade; FGFR3 ...FGFR4 ligand binding and activation / FGFR1b ligand binding and activation / FGFR3b ligand binding and activation / FGFR3c ligand binding and activation / FGFR1c ligand binding and activation / FGFR2c ligand binding and activation / FGFR2b ligand binding and activation / Phospholipase C-mediated cascade: FGFR1 / Phospholipase C-mediated cascade; FGFR2 / Phospholipase C-mediated cascade; FGFR3 / Phospholipase C-mediated cascade; FGFR4 / Downstream signaling of activated FGFR1 / SHC-mediated cascade:FGFR1 / FRS-mediated FGFR1 signaling / SHC-mediated cascade:FGFR2 / FRS-mediated FGFR2 signaling / SHC-mediated cascade:FGFR3 / FRS-mediated FGFR3 signaling / FRS-mediated FGFR4 signaling / SHC-mediated cascade:FGFR4 / PI-3K cascade:FGFR1 / PI-3K cascade:FGFR2 / PI-3K cascade:FGFR3 / PI-3K cascade:FGFR4 / PI3K Cascade / PIP3 activates AKT signaling / RAF/MAP kinase cascade / PI5P, PP2A and IER3 Regulate PI3K/AKT Signaling / Negative regulation of FGFR1 signaling / Negative regulation of FGFR2 signaling / Negative regulation of FGFR3 signaling / Negative regulation of FGFR4 signaling / mesonephric epithelium development / branch elongation involved in ureteric bud branching / regulation of endothelial tube morphogenesis / regulation of endothelial cell chemotaxis to fibroblast growth factor / fibroblast growth factor receptor binding / organ induction / S100 protein binding / positive regulation of sprouting angiogenesis / positive regulation of intracellular signal transduction / positive regulation of cell division / fibroblast growth factor receptor signaling pathway / activation of protein kinase B activity / neurogenesis / extracellular matrix / positive regulation of endothelial cell migration / lung development / regulation of cell migration / epithelial cell proliferation / positive regulation of epithelial cell proliferation / growth factor activity / wound healing / positive regulation of cholesterol biosynthetic process / integrin binding / positive regulation of angiogenesis / heparin binding / cellular response to heat / cell cortex / angiogenesis / positive regulation of ERK1 and ERK2 cascade / positive regulation of MAPK cascade / positive regulation of cell migration / positive regulation of cell population proliferation / positive regulation of transcription by RNA polymerase II / extracellular space / extracellular region / nucleoplasm / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / 解像度: 2.7 Å X線回折 / 解像度: 2.7 Å | ||||||
 データ登録者 データ登録者 | Zhu, X. / Komiya, H. / Chirino, A. / Faham, S. / Fox, G.M. / Arakawa, T. / Hsu, B.T. / Rees, D.C. | ||||||
 引用 引用 |  ジャーナル: Science / 年: 1991 ジャーナル: Science / 年: 1991タイトル: Three-dimensional structures of acidic and basic fibroblast growth factors. 著者: Zhu, X. / Komiya, H. / Chirino, A. / Faham, S. / Fox, G.M. / Arakawa, T. / Hsu, B.T. / Rees, D.C. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1bar.cif.gz 1bar.cif.gz | 63.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1bar.ent.gz pdb1bar.ent.gz | 46.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1bar.json.gz 1bar.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1bar_validation.pdf.gz 1bar_validation.pdf.gz | 375.7 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1bar_full_validation.pdf.gz 1bar_full_validation.pdf.gz | 406.2 KB | 表示 | |
| XML形式データ |  1bar_validation.xml.gz 1bar_validation.xml.gz | 10 KB | 表示 | |
| CIF形式データ |  1bar_validation.cif.gz 1bar_validation.cif.gz | 14.5 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ba/1bar https://data.pdbj.org/pub/pdb/validation_reports/ba/1bar ftp://data.pdbj.org/pub/pdb/validation_reports/ba/1bar ftp://data.pdbj.org/pub/pdb/validation_reports/ba/1bar | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 4 |
| ||||||||
| 5 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 15789.911 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)   #2: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.27 Å3/Da / 溶媒含有率: 62.4 % | |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | *PLUS 手法: 蒸気拡散法 | |||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 放射 | 散乱光タイプ: x-ray |
|---|---|
| 放射波長 | 相対比: 1 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | Rfactor Rwork: 0.177 / Rfactor obs: 0.177 / 最高解像度: 2.7 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 最高解像度: 2.7 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称:  X-PLOR / 分類: refinement X-PLOR / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS 最高解像度: 2.7 Å / Rfactor obs: 0.177 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS タイプ: x_angle_d / Dev ideal: 2.3 |
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj


