+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1a2p | ||||||
|---|---|---|---|---|---|---|---|
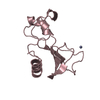
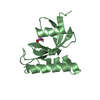
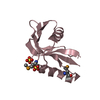
| タイトル | BARNASE WILDTYPE STRUCTURE AT 1.5 ANGSTROMS RESOLUTION | ||||||
 要素 要素 | BARNASE | ||||||
 キーワード キーワード | RIBONUCLEASE / MICROBIAL RIBONUCLEASE / ALPHA/BETA PROTEIN | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報加水分解酵素; エステル加水分解酵素; 3'-リン酸モノエステル産生エンドリボヌクレアーゼ / RNA endonuclease activity / RNA binding / extracellular region 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / 解像度: 1.5 Å シンクロトロン / 解像度: 1.5 Å | ||||||
 データ登録者 データ登録者 | Martin, C. / Richard, V. / Salem, M. / Hartley, R.W. / Mauguen, Y. | ||||||
 引用 引用 |  ジャーナル: Acta Crystallogr.,Sect.D / 年: 1999 ジャーナル: Acta Crystallogr.,Sect.D / 年: 1999タイトル: Refinement and structural analysis of barnase at 1.5 A resolution. 著者: Martin, C. / Richard, V. / Salem, M. / Hartley, R. / Mauguen, Y. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1a2p.cif.gz 1a2p.cif.gz | 159.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1a2p.ent.gz pdb1a2p.ent.gz | 127.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1a2p.json.gz 1a2p.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1a2p_validation.pdf.gz 1a2p_validation.pdf.gz | 424.2 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1a2p_full_validation.pdf.gz 1a2p_full_validation.pdf.gz | 424.6 KB | 表示 | |
| XML形式データ |  1a2p_validation.xml.gz 1a2p_validation.xml.gz | 18.5 KB | 表示 | |
| CIF形式データ |  1a2p_validation.cif.gz 1a2p_validation.cif.gz | 27.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/a2/1a2p https://data.pdbj.org/pub/pdb/validation_reports/a2/1a2p ftp://data.pdbj.org/pub/pdb/validation_reports/a2/1a2p ftp://data.pdbj.org/pub/pdb/validation_reports/a2/1a2p | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||
| 2 | 
| ||||||||||||
| 3 | 
| ||||||||||||
| 単位格子 |
| ||||||||||||
| 非結晶学的対称性 (NCS) | NCS oper:
|
- 要素
要素
| #1: タンパク質 | 分子量: 12398.721 Da / 分子数: 3 / 由来タイプ: 組換発現 由来: (組換発現)  プラスミド: PMJ1002 / 発現宿主:  参照: UniProt: P00648, 加水分解酵素; エステル加水分解酵素; 3'-リン酸モノエステル産生エンドリボヌクレアーゼ #2: 化合物 | #3: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.24 Å3/Da / 溶媒含有率: 45 % | |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | pH: 7.5 / 詳細: pH 7.5 | |||||||||||||||||||||||||
| 結晶 | *PLUS | |||||||||||||||||||||||||
| 結晶化 | *PLUS 手法: 蒸気拡散法, ハンギングドロップ法 | |||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 287 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: LURE シンクロトロン / サイト: LURE  / ビームライン: DW32 / 波長: 0.92 / ビームライン: DW32 / 波長: 0.92 |
| 検出器 | タイプ: MARRESEARCH / 検出器: IMAGE PLATE / 日付: 1993年11月1日 / 詳細: MULTILAYER MIRROR |
| 放射 | モノクロメーター: SI(111) / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.92 Å / 相対比: 1 |
| 反射 | 解像度: 1.5→19.9 Å / Num. obs: 48463 / % possible obs: 96.9 % / 冗長度: 2.5 % / Rsym value: 0.038 / Net I/σ(I): 11.9 |
| 反射 シェル | 解像度: 1.5→1.58 Å / 冗長度: 1.9 % / Mean I/σ(I) obs: 4.7 / Rsym value: 0.158 / % possible all: 94.5 |
| 反射 | *PLUS Rmerge(I) obs: 0.038 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 1.5→8 Å / Num. parameters: 27377 / Num. restraintsaints: 33057 / 交差検証法: FREE R-VALUE / σ(F): 0 / 立体化学のターゲット値: ENGH AND HUBER 詳細: INITIAL POSITIONAL AND B-FACTOR REFINEMENT WAS CARRIED OUT WITH X-PLOR (BRUNGER, 1992) FOR DATA IN THE RESOLUTION RANGE 7.0 - 1.5 ANGSTROMS. AT R-VALUES OF R=0.174 FOR F.GREATER THAN 3 ...詳細: INITIAL POSITIONAL AND B-FACTOR REFINEMENT WAS CARRIED OUT WITH X-PLOR (BRUNGER, 1992) FOR DATA IN THE RESOLUTION RANGE 7.0 - 1.5 ANGSTROMS. AT R-VALUES OF R=0.174 FOR F.GREATER THAN 3 SIGMA(F), THE REFINEMENT WAS CONTINUED WITH THE PROGRAM SHELXL-93. ANISOU RECORDS CONTAIN ANISOTROPIC DISPLACEMENT PARAMETERS U11 U22 U33 U23 U13 U12 (ANGSTROMS**2) MULTIPLIED BY 10000. ISOTROPIC EQUIVALENTS OF ANISOTROPIC TEMPERATURE FACTORS ARE ALSO PRESENTED IN THIS ENTRY.
| |||||||||||||||||||||||||||||||||
| Refine analyze | Num. disordered residues: 10 / Occupancy sum hydrogen: 2417.5 / Occupancy sum non hydrogen: 2972 | |||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.5→8 Å
| |||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj


