+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9401 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CH505 SOSIP.664 trimer in complex with DH522.2 Fab | |||||||||
 マップデータ マップデータ | CH505 SOSIP.664 trimer in complex with DH522.2 Fab | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 28.1 Å | |||||||||
 データ登録者 データ登録者 | Fera D / Harrison SC | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2017 ジャーナル: Nat Commun / 年: 2017タイトル: Initiation of HIV neutralizing B cell lineages with sequential envelope immunizations. 著者: Wilton B Williams / Jinsong Zhang / Chuancang Jiang / Nathan I Nicely / Daniela Fera / Kan Luo / M Anthony Moody / Hua-Xin Liao / S Munir Alam / Thomas B Kepler / Akshaya Ramesh / Kevin Wiehe ...著者: Wilton B Williams / Jinsong Zhang / Chuancang Jiang / Nathan I Nicely / Daniela Fera / Kan Luo / M Anthony Moody / Hua-Xin Liao / S Munir Alam / Thomas B Kepler / Akshaya Ramesh / Kevin Wiehe / James A Holland / Todd Bradley / Nathan Vandergrift / Kevin O Saunders / Robert Parks / Andrew Foulger / Shi-Mao Xia / Mattia Bonsignori / David C Montefiori / Mark Louder / Amanda Eaton / Sampa Santra / Richard Scearce / Laura Sutherland / Amanda Newman / Hilary Bouton-Verville / Cindy Bowman / Howard Bomze / Feng Gao / Dawn J Marshall / John F Whitesides / Xiaoyan Nie / Garnett Kelsoe / Steven G Reed / Christopher B Fox / Kim Clary / Marguerite Koutsoukos / David Franco / John R Mascola / Stephen C Harrison / Barton F Haynes / Laurent Verkoczy /    要旨: A strategy for HIV-1 vaccine development is to define envelope (Env) evolution of broadly neutralizing antibodies (bnAbs) in infection and to recreate those events by vaccination. Here, we report ...A strategy for HIV-1 vaccine development is to define envelope (Env) evolution of broadly neutralizing antibodies (bnAbs) in infection and to recreate those events by vaccination. Here, we report host tolerance mechanisms that limit the development of CD4-binding site (CD4bs), HCDR3-binder bnAbs via sequential HIV-1 Env vaccination. Vaccine-induced macaque CD4bs antibodies neutralize 7% of HIV-1 strains, recognize open Env trimers, and accumulate relatively modest somatic mutations. In naive CD4bs, unmutated common ancestor knock-in mice EnvB cell clones develop anergy and partial deletion at the transitional to mature B cell stage, but become Env upon receptor editing. In comparison with repetitive Env immunizations, sequential Env administration rescue anergic Env (non-edited) precursor B cells. Thus, stepwise immunization initiates CD4bs-bnAb responses, but immune tolerance mechanisms restrict their development, suggesting that sequential immunogen-based vaccine regimens will likely need to incorporate strategies to expand bnAb precursor pools. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9401.map.gz emd_9401.map.gz | 2.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9401-v30.xml emd-9401-v30.xml emd-9401.xml emd-9401.xml | 13.9 KB 13.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_9401.png emd_9401.png | 22.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9401 http://ftp.pdbj.org/pub/emdb/structures/EMD-9401 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9401 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9401 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_9401_validation.pdf.gz emd_9401_validation.pdf.gz | 77.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_9401_full_validation.pdf.gz emd_9401_full_validation.pdf.gz | 76.7 KB | 表示 | |
| XML形式データ |  emd_9401_validation.xml.gz emd_9401_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9401 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9401 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9401 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9401 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9401.map.gz / 形式: CCP4 / 大きさ: 18.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9401.map.gz / 形式: CCP4 / 大きさ: 18.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
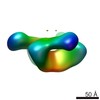
| 注釈 | CH505 SOSIP.664 trimer in complex with DH522.2 Fab | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.13 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : CH505 SOSIP.664 in complex with DH522.2 Fab
| 全体 | 名称: CH505 SOSIP.664 in complex with DH522.2 Fab |
|---|---|
| 要素 |
|
-超分子 #1: CH505 SOSIP.664 in complex with DH522.2 Fab
| 超分子 | 名称: CH505 SOSIP.664 in complex with DH522.2 Fab / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|
-超分子 #2: CH505 SOSIP.664
| 超分子 | 名称: CH505 SOSIP.664 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: DH522.2 Fab
| 超分子 | 名称: DH522.2 Fab / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: CH505 SOSIP.664
| 分子 | 名称: CH505 SOSIP.664 / タイプ: other / ID: 1 / 分類: other |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 配列 | 文字列: AENLWVTVYY GVPVWKEAKT TLFCASDAKA YEKEVHNVWA THACVPTDPN PQEMVLKNVT ENFNMWKNDM VDQMHEDVIS LWDQSLKPCV KLTPLCVTLN CTNATASNSS IIEGMKNCSF NITTELRDKR EKKNALFYKL DIVQLDGNSS QYRLINCNTS VITQACPKVS ...文字列: AENLWVTVYY GVPVWKEAKT TLFCASDAKA YEKEVHNVWA THACVPTDPN PQEMVLKNVT ENFNMWKNDM VDQMHEDVIS LWDQSLKPCV KLTPLCVTLN CTNATASNSS IIEGMKNCSF NITTELRDKR EKKNALFYKL DIVQLDGNSS QYRLINCNTS VITQACPKVS FDPIPIHYCA PAGYAILKCN NKTFTGTGPC NNVSTVQCTH GIKPVVSTQL LLNGSLAEGE IIIRSENITN NVKTIIVHLN ESVKIECTRP NNKTRTSIRI GPGQAFYATG QVIGDIREAY CNINESKWNE TLQRVSKKLK EYFPHKNITF QPSSGGDLEI TTHSFNCGGE FFYCNTSSLF NRTYMANSTD MANSTETNST RTITIHCRIK QIINMWQEVG RAMYAPPIAG NITCISNITG LLLTRDGGKN NTETFRPGGG NMKDNWRSEL YKYKVVKIEP LGVAPTRCKR RVVGRRRRRR AVGIGAVFLG FLGAAGSTMG AASMTLTVQA RNLLSGIVQQ QSNLLRAPEA QQHLLKLTVW GIKQLQARVL AVERYLRDQQ LLGIWGCSGK LICCTNVPWN SSWSNRNLSE IWDNMTWLQW DKEISNYTQI IYGLLEESQN QQEKNEQDLL ALD |
-分子 #2: DH522.2 Fab Heavy Chain
| 分子 | 名称: DH522.2 Fab Heavy Chain / タイプ: other / ID: 2 / 分類: other |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVQLQESGPG LVKPSETLSL TCAVSGGSIG DDYYWNWIRQ SPGKGLEWIG SIYGSFGGTN FNPSLKNRVT ISMDTSNNQV SLKLNSVTAA DTAVYYCARG SHSIVVLFGY YFDYWGQGVL VTVSSASTKG PSVFPLAPSS RSTSESTAAL GCLVKD YFP EPVTVSWNSG ...文字列: QVQLQESGPG LVKPSETLSL TCAVSGGSIG DDYYWNWIRQ SPGKGLEWIG SIYGSFGGTN FNPSLKNRVT ISMDTSNNQV SLKLNSVTAA DTAVYYCARG SHSIVVLFGY YFDYWGQGVL VTVSSASTKG PSVFPLAPSS RSTSESTAAL GCLVKD YFP EPVTVSWNSG SLTSGVHTFP AVLQSSGLYS LSSVVTVPSS SLGTQTYVCN VNHKPSN TK VDKRVEIKT |
-分子 #3: DH522.2 Fab Light Chain
| 分子 | 名称: DH522.2 Fab Light Chain / タイプ: other / ID: 3 / 分類: other |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QSALTQPPSV SKSLGQSVTI SCSGTTNDIG AYNGVS WYQ HHSDTAPRLL IYEVNKRPSG VSDRFSGSKS GNTASLTISG LQAEDEADYY CGSYRSG ST WVFGGGTRLT VLGQPKASPT VTLFPPSSEE LQANKATLVC LISDFYPGVV KVAWKADG S AVNAGVETTT ...文字列: QSALTQPPSV SKSLGQSVTI SCSGTTNDIG AYNGVS WYQ HHSDTAPRLL IYEVNKRPSG VSDRFSGSKS GNTASLTISG LQAEDEADYY CGSYRSG ST WVFGGGTRLT VLGQPKASPT VTLFPPSSEE LQANKATLVC LISDFYPGVV KVAWKADG S AVNAGVETTT PSKQSNNKYA ASSYLSLTSD QWKSHKSYSC QVTHEGSTVE KTVAPAECS |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.01 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||
| 染色 | タイプ: NEGATIVE / 材質: uranyl formate |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 12 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 平均電子線量: 25.0 e/Å2 |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
- 画像解析
画像解析
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 28.1 Å / 解像度の算出法: FSC 0.5 CUT-OFF / 使用した粒子像数: 15269 |
|---|---|
| 初期 角度割当 | タイプ: COMMON LINE |
| 最終 角度割当 | タイプ: COMMON LINE |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)