+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9046 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM of the OmcS nanowires from Geobacter sulfurreducens | ||||||||||||
 マップデータ マップデータ | OmcS nanowire from Geobacter sulfurreducens | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | helical symmetry / cytochrome nanowire / cytochrome c3 / filament / ELECTRON TRANSPORT | ||||||||||||
| 機能・相同性 | : / Multiheme cytochrome superfamily / anaerobic respiration / cell outer membrane / electron transfer activity / cell surface / metal ion binding / C-type cytochrome OmcS 機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |  Geobacter sulfurreducens PCA (バクテリア) / Geobacter sulfurreducens PCA (バクテリア) /  Geobacter sulfurreducens (strain ATCC 51573 / DSM 12127 / PCA) (バクテリア) Geobacter sulfurreducens (strain ATCC 51573 / DSM 12127 / PCA) (バクテリア) | ||||||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.7 Å | ||||||||||||
 データ登録者 データ登録者 | Wang F / Gu Y | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2019 ジャーナル: Cell / 年: 2019タイトル: Structure of Microbial Nanowires Reveals Stacked Hemes that Transport Electrons over Micrometers. 著者: Fengbin Wang / Yangqi Gu / J Patrick O'Brien / Sophia M Yi / Sibel Ebru Yalcin / Vishok Srikanth / Cong Shen / Dennis Vu / Nicole L Ing / Allon I Hochbaum / Edward H Egelman / Nikhil S Malvankar /  要旨: Long-range (>10 μm) transport of electrons along networks of Geobacter sulfurreducens protein filaments, known as microbial nanowires, has been invoked to explain a wide range of globally important ...Long-range (>10 μm) transport of electrons along networks of Geobacter sulfurreducens protein filaments, known as microbial nanowires, has been invoked to explain a wide range of globally important redox phenomena. These nanowires were previously thought to be type IV pili composed of PilA protein. Here, we report a 3.7 Å resolution cryoelectron microscopy structure, which surprisingly reveals that, rather than PilA, G. sulfurreducens nanowires are assembled by micrometer-long polymerization of the hexaheme cytochrome OmcS, with hemes packed within ∼3.5-6 Å of each other. The inter-subunit interfaces show unique structural elements such as inter-subunit parallel-stacked hemes and axial coordination of heme by histidines from neighboring subunits. Wild-type OmcS filaments show 100-fold greater conductivity than other filaments from a ΔomcS strain, highlighting the importance of OmcS to conductivity in these nanowires. This structure explains the remarkable capacity of soil bacteria to transport electrons to remote electron acceptors for respiration and energy sharing. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9046.map.gz emd_9046.map.gz | 10.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9046-v30.xml emd-9046-v30.xml emd-9046.xml emd-9046.xml | 11.7 KB 11.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_9046.png emd_9046.png | 134.6 KB | ||
| Filedesc metadata |  emd-9046.cif.gz emd-9046.cif.gz | 5.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9046 http://ftp.pdbj.org/pub/emdb/structures/EMD-9046 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9046 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9046 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_9046_validation.pdf.gz emd_9046_validation.pdf.gz | 425.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_9046_full_validation.pdf.gz emd_9046_full_validation.pdf.gz | 424.6 KB | 表示 | |
| XML形式データ |  emd_9046_validation.xml.gz emd_9046_validation.xml.gz | 4.6 KB | 表示 | |
| CIF形式データ |  emd_9046_validation.cif.gz emd_9046_validation.cif.gz | 5.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9046 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9046 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9046 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9046 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9046.map.gz / 形式: CCP4 / 大きさ: 14.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9046.map.gz / 形式: CCP4 / 大きさ: 14.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | OmcS nanowire from Geobacter sulfurreducens | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.05 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
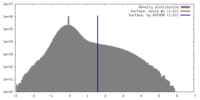
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Filament of OmcS protein
| 全体 | 名称: Filament of OmcS protein |
|---|---|
| 要素 |
|
-超分子 #1: Filament of OmcS protein
| 超分子 | 名称: Filament of OmcS protein / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Geobacter sulfurreducens PCA (バクテリア) Geobacter sulfurreducens PCA (バクテリア) |
-分子 #1: C-type cytochrome OmcS
| 分子 | 名称: C-type cytochrome OmcS / タイプ: protein_or_peptide / ID: 1 / コピー数: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Geobacter sulfurreducens (strain ATCC 51573 / DSM 12127 / PCA) (バクテリア) Geobacter sulfurreducens (strain ATCC 51573 / DSM 12127 / PCA) (バクテリア)株: ATCC 51573 / DSM 12127 / PCA |
| 分子量 | 理論値: 42.986008 KDa |
| 配列 | 文字列: FHSGGVAECE GCHTMHNSLG GAVMNSATAQ FTTGPMLLQG ATQSSSCLNC HQHAGDTGPS SYHISTAEAD MPAGTAPLQM TPGGDFGWV KKTYTWNVRG LNTSEGERKG HNIVAGDYNY VADTTLTTAP GGTYPANQLH CSSCHDPHGK YRRFVDGSIA T TGLPIKNS ...文字列: FHSGGVAECE GCHTMHNSLG GAVMNSATAQ FTTGPMLLQG ATQSSSCLNC HQHAGDTGPS SYHISTAEAD MPAGTAPLQM TPGGDFGWV KKTYTWNVRG LNTSEGERKG HNIVAGDYNY VADTTLTTAP GGTYPANQLH CSSCHDPHGK YRRFVDGSIA T TGLPIKNS GSYQNSNDPT AWGAVGAYRI LGGTGYQPKS LSGSYAFANQ VPAAVAPSTY NRTEATTQTR VAYGQGMSEW CA NCHTDIH NSAYPTNLRH PAGNGAKFGA TIAGLYNSYK KSGDLTGTQA SAYLSLAPFE EGTADYTVLK GHAKIDDTAL TGA DATSNV NCLSCHRAHA SGFDSMTRFN LAYEFTTIAD ASGNSIYGTD PNTSSLQGRS VNEMTAAYYG RTADKFAPYQ RALC NKCHA KD UniProtKB: C-type cytochrome OmcS |
-分子 #2: HEME C
| 分子 | 名称: HEME C / タイプ: ligand / ID: 2 / コピー数: 42 / 式: HEC |
|---|---|
| 分子量 | 理論値: 618.503 Da |
| Chemical component information |  ChemComp-HEC: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 実像数: 1447 / 平均電子線量: 2.3 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 46.7 Å 想定した対称性 - らせんパラメータ - ΔΦ: -83.1 ° 想定した対称性 - らせんパラメータ - 軸対称性: C1 (非対称) アルゴリズム: BACK PROJECTION / 解像度のタイプ: BY AUTHOR / 解像度: 3.7 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: SPIDER / 詳細: Model:Map FSC 0.38 cutoff / 使用した粒子像数: 28293 |
|---|---|
| 初期モデル | モデルのタイプ: OTHER / 詳細: featureless cylinder |
| 最終 角度割当 | タイプ: NOT APPLICABLE |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: BACKBONE TRACE |
|---|---|
| 得られたモデル |  PDB-6ef8: |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)