+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8830 | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structural and functional impacts of ER coactivator sequential recruitment | |||||||||||||||||||||
 マップデータ マップデータ | density map of ERE/ERa%uF061/SRC-3a/p300/CARM1 complex. | |||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報histone H3R17 methyltransferase activity / negative regulation of dendrite development / histone H3R2 methyltransferase activity / protein-arginine omega-N asymmetric methyltransferase activity / type I protein arginine methyltransferase / : / protein methyltransferase activity / regulation of intracellular estrogen receptor signaling pathway / replication fork reversal / protein-arginine N-methyltransferase activity ...histone H3R17 methyltransferase activity / negative regulation of dendrite development / histone H3R2 methyltransferase activity / protein-arginine omega-N asymmetric methyltransferase activity / type I protein arginine methyltransferase / : / protein methyltransferase activity / regulation of intracellular estrogen receptor signaling pathway / replication fork reversal / protein-arginine N-methyltransferase activity / positive regulation of epithelial cell apoptotic process / histone methyltransferase activity / positive regulation of transcription by RNA polymerase I / nuclear replication fork / response to cAMP / positive regulation of fat cell differentiation / : / Regulation of lipid metabolism by PPARalpha / BMAL1:CLOCK,NPAS2 activates circadian expression / Activation of gene expression by SREBF (SREBP) / TP53 Regulates Transcription of Genes Involved in G2 Cell Cycle Arrest / Heme signaling / Transcriptional activation of mitochondrial biogenesis / PPARA activates gene expression / Cytoprotection by HMOX1 / beta-catenin binding / Transcriptional regulation of white adipocyte differentiation / RMTs methylate histone arginines / : / methylation / Estrogen-dependent gene expression / DNA-binding transcription factor binding / transcription coactivator activity / transcription cis-regulatory region binding / chromatin remodeling / positive regulation of cell population proliferation / regulation of DNA-templated transcription / nucleoplasm / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||||||||
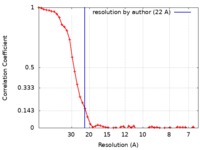
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 22.0 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Yi P / Wang Z / Feng Q / Chou CK / Pintilie G / Shen H / Foulds CE / Fan GZ / Serysheva I / Ludtke S ...Yi P / Wang Z / Feng Q / Chou CK / Pintilie G / Shen H / Foulds CE / Fan GZ / Serysheva I / Ludtke S / Schmid MF / Hung MC / Chiu W / OMalley BW | |||||||||||||||||||||
| 資金援助 |  米国, 6件 米国, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2017 ジャーナル: Mol Cell / 年: 2017タイトル: Structural and Functional Impacts of ER Coactivator Sequential Recruitment. 著者: Ping Yi / Zhao Wang / Qin Feng / Chao-Kai Chou / Grigore D Pintilie / Hong Shen / Charles E Foulds / Guizhen Fan / Irina Serysheva / Steven J Ludtke / Michael F Schmid / Mien-Chie Hung / Wah ...著者: Ping Yi / Zhao Wang / Qin Feng / Chao-Kai Chou / Grigore D Pintilie / Hong Shen / Charles E Foulds / Guizhen Fan / Irina Serysheva / Steven J Ludtke / Michael F Schmid / Mien-Chie Hung / Wah Chiu / Bert W O'Malley /  要旨: Nuclear receptors recruit multiple coactivators sequentially to activate transcription. This "ordered" recruitment allows different coactivator activities to engage the nuclear receptor complex at ...Nuclear receptors recruit multiple coactivators sequentially to activate transcription. This "ordered" recruitment allows different coactivator activities to engage the nuclear receptor complex at different steps of transcription. Estrogen receptor (ER) recruits steroid receptor coactivator-3 (SRC-3) primary coactivator and secondary coactivators, p300/CBP and CARM1. CARM1 recruitment lags behind the binding of SRC-3 and p300 to ER. Combining cryo-electron microscopy (cryo-EM) structure analysis and biochemical approaches, we demonstrate that there is a close crosstalk between early- and late-recruited coactivators. The sequential recruitment of CARM1 not only adds a protein arginine methyltransferase activity to the ER-coactivator complex, it also alters the structural organization of the pre-existing ERE/ERα/SRC-3/p300 complex. It induces a p300 conformational change and significantly increases p300 HAT activity on histone H3K18 residues, which, in turn, promotes CARM1 methylation activity on H3R17 residues to enhance transcriptional activity. This study reveals a structural role for a coactivator sequential recruitment and biochemical process in ER-mediated transcription. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8830.map.gz emd_8830.map.gz | 6.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8830-v30.xml emd-8830-v30.xml emd-8830.xml emd-8830.xml | 14.9 KB 14.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_8830_fsc.xml emd_8830_fsc.xml | 4.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_8830.png emd_8830.png | 306.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8830 http://ftp.pdbj.org/pub/emdb/structures/EMD-8830 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8830 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8830 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_8830_validation.pdf.gz emd_8830_validation.pdf.gz | 79.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_8830_full_validation.pdf.gz emd_8830_full_validation.pdf.gz | 78.7 KB | 表示 | |
| XML形式データ |  emd_8830_validation.xml.gz emd_8830_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8830 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8830 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8830 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8830 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8830.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8830.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | density map of ERE/ERa%uF061/SRC-3a/p300/CARM1 complex. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.24 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : ER coactivators complex
| 全体 | 名称: ER coactivators complex |
|---|---|
| 要素 |
|
-超分子 #1: ER coactivators complex
| 超分子 | 名称: ER coactivators complex / タイプ: complex / ID: 1 / 親要素: 0 / 詳細: map of ER complex with CARM1 bind |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:  Baculoviridae (ウイルス) / 組換細胞: sf9 Baculoviridae (ウイルス) / 組換細胞: sf9 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: quantifoil / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - Film thickness: 30.0 nm / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR | ||||||||||||
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / デジタル化 - サンプリング間隔: 5.0 µm / デジタル化 - 画像ごとのフレーム数: 2-40 / 撮影したグリッド数: 5 / 平均露光時間: 8.0 sec. / 平均電子線量: 20.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 倍率(公称値): 23000 |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)