+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8679 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Conformational Landscape of the p28-Bound Human Proteasome Regulatory Particle | |||||||||
 マップデータ マップデータ | Final map of human proteasome RP in the TA3 state corrected with a B-factor of -350 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | p28 / 26S proteasome / regulatory particle / 19S / gankyrin / HYDROLASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報proteasome regulatory particle assembly / thyrotropin-releasing hormone receptor binding / nuclear proteasome complex / host-mediated perturbation of viral transcription / positive regulation of inclusion body assembly / positive regulation of cyclin-dependent protein serine/threonine kinase activity / proteasome accessory complex / proteasome regulatory particle / cytosolic proteasome complex / positive regulation of proteasomal protein catabolic process ...proteasome regulatory particle assembly / thyrotropin-releasing hormone receptor binding / nuclear proteasome complex / host-mediated perturbation of viral transcription / positive regulation of inclusion body assembly / positive regulation of cyclin-dependent protein serine/threonine kinase activity / proteasome accessory complex / proteasome regulatory particle / cytosolic proteasome complex / positive regulation of proteasomal protein catabolic process / proteasome-activating activity / proteasome regulatory particle, base subcomplex / negative regulation of programmed cell death / Regulation of ornithine decarboxylase (ODC) / Proteasome assembly / Cross-presentation of soluble exogenous antigens (endosomes) / Somitogenesis / negative regulation of NF-kappaB transcription factor activity / transcription factor binding / negative regulation of release of cytochrome c from mitochondria / regulation of protein catabolic process / negative regulation of DNA damage response, signal transduction by p53 class mediator / proteasome storage granule / general transcription initiation factor binding / blastocyst development / positive regulation of RNA polymerase II transcription preinitiation complex assembly / negative regulation of MAPK cascade / SARS-CoV-1 targets host intracellular signalling and regulatory pathways / enzyme regulator activity / inclusion body / ERAD pathway / positive regulation of protein ubiquitination / Regulation of activated PAK-2p34 by proteasome mediated degradation / Autodegradation of Cdh1 by Cdh1:APC/C / N-glycan trimming in the ER and Calnexin/Calreticulin cycle / APC/C:Cdc20 mediated degradation of Securin / Asymmetric localization of PCP proteins / Ubiquitin-dependent degradation of Cyclin D / proteasome complex / NIK-->noncanonical NF-kB signaling / SCF-beta-TrCP mediated degradation of Emi1 / TNFR2 non-canonical NF-kB pathway / AUF1 (hnRNP D0) binds and destabilizes mRNA / TBP-class protein binding / Vpu mediated degradation of CD4 / Assembly of the pre-replicative complex / Ubiquitin-Mediated Degradation of Phosphorylated Cdc25A / Degradation of DVL / Cdc20:Phospho-APC/C mediated degradation of Cyclin A / Dectin-1 mediated noncanonical NF-kB signaling / Degradation of AXIN / Hh mutants are degraded by ERAD / Activation of NF-kappaB in B cells / Degradation of GLI1 by the proteasome / Hedgehog ligand biogenesis / G2/M Checkpoints / Defective CFTR causes cystic fibrosis / GSK3B and BTRC:CUL1-mediated-degradation of NFE2L2 / Autodegradation of the E3 ubiquitin ligase COP1 / Negative regulation of NOTCH4 signaling / Vif-mediated degradation of APOBEC3G / Regulation of RUNX3 expression and activity / Hedgehog 'on' state / Degradation of GLI2 by the proteasome / GLI3 is processed to GLI3R by the proteasome / FBXL7 down-regulates AURKA during mitotic entry and in early mitosis / APC/C:Cdh1 mediated degradation of Cdc20 and other APC/C:Cdh1 targeted proteins in late mitosis/early G1 / MAPK6/MAPK4 signaling / Degradation of beta-catenin by the destruction complex / ABC-family proteins mediated transport / Oxygen-dependent proline hydroxylation of Hypoxia-inducible Factor Alpha / P-body / CDK-mediated phosphorylation and removal of Cdc6 / CLEC7A (Dectin-1) signaling / SCF(Skp2)-mediated degradation of p27/p21 / Regulation of expression of SLITs and ROBOs / FCERI mediated NF-kB activation / Regulation of PTEN stability and activity / Interleukin-1 signaling / Orc1 removal from chromatin / Regulation of RAS by GAPs / Regulation of RUNX2 expression and activity / cytoplasmic ribonucleoprotein granule / The role of GTSE1 in G2/M progression after G2 checkpoint / Separation of Sister Chromatids / KEAP1-NFE2L2 pathway / UCH proteinases / osteoblast differentiation / Downstream TCR signaling / Antigen processing: Ubiquitination & Proteasome degradation / positive regulation of proteasomal ubiquitin-dependent protein catabolic process / RUNX1 regulates transcription of genes involved in differentiation of HSCs / Neddylation / ER-Phagosome pathway / microtubule cytoskeleton / positive regulation of cell growth / ubiquitin-dependent protein catabolic process / cytoplasmic vesicle / blood microparticle / secretory granule lumen 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 7.3 Å | |||||||||
 データ登録者 データ登録者 | Lu Y / Wu J | |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2017 ジャーナル: Mol Cell / 年: 2017タイトル: Conformational Landscape of the p28-Bound Human Proteasome Regulatory Particle. 著者: Ying Lu / Jiayi Wu / Yuanchen Dong / Shuobing Chen / Shuangwu Sun / Yong-Bei Ma / Qi Ouyang / Daniel Finley / Marc W Kirschner / Youdong Mao /   要旨: The proteasome holoenzyme is activated by its regulatory particle (RP) consisting of two subcomplexes, the lid and the base. A key event in base assembly is the formation of a heterohexameric ring of ...The proteasome holoenzyme is activated by its regulatory particle (RP) consisting of two subcomplexes, the lid and the base. A key event in base assembly is the formation of a heterohexameric ring of AAA-ATPases, which is guided by at least four RP assembly chaperones in mammals: PAAF1, p28/gankyrin, p27/PSMD9, and S5b. Using cryogenic electron microscopy, we analyzed the non-AAA structure of the p28-bound human RP at 4.5 Å resolution and determined seven distinct conformations of the Rpn1-p28-AAA subcomplex within the p28-bound RP at subnanometer resolutions. Remarkably, the p28-bound AAA ring does not form a channel in the free RP and spontaneously samples multiple "open" and "closed" topologies at the Rpt2-Rpt6 and Rpt3-Rpt4 interfaces. Our analysis suggests that p28 assists the proteolytic core particle to select a specific conformation of the ATPase ring for RP engagement and is released in a shoehorn-like fashion in the last step of the chaperone-mediated proteasome assembly. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8679.map.gz emd_8679.map.gz | 81.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8679-v30.xml emd-8679-v30.xml emd-8679.xml emd-8679.xml | 20.9 KB 20.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8679.png emd_8679.png | 67.8 KB | ||
| Filedesc metadata |  emd-8679.cif.gz emd-8679.cif.gz | 6.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8679 http://ftp.pdbj.org/pub/emdb/structures/EMD-8679 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8679 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8679 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_8679_validation.pdf.gz emd_8679_validation.pdf.gz | 466.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_8679_full_validation.pdf.gz emd_8679_full_validation.pdf.gz | 465.7 KB | 表示 | |
| XML形式データ |  emd_8679_validation.xml.gz emd_8679_validation.xml.gz | 6.4 KB | 表示 | |
| CIF形式データ |  emd_8679_validation.cif.gz emd_8679_validation.cif.gz | 7.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8679 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8679 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8679 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8679 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  5vhnMC  8672C  8674C  8675C  8676C  8677C  8678C  8680C  8681C  8682C  8683C  8684C  5vgzC  5vhfC  5vhhC  5vhiC  5vhjC  5vhmC  5vhoC  5vhpC  5vhqC  5vhrC  5vhsC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10091 (タイトル: Conformational Landscape of the p28-Bound Human Proteasome Regulatory Particle EMPIAR-10091 (タイトル: Conformational Landscape of the p28-Bound Human Proteasome Regulatory ParticleData size: 70.0 Data #1: Classified single-particle datasets for multiple conformations of p28-bound human regulatory complex [picked particles - multiframe - processed]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8679.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8679.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Final map of human proteasome RP in the TA3 state corrected with a B-factor of -350 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.86 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
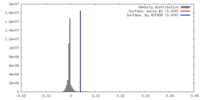
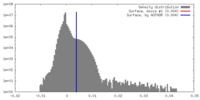
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Proteasome regulatory particle
| 全体 | 名称: Proteasome regulatory particle |
|---|---|
| 要素 |
|
-超分子 #1: Proteasome regulatory particle
| 超分子 | 名称: Proteasome regulatory particle / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: 26S proteasome non-ATPase regulatory subunit 10
| 分子 | 名称: 26S proteasome non-ATPase regulatory subunit 10 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 24.14249 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: CVSNLMVCNL AYSGKLEELK ESILADKSLA TRTDQDSRTA LHWACSAGHT EIVEFLLQLG VPVNDKDDAG WSPLHIAASA GRDEIVKAL LGKGAQVNAV NQNGCTPLHY AASKNRHEIA VMLLEGGANP DAKDHYEATA MHRAAAKGNL KMIHILLYYK A STNIQDTE ...文字列: CVSNLMVCNL AYSGKLEELK ESILADKSLA TRTDQDSRTA LHWACSAGHT EIVEFLLQLG VPVNDKDDAG WSPLHIAASA GRDEIVKAL LGKGAQVNAV NQNGCTPLHY AASKNRHEIA VMLLEGGANP DAKDHYEATA MHRAAAKGNL KMIHILLYYK A STNIQDTE GNTPLHLACD EERVEEAKLL VSQGASIYIE NKEEKTPLQV AKGGLGLILK RMVEG UniProtKB: 26S proteasome non-ATPase regulatory subunit 10 |
-分子 #2: 26S proteasome regulatory subunit 7
| 分子 | 名称: 26S proteasome regulatory subunit 7 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 29.746465 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: PTVTMMQVEE KPDVTYSDVG GCKEQIEKLR EVVETPLLHP ERFVNLGIEP PKGVLLFGPP GTGKTLCARA VANRTDACFI RVIGSELVQ KYVGEGARMV RELFEMARTK KACLIFFDEI DAIGGARFDD GAGGDNEVQR TMLELINQLD GFDPRGNIKV L MATNRPDT ...文字列: PTVTMMQVEE KPDVTYSDVG GCKEQIEKLR EVVETPLLHP ERFVNLGIEP PKGVLLFGPP GTGKTLCARA VANRTDACFI RVIGSELVQ KYVGEGARMV RELFEMARTK KACLIFFDEI DAIGGARFDD GAGGDNEVQR TMLELINQLD GFDPRGNIKV L MATNRPDT LDPALMRPGR LDRKIEFSLP DLEGRTHIFK IHARSMSVER DIRFELLARL CPNSTGAEIR SVCTEAGMFA IR ARRKIAT EKDFLEAVNK VIKSYAKFS UniProtKB: 26S proteasome regulatory subunit 7 |
-分子 #3: 26S proteasome regulatory subunit 4
| 分子 | 名称: 26S proteasome regulatory subunit 4 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 29.901344 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: TDPLVTVMKV EKAPQETYAD IGGLDNQIQE IKESVELPLT HPEYYEEMGI KPPKGVILYG PPGTGKTLLA KAVANQTSAT FLRVVGSEL IQKYLGDGPK LVRELFRVAE EHAPSIVFID EIDAIGTKRY DSNSGGEREI QRTMLELLNQ LDGFDSRGDV K VIMATNRI ...文字列: TDPLVTVMKV EKAPQETYAD IGGLDNQIQE IKESVELPLT HPEYYEEMGI KPPKGVILYG PPGTGKTLLA KAVANQTSAT FLRVVGSEL IQKYLGDGPK LVRELFRVAE EHAPSIVFID EIDAIGTKRY DSNSGGEREI QRTMLELLNQ LDGFDSRGDV K VIMATNRI ETLDPALIRP GRIDRKIEFP LPDEKTKKRI FQIHTSRMTL ADDVTLDDLI MAKDDLSGAD IKAICTEAGL MA LRERRMK VTNEDFKKSK ENVLYKKQE UniProtKB: 26S proteasome regulatory subunit 4 |
-分子 #4: 26S proteasome regulatory subunit 6B
| 分子 | 名称: 26S proteasome regulatory subunit 6B / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 29.497975 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: PEADSSIMML TSDQKPDVMY ADIGGMDIQK QEVREAVELP LTHFELYKQI GIDPPRGVLM YGPPGCGKTM LAKAVAHHTT AAFIRVVGS EFVQKYLGEG PRMVRDVFRL AKENAPAIIF IDEIDAIATK RFDAQTGADR EVQRILLELL NQMDGFDQNV N VKVIMATN ...文字列: PEADSSIMML TSDQKPDVMY ADIGGMDIQK QEVREAVELP LTHFELYKQI GIDPPRGVLM YGPPGCGKTM LAKAVAHHTT AAFIRVVGS EFVQKYLGEG PRMVRDVFRL AKENAPAIIF IDEIDAIATK RFDAQTGADR EVQRILLELL NQMDGFDQNV N VKVIMATN RADTLDPALL RPGRLDRKIE FPLPDRRQKR LIFSTITSKM NLSEEVDLED YVARPDKISG ADINSICQES GM LAVRENR YIVLAKDFEK AYKTV UniProtKB: 26S proteasome regulatory subunit 6B |
-分子 #5: 26S proteasome regulatory subunit 10B
| 分子 | 名称: 26S proteasome regulatory subunit 10B / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 30.933523 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: VDPLVYNMSH EDPGNVSYSE IGGLSEQIRE LREVIELPLT NPELFQRVGI IPPKGCLLYG PPGTGKTLLA RAVASQLDCN FLKVVSSSI VDKYIGESAR LIREMFNYAR DHQPCIIFMD EIDAIGGRRF SEGTSADREI QRTLMELLNQ MDGFDTLHRV K MIMATNRP ...文字列: VDPLVYNMSH EDPGNVSYSE IGGLSEQIRE LREVIELPLT NPELFQRVGI IPPKGCLLYG PPGTGKTLLA RAVASQLDCN FLKVVSSSI VDKYIGESAR LIREMFNYAR DHQPCIIFMD EIDAIGGRRF SEGTSADREI QRTLMELLNQ MDGFDTLHRV K MIMATNRP DTLDPALLRP GRLDRKIHID LPNEQARLDI LKIHAGPITK HGEIDYEAIV KLSDGFNGAD LRNVCTEAGM FA IRADHDF VVQEDFMKAV RKVADSKKLE SKLDYKPV UniProtKB: 26S proteasome regulatory subunit 10B |
-分子 #6: 26S proteasome regulatory subunit 6A
| 分子 | 名称: 26S proteasome regulatory subunit 6A / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 29.773246 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: TEYDSRVKAM EVDERPTEQY SDIGGLDKQI QELVEAIVLP MNHKEKFENL GIQPPKGVLM YGPPGTGKTL LARACAAQTK ATFLKLAGP QLVQMFIGDG AKLVRDAFAL AKEKAPSIIF IDELDAIGTK RFDSEKAGDR EVQRTMLELL NQLDGFQPNT Q VKVIAATN ...文字列: TEYDSRVKAM EVDERPTEQY SDIGGLDKQI QELVEAIVLP MNHKEKFENL GIQPPKGVLM YGPPGTGKTL LARACAAQTK ATFLKLAGP QLVQMFIGDG AKLVRDAFAL AKEKAPSIIF IDELDAIGTK RFDSEKAGDR EVQRTMLELL NQLDGFQPNT Q VKVIAATN RVDILDPALL RSGRLDRKIE FPMPNEEARA RIMQIHSRKM NVSPDVNYEE LARCTDDFNG AQCKAVCVEA GM IALRRGA TELTHEDYME GILEVQAKKK UniProtKB: 26S proteasome regulatory subunit 6A |
-分子 #7: 26S proteasome regulatory subunit 8
| 分子 | 名称: 26S proteasome regulatory subunit 8 / タイプ: protein_or_peptide / ID: 7 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 29.501363 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: KVDPLVSLMM VEKVPDSTYE MIGGLDKQIK EIKEVIELPV KHPELFEALG IAQPKGVLLY GPPGTGKTLL ARAVAHHTDC TFIRVSGSE LVQKFIGEGA RMVRELFVMA REHAPSIIFM DEIDSIGSSR LEGGSGGDSE VQRTMLELLN QLDGFEATKN I KVIMATNR ...文字列: KVDPLVSLMM VEKVPDSTYE MIGGLDKQIK EIKEVIELPV KHPELFEALG IAQPKGVLLY GPPGTGKTLL ARAVAHHTDC TFIRVSGSE LVQKFIGEGA RMVRELFVMA REHAPSIIFM DEIDSIGSSR LEGGSGGDSE VQRTMLELLN QLDGFEATKN I KVIMATNR IDILDSALLR PGRIDRKIEF PPPNEEARLD ILKIHSRKMN LTRGINLRKI AELMPGASGA EVKGVCTEAG MY ALRERRV HVTQEDFEMA VAKVMQKDS UniProtKB: 26S proteasome regulatory subunit 8 |
-分子 #8: 26S proteasome non-ATPase regulatory subunit 2
| 分子 | 名称: 26S proteasome non-ATPase regulatory subunit 2 / タイプ: protein_or_peptide / ID: 8 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 93.790188 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: RDKAPVQPQQ SPAAAPGGTD EKPSGKERRD AGDKDKEQEL SEEDKQLQDE LEMLVERLGE KDTSLYRPAL EELRRQIRSS TTSMTSVPK PLKFLRPHYG KLKEIYENMA PGENKRFAAD IISVLAMTMS GERECLKYRL VGSQEELASW GHEYVRHLAG E VAKEWQEL ...文字列: RDKAPVQPQQ SPAAAPGGTD EKPSGKERRD AGDKDKEQEL SEEDKQLQDE LEMLVERLGE KDTSLYRPAL EELRRQIRSS TTSMTSVPK PLKFLRPHYG KLKEIYENMA PGENKRFAAD IISVLAMTMS GERECLKYRL VGSQEELASW GHEYVRHLAG E VAKEWQEL DDAEKVQREP LLTLVKEIVP YNMAHNAEHE ACDLLMEIEQ VDMLEKDIDE NAYAKVCLYL TSCVNYVPEP EN SALLRCA LGVFRKFSRF PEALRLALML NDMELVEDIF TSCKDVVVQK QMAFMLGRHG VFLELSEDVE EYEDLTEIMS NVQ LNSNFL ALARELDIME PKVPDDIYKT HLENNRFGGS GSQVDSARMN LASSFVNGFV NAAFGQDKLL TDDGNKWLYK NKDH GMLSA AASLGMILLW DVDGGLTQID KYLYSSEDYI KSGALLACGI VNSGVRNECD PALALLSDYV LHNSNTMRLG SIFGL GLAY AGSNREDVLT LLLPVMGDSK SSMEVAGVTA LACGMIAVGS CNGDVTSTIL QTIMEKSETE LKDTYARWLP LGLGLN HLG KGEAIEAILA ALEVVSEPFR SFANTLVDVC AYAGSGNVLK VQQLLHICSE HFDSKEKEED KDKKEKKDKD KKEAPAD MG AHQGVAVLGI ALIAMGEEIG AEMALRTFGH LLRYGEPTLR RAVPLALALI SVSNPRLNIL DTLSKFSHDA DPEVSYNS I FAMGMVGSGT NNARLAAMLR QLAQYHAKDP NNLFMVRLAQ GLTHLGKGTL TLCPYHSDRQ LMSQVAVAGL LTVLVSFLD VRNIILGKSH YVLYGLVAAM QPRMLVTFDE ELRPLPVSVR VGQAVDVV UniProtKB: 26S proteasome non-ATPase regulatory subunit 2 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 7.3 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 21031 |
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 角度割当 | タイプ: PROJECTION MATCHING |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)