+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8315 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the STRA6 receptor for retinol uptake in complex with calmodulin | |||||||||
 マップデータ マップデータ | Reconstruction of STRA6/CaM complex | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Vitamin A / retinol / STRA6 / membrane / MEMBRANE PROTEIN-CALCIUM BINDING PROTEIN complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報vitamin A import into cell / retinol transport / retinol transmembrane transporter activity / chordate embryonic development / retinal binding / retinol binding / enzyme regulator activity / signaling receptor activity / molecular adaptor activity / calmodulin binding ...vitamin A import into cell / retinol transport / retinol transmembrane transporter activity / chordate embryonic development / retinal binding / retinol binding / enzyme regulator activity / signaling receptor activity / molecular adaptor activity / calmodulin binding / calcium ion binding / identical protein binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
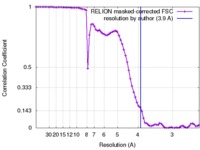
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.9 Å | |||||||||
 データ登録者 データ登録者 | Clarke OB / Chen Y | |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2016 ジャーナル: Science / 年: 2016タイトル: Structure of the STRA6 receptor for retinol uptake. 著者: Yunting Chen / Oliver B Clarke / Jonathan Kim / Sean Stowe / Youn-Kyung Kim / Zahra Assur / Michael Cavalier / Raquel Godoy-Ruiz / Desiree C von Alpen / Chiara Manzini / William S Blaner / ...著者: Yunting Chen / Oliver B Clarke / Jonathan Kim / Sean Stowe / Youn-Kyung Kim / Zahra Assur / Michael Cavalier / Raquel Godoy-Ruiz / Desiree C von Alpen / Chiara Manzini / William S Blaner / Joachim Frank / Loredana Quadro / David J Weber / Lawrence Shapiro / Wayne A Hendrickson / Filippo Mancia /  要旨: Vitamin A homeostasis is critical to normal cellular function. Retinol-binding protein (RBP) is the sole specific carrier in the bloodstream for hydrophobic retinol, the main form in which vitamin A ...Vitamin A homeostasis is critical to normal cellular function. Retinol-binding protein (RBP) is the sole specific carrier in the bloodstream for hydrophobic retinol, the main form in which vitamin A is transported. The integral membrane receptor STRA6 mediates cellular uptake of vitamin A by recognizing RBP-retinol to trigger release and internalization of retinol. We present the structure of zebrafish STRA6 determined to 3.9-angstrom resolution by single-particle cryo-electron microscopy. STRA6 has one intramembrane and nine transmembrane helices in an intricate dimeric assembly. Unexpectedly, calmodulin is bound tightly to STRA6 in a noncanonical arrangement. Residues involved with RBP binding map to an archlike structure that covers a deep lipophilic cleft. This cleft is open to the membrane, suggesting a possible mode for internalization of retinol through direct diffusion into the lipid bilayer. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8315.map.gz emd_8315.map.gz | 28.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8315-v30.xml emd-8315-v30.xml emd-8315.xml emd-8315.xml | 17.6 KB 17.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_8315_fsc.xml emd_8315_fsc.xml | 7.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_8315.png emd_8315.png | 147.5 KB | ||
| マスクデータ |  emd_8315_msk_1.map emd_8315_msk_1.map | 30.5 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-8315.cif.gz emd-8315.cif.gz | 6.3 KB | ||
| その他 |  emd_8315_half_map_1.map.gz emd_8315_half_map_1.map.gz emd_8315_half_map_2.map.gz emd_8315_half_map_2.map.gz | 22.5 MB 22.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8315 http://ftp.pdbj.org/pub/emdb/structures/EMD-8315 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8315 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8315 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_8315_validation.pdf.gz emd_8315_validation.pdf.gz | 973.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_8315_full_validation.pdf.gz emd_8315_full_validation.pdf.gz | 973.2 KB | 表示 | |
| XML形式データ |  emd_8315_validation.xml.gz emd_8315_validation.xml.gz | 12.6 KB | 表示 | |
| CIF形式データ |  emd_8315_validation.cif.gz emd_8315_validation.cif.gz | 17.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8315 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8315 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8315 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8315 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8315.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8315.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of STRA6/CaM complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.255 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_8315_msk_1.map emd_8315_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Reconstruction of STRA6/CaM complex, half map 1
| ファイル | emd_8315_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of STRA6/CaM complex, half map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Reconstruction of STRA6/CaM complex, half map 2
| ファイル | emd_8315_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of STRA6/CaM complex, half map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of zebrafish (D. rerio) STRA6 with copurified calmodulin ...
| 全体 | 名称: Complex of zebrafish (D. rerio) STRA6 with copurified calmodulin reconstituted in amphipol A8-35 |
|---|---|
| 要素 |
|
-超分子 #1: Complex of zebrafish (D. rerio) STRA6 with copurified calmodulin ...
| 超分子 | 名称: Complex of zebrafish (D. rerio) STRA6 with copurified calmodulin reconstituted in amphipol A8-35 タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Calmodulin
| 分子 | 名称: Calmodulin / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 16.82552 KDa |
| 配列 | 文字列: MADQLTEEQI AEFKEAFSLF DKDGDGTITT KELGTVMRSL GQNPTEAELQ DMINEVDADG NGTIDFPEFL TMMARKMKDT DSEEEIREA FRVFDKDGNG FISAAELRHV MTNLGEKLTD EEVDEMIREA DIDGDGQVNY EEFVTMMTSK |
-分子 #2: STRA6
| 分子 | 名称: STRA6 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 75.551523 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSAETVNNYD YSDWYENAAP TKAPVEVIPP CDPTADEGLF HICIAAISLV VMLVLAILAR RQKLSDNQRG LTGLLSPVNF LDHTQHKGL AVAVYGVLFC KLVGMVLSHH PLPFTKEVAN KEFWMILALL YYPTLYYPLL ACGTLHNKVG YVLGSLLSWT H FGILVWQK ...文字列: MSAETVNNYD YSDWYENAAP TKAPVEVIPP CDPTADEGLF HICIAAISLV VMLVLAILAR RQKLSDNQRG LTGLLSPVNF LDHTQHKGL AVAVYGVLFC KLVGMVLSHH PLPFTKEVAN KEFWMILALL YYPTLYYPLL ACGTLHNKVG YVLGSLLSWT H FGILVWQK VDCPKTPQIY KYYALFGSLP QIACLAFLSF QYPLLLFKGL QNTETANASE DLSSSYYRDY VKKILKKKKP TK ISSSTSK PKLFDRLRDA VKSYIYTPED VFRFPLKLAI SVVVAFIALY QMALLLISGV LPTLHIVRRG VDENIAFLLA GFN IILSND RQEVVRIVVY YLWCVEICYV SAVTLSCLVN LLMLMRSMVL HRSNLKGLYR GDSLNVFNCH RSIRPSRPAL VCWM GFTSY QAAFLCLGMA IQTLVFFICI LFAVFLIIIP ILWGTNLMLF HIIGNLWPFW LTLVLAALIQ HVASRFLFIR KDGGT RDLN NRGSLFLLSY ILFLVNVMIG VVLGIWRVVI TALFNIVHLG RLDISLLNRN VEAFDPGYRC YSHYLKIEVS QSHPVM KAF CGLLLQSSGQ DGLSAQRIRD AEEGIQLVQQ EKKQNKVSNA KRARAHWQLL YTLVNNPSLV GSRKHFQCQS SESFING AL SRTSKEGSKK DGSVKEPNKE AESAAASN UniProtKB: Receptor for retinol uptake stra6 |
-分子 #3: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 3 / コピー数: 8 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-分子 #4: CHOLESTEROL
| 分子 | 名称: CHOLESTEROL / タイプ: ligand / ID: 4 / コピー数: 2 / 式: CLR |
|---|---|
| 分子量 | 理論値: 386.654 Da |
| Chemical component information |  ChemComp-CLR: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.6 mg/mL |
|---|---|
| 緩衝液 | pH: 7 |
| グリッド | モデル: Quantifoil, UltrAuFoil, R1.2/1.3 / 材質: GOLD / メッシュ: 400 / 前処理 - タイプ: PLASMA CLEANING |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: 3 uL sample applied, 3-4 second blot time, 30 second wait time, blot force 3, grid blotted from both sides, plunged into liquid ethane (FEI VITROBOT MARK IV).. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F30 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 100.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Tecnai F30 / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-5sy1: |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)