+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8226 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Tomographic subvolume average of membrane-bound Ebola (EBOV-Makona) glycoprotein bound to c13C6 antibody | |||||||||
 マップデータ マップデータ | Membrane-bound Ebola (EBOV-Makona) glycoprotein bound to c13C6 antibody | |||||||||
 試料 試料 | virus-like particle / antibody complex != Ebola virus sp. virus-like particle / antibody complex
| |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  Ebola virus sp. (エボラウイルス) Ebola virus sp. (エボラウイルス) | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 25.0 Å | |||||||||
 データ登録者 データ登録者 | Tran EEH / Subramaniam S | |||||||||
 引用 引用 |  ジャーナル: J Virol / 年: 2016 ジャーナル: J Virol / 年: 2016タイトル: Mapping of Ebolavirus Neutralization by Monoclonal Antibodies in the ZMapp Cocktail Using Cryo-Electron Tomography and Studies of Cellular Entry. 著者: Erin E H Tran / Elizabeth A Nelson / Pranay Bonagiri / James A Simmons / Charles J Shoemaker / Connie S Schmaljohn / Gary P Kobinger / Larry Zeitlin / Sriram Subramaniam / Judith M White /   要旨: ZMapp, a cocktail of three monoclonal antibodies (MAbs; c2G4, c4G7, and c13C6) against the ebolavirus (EBOV) glycoprotein (GP), shows promise for combatting outbreaks of EBOV, as occurred in West ...ZMapp, a cocktail of three monoclonal antibodies (MAbs; c2G4, c4G7, and c13C6) against the ebolavirus (EBOV) glycoprotein (GP), shows promise for combatting outbreaks of EBOV, as occurred in West Africa in 2014. Prior studies showed that Fabs from these MAbs bind a soluble EBOV GP ectodomain and that MAbs c2G4 and c4G7, but not c13C6, neutralize infections in cell cultures. Using cryo-electron tomography, we extended these findings by characterizing the structures of c2G4, c4G7, and c13C6 IgGs bound to native, full-length GP from the West African 2014 isolate embedded in filamentous viruslike particles (VLPs). As with the isolated ectodomain, c13C6 bound to the glycan cap, whereas c2G4 and c4G7 bound to the base region of membrane-bound GP. The tomographic data suggest that all three MAbs bind with high occupancy and that the base-binding antibodies can potentially bridge neighboring GP spikes. Functional studies indicated that c2G4 and c4G7, but not c13C6, competitively inhibit entry of VLPs bearing EBOV GP into the host cell cytoplasm, without blocking trafficking of VLPs to NPC1(+) endolysosomes, where EBOV fuses. Moreover, c2G4 and c4G7 bind to and can block entry mediated by the primed (19-kDa) form of GP without impeding binding of the C-loop of NPC1, the endolysosomal receptor for EBOV. The most likely mode of action of c2G4 and c4G7 is therefore by inhibiting conformational changes in primed, NPC1-bound GP that initiate fusion between the viral and target membranes, similar to the action of certain broadly neutralizing antibodies against influenza hemagglutinin and HIV Env. IMPORTANCE: The recent West African outbreak of ebolavirus caused the deaths of more than 11,000 individuals. Hence, there is an urgent need to be prepared with vaccines and therapeutics for similar ...IMPORTANCE: The recent West African outbreak of ebolavirus caused the deaths of more than 11,000 individuals. Hence, there is an urgent need to be prepared with vaccines and therapeutics for similar future disasters. ZMapp, a cocktail of three MAbs directed against the ebolavirus glycoprotein, is a promising anti-ebolavirus therapeutic. Using cryo-electron tomography, we provide structural information on how each of the MAbs in this cocktail binds to the ebolavirus glycoprotein as it is displayed-embedded in the membrane and present at high density-on filamentous viruslike particles that recapitulate the surface structure and entry functions of ebolavirus. Moreover, after confirming that two of the MAbs bind to the same region in the base of the glycoprotein, we show that they competitively block the entry function of the glycoprotein and that they can do so after the glycoprotein is proteolytically primed and bound to its intracellular receptor, Niemann-Pick C1. These findings should inform future developments of ebolavirus therapeutics. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8226.map.gz emd_8226.map.gz | 998.3 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8226-v30.xml emd-8226-v30.xml emd-8226.xml emd-8226.xml | 13 KB 13 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8226.png emd_8226.png | 43.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8226 http://ftp.pdbj.org/pub/emdb/structures/EMD-8226 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8226 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8226 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_8226_validation.pdf.gz emd_8226_validation.pdf.gz | 78.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_8226_full_validation.pdf.gz emd_8226_full_validation.pdf.gz | 77.4 KB | 表示 | |
| XML形式データ |  emd_8226_validation.xml.gz emd_8226_validation.xml.gz | 492 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8226 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8226 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8226 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8226 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8226.map.gz / 形式: CCP4 / 大きさ: 1.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8226.map.gz / 形式: CCP4 / 大きさ: 1.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Membrane-bound Ebola (EBOV-Makona) glycoprotein bound to c13C6 antibody | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
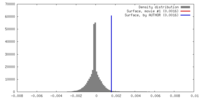
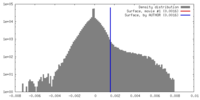
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : virus-like particle / antibody complex
| 全体 | 名称: virus-like particle / antibody complex |
|---|---|
| 要素 |
|
-超分子 #2: Ebola virus sp.
| 超分子 | 名称: Ebola virus sp. / タイプ: virus / ID: 2 / 親要素: 1 詳細: 293T/17 cells were transfected with cDNAs encoding EBOV GP, VP40, mCherry-tagged VP40, and beta-lactamase-tagged VP40. After transfection, VLPs were purified from the cell medium by ...詳細: 293T/17 cells were transfected with cDNAs encoding EBOV GP, VP40, mCherry-tagged VP40, and beta-lactamase-tagged VP40. After transfection, VLPs were purified from the cell medium by centrifugation through a sucrose cushion. NCBI-ID: 205488 / 生物種: Ebola virus sp. / Sci species strain: EBOV-Makona VLP / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: Yes / ウイルス・中空状態: Yes |
|---|---|
| Host system | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK 293T/17 / 組換プラスミド: pWRG7077 Homo sapiens (ヒト) / 組換細胞: HEK 293T/17 / 組換プラスミド: pWRG7077 |
-超分子 #1: virus-like particle / antibody complex
| 超分子 | 名称: virus-like particle / antibody complex / タイプ: complex / ID: 1 / 親要素: 0 |
|---|
-超分子 #3: c13C6 antibody
| 超分子 | 名称: c13C6 antibody / タイプ: complex / ID: 3 / 親要素: 1 詳細: Whole chimerized human IgG from ZMapp antibody cocktail that binds the head region of Ebola virus glycoproteins |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 詳細: 10% sucrose-HM (20 mM HEPES, 20 mM MES, 130 mM NaCl) |
| グリッド | モデル: Quantifoil Multi-A / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 雰囲気: OTHER |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 294 K / 装置: LEICA EM GP 詳細: Blot for 6 seconds before plunging into liquid ethane (LEICA EM GP).. |
| 詳細 | 8 micrograms of Ebola VLPs were incubated with 12.5 micrograms of c13C6 IgG on ice for ~30-60 minutes before plunge-freezing |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: Gatan, Inc エネルギーフィルター - エネルギー下限: 0 eV エネルギーフィルター - エネルギー上限: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 0-4 / 撮影したグリッド数: 1 / 平均露光時間: 2.0 sec. / 平均電子線量: 2.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 倍率(補正後): 23000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 64000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - 点群: C3 (3回回転対称) / 解像度のタイプ: BY AUTHOR / 解像度: 25.0 Å / 解像度の算出法: OTHER 詳細: The average resolution of all maps was estimated based on comparison with features observed when X-ray structures are filtered to this resolution. 使用したサブトモグラム数: 75 |
|---|---|
| 抽出 | トモグラム数: 13 / 使用した粒子像数: 1591 / 手法: Volumes were picked automatically. |
| 最終 角度割当 | タイプ: OTHER |
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: OTHER 当てはまり具合の基準: Fitting was based on locations of antibody escape mutant residues. |
|---|
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)