+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8140 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
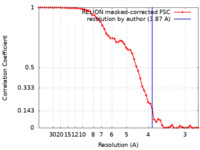
| タイトル | 3.87 Angstrom structure of the nucleosome core particle obtained by phase-plate cryo-EM | |||||||||
 マップデータ マップデータ | None | |||||||||
 試料 試料 |
| |||||||||
| 生物種 | ||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.87 Å | |||||||||
 データ登録者 データ登録者 | Chua EYD / Vogirala VK / Inian O / Wong ASW / Plitzko JM / Danev R / Sandin S | |||||||||
| 資金援助 |  シンガポール, 1件 シンガポール, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2016 ジャーナル: Nucleic Acids Res / 年: 2016タイトル: 3.9 Å structure of the nucleosome core particle determined by phase-plate cryo-EM. 著者: Eugene Y D Chua / Vinod K Vogirala / Oviya Inian / Andrew S W Wong / Lars Nordenskiöld / Juergen M Plitzko / Radostin Danev / Sara Sandin /   要旨: The Volta phase plate is a recently developed electron cryo-microscopy (cryo-EM) device that enables contrast enhancement of biological samples. Here we have evaluated the potential of combining ...The Volta phase plate is a recently developed electron cryo-microscopy (cryo-EM) device that enables contrast enhancement of biological samples. Here we have evaluated the potential of combining phase-plate imaging and single particle analysis to determine the structure of a small protein-DNA complex. To test the method, we made use of a 200 kDa Nucleosome Core Particle (NCP) reconstituted with 601 DNA for which a high-resolution X-ray crystal structure is known. We find that the phase plate provides a significant contrast enhancement that permits individual NCPs and DNA to be clearly identified in amorphous ice. The refined structure from 26,060 particles has an overall resolution of 3.9 Å and the density map exhibits structural features consistent with the estimated resolution, including clear density for amino acid side chains and DNA features such as the phosphate backbone. Our results demonstrate that phase-plate cryo-EM promises to become an important method to determine novel near-atomic resolution structures of small and challenging samples, such as nucleosomes in complex with nucleosome-binding factors. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8140.map.gz emd_8140.map.gz | 14.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8140-v30.xml emd-8140-v30.xml emd-8140.xml emd-8140.xml | 18.6 KB 18.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_8140_fsc.xml emd_8140_fsc.xml | 5.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_8140.png emd_8140.png | 21.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8140 http://ftp.pdbj.org/pub/emdb/structures/EMD-8140 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8140 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8140 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_8140_validation.pdf.gz emd_8140_validation.pdf.gz | 78.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_8140_full_validation.pdf.gz emd_8140_full_validation.pdf.gz | 77.5 KB | 表示 | |
| XML形式データ |  emd_8140_validation.xml.gz emd_8140_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8140 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8140 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8140 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8140 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8140.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8140.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | None | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.38 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Nucleosome core particle with 145 base pairs of the Widom 601 seq...
| 全体 | 名称: Nucleosome core particle with 145 base pairs of the Widom 601 sequence. |
|---|---|
| 要素 |
|
-超分子 #1: Nucleosome core particle with 145 base pairs of the Widom 601 seq...
| 超分子 | 名称: Nucleosome core particle with 145 base pairs of the Widom 601 sequence. タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 実験値: 200 KDa |
-超分子 #2: Widom 601 DNA, 145 base pairs
| 超分子 | 名称: Widom 601 DNA, 145 base pairs / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 実験値: 90 KDa |
-超分子 #3: Histone octamer
| 超分子 | 名称: Histone octamer / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#5 |
|---|---|
| 由来(天然) | 生物種: |
| 組換発現 | 生物種:  |
| 分子量 | 実験値: 110 KDa |
-分子 #1: Widom 601 DNA, 145 base pairs
| 分子 | 名称: Widom 601 DNA, 145 base pairs / タイプ: dna / ID: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 配列 | 文字列: ATCAGAATCC CGGTGCCGAG GCCGCTCAAT TGGTCGTAGA CAGCTCTAGC ACCGCTTAAA CGCACGTACG CGCTGTCCCC CGCGTTTTAA CCGCCAAGGG GATTACTCCC TAGTCTCCAG GCACGTGTCA GATATATACA TCGAT |
-分子 #2: Histone 2A
| 分子 | 名称: Histone 2A / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SGRGKQGGKT RAKAKTRSSR AGLQFPVGRV HRLLRKGNYA ERVGAGAPVY LAAVLEYLTA EILELAGNAA RDNKKTRIIP RHLQLAVRND EELNKLLGRV TIAQGGVLPN IQSVLLPKKT ESSKSKSK |
-分子 #3: Histone 2B
| 分子 | 名称: Histone 2B / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 組換発現 | 生物種:  |
| 配列 | 文字列: PEPAKSAPAP KKGSKKAVTK TQKKDGKKRR KTRKESYAIY VYKVLKQVHP DTGISSKAMS IMNSFVNDVF ERIAGEASRL AHYNKRSTIT SREIQTAVRL LLPGELAKHA VSEGTKAVTK YTSAK |
-分子 #4: Histone 3
| 分子 | 名称: Histone 3 / タイプ: protein_or_peptide / ID: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 組換発現 | 生物種:  |
| 配列 | 文字列: ARTKQTARKS TGGKAPRKQL ATKAARKSAP ATGGVKKPHR YRPGTVALRE IRRYQKSTEL LIRKLPFQRL VREIAQDFKT DLRFQSSAVM ALQEASEAYL VALFEDTNLC AIHAKRVTIM PKDIQLARRI RGERA |
-分子 #5: Histone 4
| 分子 | 名称: Histone 4 / タイプ: protein_or_peptide / ID: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SGRGKGGKGL GKGGAKRHRK VLRDNIQGIT KPAIRRLARR GGVKRISGLI YEETRGVLKV FLENVIRDAV TYTEHAKRKT VTAMDVVYAL KRQGRTLYGF GG |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.5 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R2/2 or R1/4 / 材質: COPPER/RHODIUM / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR | ||||||||||||
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK III / 詳細: 3-4 seconds blotting time. | ||||||||||||
| 詳細 | This sample was monodispersed |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | 位相板: VOLTA PHASE PLATE |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 1-20 / 平均露光時間: 10.0 sec. / 平均電子線量: 31.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| 精密化 | 空間: REAL / プロトコル: OTHER / 温度因子: 78.96 |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)