+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7639 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Single-Molecule 3D Image of neurexin 1 alpha by Individual Particle Electron Tomography (No. 001) | ||||||||||||
 マップデータ マップデータ | neurexin 1 alpha | ||||||||||||
 試料 試料 |
| ||||||||||||
| 生物種 |  | ||||||||||||
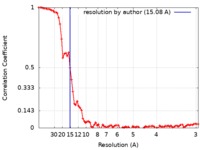
| 手法 | 電子線トモグラフィー法 / ネガティブ染色法 / 解像度: 15.08 Å | ||||||||||||
 データ登録者 データ登録者 | Liu JF / Misra A / Reddy S / White MA / Ren G / Rudenko G | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2018 ジャーナル: J Mol Biol / 年: 2018タイトル: Structural Plasticity of Neurexin 1α: Implications for its Role as Synaptic Organizer. 著者: Jianfang Liu / Anurag Misra / M V V V Sekhar Reddy / Mark Andrew White / Gang Ren / Gabby Rudenko /  要旨: α-Neurexins are synaptic organizing molecules implicated in neuropsychiatric disorders. They bind and arrange an array of different partners in the synaptic cleft. The extracellular region of ...α-Neurexins are synaptic organizing molecules implicated in neuropsychiatric disorders. They bind and arrange an array of different partners in the synaptic cleft. The extracellular region of neurexin 1α (n1α) contains six LNS domains (L1-L6) interspersed by three Egf-like repeats. N1α must encode highly evolved structure-function relationships in order to fit into the narrow confines of the synaptic cleft, and also recruit its large, membrane-bound partners. Internal molecular flexibility could provide a solution; however, it is challenging to delineate because currently no structural methods permit high-resolution structure determination of large, flexible, multi-domain protein molecules. To investigate the structural plasticity of n1α, in particular the conformation of domains that carry validated binding sites for different protein partners, we used a panel of structural techniques. Individual particle electron tomography revealed that the N-terminally and C-terminally tethered domains, L1 and L6, have a surprisingly limited range of conformational freedom with respect to the linear central core containing L2 through L5. A 2.8-Å crystal structure revealed an unexpected arrangement of the L2 and L3 domains. Small-angle X-ray scattering and electron tomography indicated that incorporation of the alternative splice insert SS6 relieves the restricted conformational freedom between L5 and L6, suggesting that SS6 may work as a molecular toggle. The architecture of n1α thus encodes a combination of rigid and flexibly tethered domains that are uniquely poised to work together to promote its organizing function in the synaptic cleft, and may permit allosterically regulated and/or concerted protein partner binding. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7639.map.gz emd_7639.map.gz | 39.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7639-v30.xml emd-7639-v30.xml emd-7639.xml emd-7639.xml | 10.8 KB 10.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_7639_fsc.xml emd_7639_fsc.xml | 7.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_7639.png emd_7639.png | 60.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7639 http://ftp.pdbj.org/pub/emdb/structures/EMD-7639 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7639 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7639 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_7639_validation.pdf.gz emd_7639_validation.pdf.gz | 78.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_7639_full_validation.pdf.gz emd_7639_full_validation.pdf.gz | 77.6 KB | 表示 | |
| XML形式データ |  emd_7639_validation.xml.gz emd_7639_validation.xml.gz | 495 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7639 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7639 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7639 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7639 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7640C  7641C  7642C  7643C  7644C  7645C  7646C  7647C  7648C 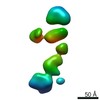 7649C  7650C 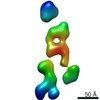 7651C  7652C 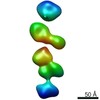 7653C 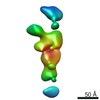 7654C  7655C  7656C  7657C  7658C  7659C  7660C  7661C  7662C  7663C  7664C  7665C  7666C  7667C  7668C  7669C  7670C  7671C  7672C  7673C  7674C  7675C  7676C  7677C  7678C  7679C 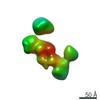 7680C  7681C  7682C  7683C  7684C  7685C  7686C  7687C  7688C  7689C  7690C  7691C  7692C  7693C  7694C  7695C  7696C  7697C  7698C  7699C  7700C  7701C  7702C  7703C  7704C  7705C  7706C  7707C  7708C  7709C  7710C  7711C  7712C  7713C 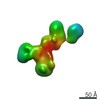 7714C  7715C  7716C  7717C  7718C  7719C 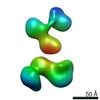 7720C  7721C  7722C  7723C  7724C  7725C  7726C  7727C  7728C  7729C  7730C  7731C  7732C  7733C  7734C  7735C  7736C  7737C  7738C  7739C  7740C  7741C  7742C  7743C  7744C  7745C  7746C  7747C  7748C  7749C  7750C  7751C  7752C  7753C  7754C  7755C  7756C  7757C  7758C  7759C  7760C  7761C  7762C  7763C  7764C  7765C  7766C  7767C  7768C  6cw1C C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7639.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7639.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | neurexin 1 alpha | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.48 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : neurexin 1 alpha
| 全体 | 名称: neurexin 1 alpha |
|---|---|
| 要素 |
|
-超分子 #1: neurexin 1 alpha
| 超分子 | 名称: neurexin 1 alpha / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 137 KDa |
| 組換発現 | 生物種:  unidentified baculovirus (ウイルス) unidentified baculovirus (ウイルス) |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 電子線トモグラフィー法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 / 詳細: 25 mM Tris pH 8, 100 mM NaCl, 3 mM CaCl2 |
|---|---|
| 染色 | タイプ: NEGATIVE / 材質: uranyl formate 詳細: The grid was stained for 15 s by sequential submersion in two drops of uranyl formate (UF). |
| グリッド | 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.039 kPa |
| 詳細 | The purified neurexin 1 alpha proteins were stored in 25 mM Tris pH 8, 100 mM NaCl in flash-frozen aliquots. |
| 切片作成 | その他: NO SECTIONING |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | ZEISS LIBRA120PLUS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: In-column Omega Filter |
| 撮影 | フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 実像数: 81 / 平均露光時間: 1.0 sec. / 平均電子線量: 15.0 e/Å2 |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.2 mm / 倍率(公称値): 80000 |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)