+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7134 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | AlfA Filament bound to AMPPNP | ||||||||||||
 マップデータ マップデータ | AlfA Filament | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | actin / plasmid segregation / filament / CYTOSOLIC PROTEIN | ||||||||||||
| 機能・相同性 | Actin-like protein, N-terminal / Actin like proteins N terminal domain / ATPase, nucleotide binding domain / Actin-like protein N-terminal domain-containing protein 機能・相同性情報 機能・相同性情報 | ||||||||||||
| 生物種 |  | ||||||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 4.2 Å | ||||||||||||
 データ登録者 データ登録者 | Usluer GD / Kollman JM | ||||||||||||
| 資金援助 |  米国, 米国,  カナダ, カナダ,  トルコ, 3件 トルコ, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2018 ジャーナル: Proc Natl Acad Sci U S A / 年: 2018タイトル: Cryo-EM structure of the bacterial actin AlfA reveals unique assembly and ATP-binding interactions and the absence of a conserved subdomain. 著者: Gülsima D Usluer / Frank DiMaio / Shun Kai Yang / Jesse M Hansen / Jessica K Polka / R Dyche Mullins / Justin M Kollman /   要旨: Bacterial actins are an evolutionarily diverse family of ATP-dependent filaments built from protomers with a conserved structural fold. Actin-based segregation systems are encoded on many bacterial ...Bacterial actins are an evolutionarily diverse family of ATP-dependent filaments built from protomers with a conserved structural fold. Actin-based segregation systems are encoded on many bacterial plasmids and function to partition plasmids into daughter cells. The bacterial actin AlfA segregates plasmids by a mechanism distinct from other partition systems, dependent on its unique dynamic properties. Here, we report the near-atomic resolution electron cryo-microscopy structure of the AlfA filament, which reveals a strikingly divergent filament architecture resulting from the loss of a subdomain conserved in all other actins and a mode of ATP binding. Its unusual assembly interfaces and nucleotide interactions provide insight into AlfA dynamics, and expand the range of evolutionary variation accessible to actin quaternary structure. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7134.map.gz emd_7134.map.gz | 135 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7134-v30.xml emd-7134-v30.xml emd-7134.xml emd-7134.xml | 11.2 KB 11.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_7134_fsc.xml emd_7134_fsc.xml | 12 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_7134.png emd_7134.png | 93.8 KB | ||
| Filedesc metadata |  emd-7134.cif.gz emd-7134.cif.gz | 5.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7134 http://ftp.pdbj.org/pub/emdb/structures/EMD-7134 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7134 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7134 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_7134_validation.pdf.gz emd_7134_validation.pdf.gz | 598.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_7134_full_validation.pdf.gz emd_7134_full_validation.pdf.gz | 597.7 KB | 表示 | |
| XML形式データ |  emd_7134_validation.xml.gz emd_7134_validation.xml.gz | 11.4 KB | 表示 | |
| CIF形式データ |  emd_7134_validation.cif.gz emd_7134_validation.cif.gz | 15.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7134 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7134 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7134 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7134 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7134.map.gz / 形式: CCP4 / 大きさ: 144.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7134.map.gz / 形式: CCP4 / 大きさ: 144.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | AlfA Filament | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
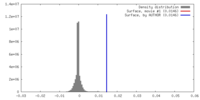
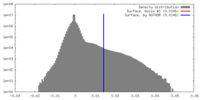
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : AlfA filament
| 全体 | 名称: AlfA filament |
|---|---|
| 要素 |
|
-超分子 #1: AlfA filament
| 超分子 | 名称: AlfA filament / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: Each protomer is bound to the non-hydrolyzable nucleotide analog AMPPNP |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 12750 kDa/nm |
-分子 #1: Bacterial actin AlfA
| 分子 | 名称: Bacterial actin AlfA / タイプ: protein_or_peptide / ID: 1 / コピー数: 9 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 31.150414 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTLTTVIDIG NFSTKYAYKD KKQIKVGSFP SILHSYKPLE DYEGMERVEY NGLDYYVGET VKNFYFGREE QMYFGNTRKG HMEGQIRLV YALYTIFKET GKKEFNLILT CPYESMVTDK KYFVQHFEGE REVIVEGKSF KFTVHNIVMA AEGLGALNFS D SLNCVIVD ...文字列: MTLTTVIDIG NFSTKYAYKD KKQIKVGSFP SILHSYKPLE DYEGMERVEY NGLDYYVGET VKNFYFGREE QMYFGNTRKG HMEGQIRLV YALYTIFKET GKKEFNLILT CPYESMVTDK KYFVQHFEGE REVIVEGKSF KFTVHNIVMA AEGLGALNFS D SLNCVIVD AGSKTLNVLY LINGSISKMD SHTINGGTID NSIMDLAKTF AKTCSNIDYD YPIVCTGGKA EEMKECLENV GY STVSSAE LGEDKPSYYV NSVGLLLKYG RKFEEMFA UniProtKB: Actin-like protein N-terminal domain-containing protein |
-分子 #2: PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 2 / コピー数: 9 / 式: ANP |
|---|---|
| 分子量 | 理論値: 506.196 Da |
| Chemical component information |  ChemComp-ANP: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 0.16 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE |
| 詳細 | Filaments were assembled in 5 mM AMPPNP for 15 minutes at room temperature |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 72.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-6bqw: |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)