+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7058 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of TRPV5 in complex with econazole | |||||||||
 マップデータ マップデータ | TRPV5 in complex with econazole | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | TRPV5 / Econazole / Transient Receptor Potential Channel / Cryo-EM / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of urine volume / calcium ion import across plasma membrane / calcium ion homeostasis / calcium ion transmembrane transport / calcium channel activity / calcium ion transport / protein homotetramerization / calmodulin binding / apical plasma membrane / metal ion binding ...regulation of urine volume / calcium ion import across plasma membrane / calcium ion homeostasis / calcium ion transmembrane transport / calcium channel activity / calcium ion transport / protein homotetramerization / calmodulin binding / apical plasma membrane / metal ion binding / identical protein binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
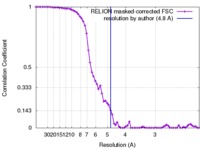
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.8 Å | |||||||||
 データ登録者 データ登録者 | Hughes TET / Lodowski DT | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2018 ジャーナル: Nat Struct Mol Biol / 年: 2018タイトル: Structural basis of TRPV5 channel inhibition by econazole revealed by cryo-EM. 著者: Taylor E T Hughes / David T Lodowski / Kevin W Huynh / Aysenur Yazici / John Del Rosario / Abhijeet Kapoor / Sandip Basak / Amrita Samanta / Xu Han / Sudha Chakrapani / Z Hong Zhou / Marta ...著者: Taylor E T Hughes / David T Lodowski / Kevin W Huynh / Aysenur Yazici / John Del Rosario / Abhijeet Kapoor / Sandip Basak / Amrita Samanta / Xu Han / Sudha Chakrapani / Z Hong Zhou / Marta Filizola / Tibor Rohacs / Seungil Han / Vera Y Moiseenkova-Bell /  要旨: The transient receptor potential vanilloid 5 (TRPV5) channel is a member of the transient receptor potential (TRP) channel family, which is highly selective for Ca, that is present primarily at the ...The transient receptor potential vanilloid 5 (TRPV5) channel is a member of the transient receptor potential (TRP) channel family, which is highly selective for Ca, that is present primarily at the apical membrane of distal tubule epithelial cells in the kidney and plays a key role in Ca reabsorption. Here we present the structure of the full-length rabbit TRPV5 channel as determined using cryo-EM in complex with its inhibitor econazole. This structure reveals that econazole resides in a hydrophobic pocket analogous to that occupied by phosphatidylinositides and vanilloids in TRPV1, thus suggesting conserved mechanisms for ligand recognition and lipid binding among TRPV channels. The econazole-bound TRPV5 structure adopts a closed conformation with a distinct lower gate that occludes Ca permeation through the channel. Structural comparisons between TRPV5 and other TRPV channels, complemented with molecular dynamics (MD) simulations of the econazole-bound TRPV5 structure, allowed us to gain mechanistic insight into TRPV5 channel inhibition by small molecules. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7058.map.gz emd_7058.map.gz | 5.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7058-v30.xml emd-7058-v30.xml emd-7058.xml emd-7058.xml | 14 KB 14 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_7058_fsc.xml emd_7058_fsc.xml | 6.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_7058.png emd_7058.png | 117.8 KB | ||
| Filedesc metadata |  emd-7058.cif.gz emd-7058.cif.gz | 6.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7058 http://ftp.pdbj.org/pub/emdb/structures/EMD-7058 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7058 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7058 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_7058_validation.pdf.gz emd_7058_validation.pdf.gz | 466.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_7058_full_validation.pdf.gz emd_7058_full_validation.pdf.gz | 465.6 KB | 表示 | |
| XML形式データ |  emd_7058_validation.xml.gz emd_7058_validation.xml.gz | 9.5 KB | 表示 | |
| CIF形式データ |  emd_7058_validation.cif.gz emd_7058_validation.cif.gz | 12.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7058 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7058 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7058 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7058 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7058.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7058.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | TRPV5 in complex with econazole | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Full-length TRPV5 in complex with econazole
| 全体 | 名称: Full-length TRPV5 in complex with econazole |
|---|---|
| 要素 |
|
-超分子 #1: Full-length TRPV5 in complex with econazole
| 超分子 | 名称: Full-length TRPV5 in complex with econazole / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Transient receptor potential cation channel subfamily V member 5
| 分子 | 名称: Transient receptor potential cation channel subfamily V member 5 タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 82.899656 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGACPPKAKG PWAQLQKLLI SWPVGEQDWE QYRDRVNMLQ QERIRDSPLL QAAKENDLRL LKILLLNQSC DFQQRGAVGE TALHVAALY DNLEAATLLM EAAPELAKEP ALCEPFVGQT ALHIAVMNQN LNLVRALLAR GASVSARATG AAFRRSPHNL I YYGEHPLS ...文字列: MGACPPKAKG PWAQLQKLLI SWPVGEQDWE QYRDRVNMLQ QERIRDSPLL QAAKENDLRL LKILLLNQSC DFQQRGAVGE TALHVAALY DNLEAATLLM EAAPELAKEP ALCEPFVGQT ALHIAVMNQN LNLVRALLAR GASVSARATG AAFRRSPHNL I YYGEHPLS FAACVGSEEI VRLLIEHGAD IRAQDSLGNT VLHILILQPN KTFACQMYNL LLSYDEHSDH LQSLELVPNH QG LTPFKLA GVEGNTVMFQ HLMQKRKHVQ WTCGPLTSTL YDLTEIDSWG EELSFLELVV SSKKREARQI LEQTPVKELV SFK WKKYGR PYFCVLASLY ILYMICFTTC CIYRPLKLRD DNRTDPRDIT ILQQKLLQEA YVTHQDNIRL VGELVTVTGA VIIL LLEIP DIFRVGASRY FGQTILGGPF HVIIITYASL VLLTMVMRLT NMNGEVVPLS FALVLGWCSV MYFARGFQML GPFTI MIQK MIFGDLMRFC WLMAVVILGF ASAFHITFQT EDPNNLGEFS DYPTALFSTF ELFLTIIDGP ANYSVDLPFM YCITYA AFA IIATLLMLNL FIAMMGDTHW RVAQERDELW RAQVVATTVM LERKMPRFLW PRSGICGYEY GLGDRWFLRV ENHHDQN PL RVLRYVEAFK CSDKEDGQEQ LSEKRPSTVE SGMLSRASVA FQTPSLSRTT SQSSNSHRGW EILRRNTLGH LNLGLDLG E GDGEEVYHF UniProtKB: Transient receptor potential cation channel subfamily V member 5 |
-分子 #2: 1-[(2R)-2-[(4-chlorobenzyl)oxy]-2-(2,4-dichlorophenyl)ethyl]-1H-i...
| 分子 | 名称: 1-[(2R)-2-[(4-chlorobenzyl)oxy]-2-(2,4-dichlorophenyl)ethyl]-1H-imidazole タイプ: ligand / ID: 2 / コピー数: 4 / 式: ECL |
|---|---|
| 分子量 | 理論値: 381.684 Da |
| Chemical component information |  ChemComp-ECL: |
-分子 #3: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 3 / コピー数: 1 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.5 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 実像数: 3313 / 平均露光時間: 0.2 sec. / 平均電子線量: 2.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 倍率(公称値): 45455 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: OTHER / 当てはまり具合の基準: correlation coefficient |
|---|---|
| 得られたモデル |  PDB-6b5v: |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)