+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6969 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Aquareovirus | ||||||||||||||||||
 マップデータ マップデータ | Icosahedral capsid of aquareovirus | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード | icosahedral capsid / symmetry-mismatch / genome / RNA-dependent RNA polymerase / VIRUS | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont entry into host cell via permeabilization of inner membrane / permeabilization of host organelle membrane involved in viral entry into host cell / host cell surface binding / viral inner capsid / viral outer capsid / 7-methylguanosine mRNA capping / viral capsid / mRNA guanylyltransferase activity / mRNA 5'-cap (guanine-N7-)-methyltransferase activity / hydrolase activity ...symbiont entry into host cell via permeabilization of inner membrane / permeabilization of host organelle membrane involved in viral entry into host cell / host cell surface binding / viral inner capsid / viral outer capsid / 7-methylguanosine mRNA capping / viral capsid / mRNA guanylyltransferase activity / mRNA 5'-cap (guanine-N7-)-methyltransferase activity / hydrolase activity / RNA helicase activity / RNA helicase / GTP binding / ATP binding / metal ion binding 類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |  Grass carp reovirus (ウイルス) Grass carp reovirus (ウイルス) | ||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Liu H / Fang Q | ||||||||||||||||||
| 資金援助 |  中国, 5件 中国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2018 ジャーナル: Proc Natl Acad Sci U S A / 年: 2018タイトル: Structure of RNA polymerase complex and genome within a dsRNA virus provides insights into the mechanisms of transcription and assembly. 著者: Xurong Wang / Fuxian Zhang / Rui Su / Xiaowu Li / Wenyuan Chen / Qingxiu Chen / Tao Yang / Jiawei Wang / Hongrong Liu / Qin Fang / Lingpeng Cheng /  要旨: Most double-stranded RNA (dsRNA) viruses transcribe RNA plus strands within a common innermost capsid shell. This process requires coordinated efforts by RNA-dependent RNA polymerase (RdRp) together ...Most double-stranded RNA (dsRNA) viruses transcribe RNA plus strands within a common innermost capsid shell. This process requires coordinated efforts by RNA-dependent RNA polymerase (RdRp) together with other capsid proteins and genomic RNA. Here we report the near-atomic resolution structure of the RdRp protein VP2 in complex with its cofactor protein VP4 and genomic RNA within an aquareovirus capsid using 200-kV cryoelectron microscopy and symmetry-mismatch reconstruction. The structure of these capsid proteins enabled us to observe the elaborate nonicosahedral structure within the double-layered icosahedral capsid. Our structure shows that the RdRp complex is anchored at the inner surface of the capsid shell and interacts with genomic dsRNA and four of the five asymmetrically arranged N termini of the capsid shell proteins under the fivefold axis, implying roles for these N termini in virus assembly. The binding site of the RNA end at VP2 is different from the RNA cap binding site identified in the crystal structure of orthoreovirus RdRp λ3, although the structures of VP2 and λ3 are almost identical. A loop, which was thought to separate the RNA template and transcript, interacts with an apical domain of the capsid shell protein, suggesting a mechanism for regulating RdRp replication and transcription. A conserved nucleoside triphosphate binding site was localized in our RdRp cofactor protein VP4 structure, and interactions between the VP4 and the genomic RNA were identified. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6969.map.gz emd_6969.map.gz | 2.5 GB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6969-v30.xml emd-6969-v30.xml emd-6969.xml emd-6969.xml | 22.9 KB 22.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_6969.png emd_6969.png | 96.6 KB | ||
| マスクデータ |  emd_6969_msk_1.map emd_6969_msk_1.map | 11.9 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-6969.cif.gz emd-6969.cif.gz | 8.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6969 http://ftp.pdbj.org/pub/emdb/structures/EMD-6969 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6969 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6969 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6969_validation.pdf.gz emd_6969_validation.pdf.gz | 795.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6969_full_validation.pdf.gz emd_6969_full_validation.pdf.gz | 795.3 KB | 表示 | |
| XML形式データ |  emd_6969_validation.xml.gz emd_6969_validation.xml.gz | 11.2 KB | 表示 | |
| CIF形式データ |  emd_6969_validation.cif.gz emd_6969_validation.cif.gz | 13.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6969 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6969 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6969 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6969 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6969.map.gz / 形式: CCP4 / 大きさ: 2.7 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6969.map.gz / 形式: CCP4 / 大きさ: 2.7 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Icosahedral capsid of aquareovirus | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.932 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_6969_msk_1.map emd_6969_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
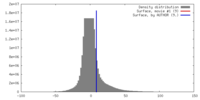
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Grass carp reovirus
| 全体 | 名称:  Grass carp reovirus (ウイルス) Grass carp reovirus (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Grass carp reovirus
| 超分子 | 名称: Grass carp reovirus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1-#6 / NCBI-ID: 128987 / 生物種: Grass carp reovirus / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|
-分子 #1: Outer capsid VP7
| 分子 | 名称: Outer capsid VP7 / タイプ: protein_or_peptide / ID: 1 / コピー数: 10 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Grass carp reovirus (ウイルス) Grass carp reovirus (ウイルス) |
| 分子量 | 理論値: 29.844648 KDa |
| 組換発現 | 生物種:  Ctenopharyngodon idella (ソウギョ) Ctenopharyngodon idella (ソウギョ) |
| 配列 | 文字列: MPLHMIPQVA HAMVRAAAAG RLTLYTRTRT ETTNFDHAEY VTCGRYTICA FCLTTLAPHA NVKTIQDSHA CSRQPNEAIR SLVEVSDKA QTALVGSRTV DYHELDVKAG FVAPTADETI APSKDIVELP FRTCDLDDSS ATACVRNHCQ AGHDGVIHLP I LSGDFKLP ...文字列: MPLHMIPQVA HAMVRAAAAG RLTLYTRTRT ETTNFDHAEY VTCGRYTICA FCLTTLAPHA NVKTIQDSHA CSRQPNEAIR SLVEVSDKA QTALVGSRTV DYHELDVKAG FVAPTADETI APSKDIVELP FRTCDLDDSS ATACVRNHCQ AGHDGVIHLP I LSGDFKLP NEHPTKPLDD THPHDKVLTR CPKTGLLLVH DTHAHATAVV ATAATRAILM HDLLTSANAD DGHQARSACY GP AFNNLTF ACHSTCASDM AHFDCGQIVG LDLHVEPSD UniProtKB: Outer capsid VP7 |
-分子 #2: N-terminus of outer capsid protein VP5
| 分子 | 名称: N-terminus of outer capsid protein VP5 / タイプ: protein_or_peptide / ID: 2 / コピー数: 10 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Grass carp reovirus (ウイルス) Grass carp reovirus (ウイルス) |
| 分子量 | 理論値: 4.302686 KDa |
| 組換発現 | 生物種:  Ctenopharyngodon idella (ソウギョ) Ctenopharyngodon idella (ソウギョ) |
| 配列 | 文字列: MGNVQTSVNT YNITGDGNSF TPTSDMTSTA APAIDLKPGV LN UniProtKB: Putative outer capsid VP4 |
-分子 #3: C-terminus of outer capsid protein VP5
| 分子 | 名称: C-terminus of outer capsid protein VP5 / タイプ: protein_or_peptide / ID: 3 / コピー数: 10 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Grass carp reovirus (ウイルス) Grass carp reovirus (ウイルス) |
| 分子量 | 理論値: 64.362086 KDa |
| 組換発現 | 生物種:  Ctenopharyngodon idella (ソウギョ) Ctenopharyngodon idella (ソウギョ) |
| 配列 | 文字列: PTGKLWRPVG TSVATIDSLA IVSDRFGQYS FVNEGMRETF SKALFDINMW QPLFQATKTG CGPIVLSSFT TTTSGYVGAT AGDALDNPV TNGVFISTVQ IMNLQRTIAA RMRDVALWQK HLDTAMTMLT PDISAGSASC NWKSLLAFAK DILPLDNLCL T YPNEFYNV ...文字列: PTGKLWRPVG TSVATIDSLA IVSDRFGQYS FVNEGMRETF SKALFDINMW QPLFQATKTG CGPIVLSSFT TTTSGYVGAT AGDALDNPV TNGVFISTVQ IMNLQRTIAA RMRDVALWQK HLDTAMTMLT PDISAGSASC NWKSLLAFAK DILPLDNLCL T YPNEFYNV AIHRYPALKP GNPDTKLPDA QAHPLGEVAG AFNAATSEVG SLVGSSSTLS QAISTMAGKD LDLIEADTPL PV SVFTPSL APRSYRPAFI KPEDAKWIAE FNNSSLIRKT LTYSGATYTV QLGPGPTRVI DMNAMIDSVL TLDVSGTILP YDT NPDLST SVPAFVLIQT SVPIQQVTTA ANITAITVVS AAGASAINLA INVRGQPRFN MLHLQATFER ETITGIPYIY GLGT FLIPS PTSSSNFSNP TLMDGLLTVT PVLLRETTYK GEVVDAIVPA TVMANQTSEE VASALANDAI VLVSNHLNKL ANVVG DAIP VASRTDDSAT SAIVSRLAVQ HKLSQVGQAS PTPPDYPLLW RRAKRAASMF VSNPSLALQV GIPVLTQSGM LSALTS GVG TALRTGSLGK GVTDASEKLR ARQSLTVAKQ AFFDQIGSLW PGK UniProtKB: Putative outer capsid VP4 |
-分子 #4: Core protein VP6
| 分子 | 名称: Core protein VP6 / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Grass carp reovirus (ウイルス) Grass carp reovirus (ウイルス) |
| 分子量 | 理論値: 44.606535 KDa |
| 組換発現 | 生物種:  Ctenopharyngodon idella (ソウギョ) Ctenopharyngodon idella (ソウギョ) |
| 配列 | 文字列: MAQRQFFGLT YNFYGQPAPL FDLNDLQELA GCYARPWTSR FSHLAISTGS LPVWSARYPS VASRNIVVNT LLGAHLNPFA GGQITSHQG ITWRDPVLSS LAPVPAIQPP PVWAVAENVL LDSNNYPTYV LNLSSMWPIN QDVHIMTMWA LSDQGPIYHL E VPVDPMPA ...文字列: MAQRQFFGLT YNFYGQPAPL FDLNDLQELA GCYARPWTSR FSHLAISTGS LPVWSARYPS VASRNIVVNT LLGAHLNPFA GGQITSHQG ITWRDPVLSS LAPVPAIQPP PVWAVAENVL LDSNNYPTYV LNLSSMWPIN QDVHIMTMWA LSDQGPIYHL E VPVDPMPA ATTAALMAYT GVPIAHLAQT AYRFAGQLPQ SPDSTMVSTI RWLSAIWFGS LTGRLNRSRT CNGFYFEFAK PA LNPDQAV LKWNDGARAA PPAAAQSSYI RCISPHWQHQ IVEVAGALMS QSVTAVTGLP ALIDEATLPA WSQGVANLTG NGQ GVVPCL DYNPVPMAAA RHLQWRQDGL ITAAQEAQLN NDYTAYALTI ERHLTAMLVA NPIAAGRMPI QPFNAADFGQ AGQT AAAVA LAQAMFV UniProtKB: Core protein VP6 |
-分子 #5: VP1
| 分子 | 名称: VP1 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Grass carp reovirus (ウイルス) Grass carp reovirus (ウイルス) |
| 分子量 | 理論値: 141.512156 KDa |
| 組換発現 | 生物種:  Ctenopharyngodon idella (ソウギョ) Ctenopharyngodon idella (ソウギョ) |
| 配列 | 文字列: MAAVFGIQLV PKLNTSTTRR TFLPLRFDLL LDRLQSTNLH GVLYRALDFN PVDRSATVIQ TYPPLNAWSP HHAFIENPLD YRDWTEFIH DRALAFVGVL TQRYPLTQNA QRYTNPLVLG AAFGDFLNAR SIDIFLDRLF YDPTQDSPIT AITKFPYQWT I DSNVTTDS ...文字列: MAAVFGIQLV PKLNTSTTRR TFLPLRFDLL LDRLQSTNLH GVLYRALDFN PVDRSATVIQ TYPPLNAWSP HHAFIENPLD YRDWTEFIH DRALAFVGVL TQRYPLTQNA QRYTNPLVLG AAFGDFLNAR SIDIFLDRLF YDPTQDSPIT AITKFPYQWT I DSNVTTDS VRTSAGCKYI TLYGYDPSRP STPATYGKHR PTYATVFYYS TLPARSRLLA NLAAGPTVLE HFDSPTYGPH LL LPQTGDV LGYSSSLISQ AALLMVESVM DALRDNANAS ASTAVTRLDQ SYHPVTSFDP STFNTLLQRA TNLALLAVQG VQS ESAIPA IPTMSDVRSF VARLMAEGDP QQWFPYRVDQ ILYWPESPFV PPIGPFYAPF RPVNFPFTTG SYTVVPDASR PLRL LPQYR NATITVQQAD DAYEDTALSP LITTHGFCVT GGVFTSIYDI SGDPTAYPPA QLVDAPNDYF DRERMARRDL FRRLR APAD RSAIKDRAVF DFLASLVNPT TANPVLDTSF SMAYLGASSA HANADEPVIL ADIRSGSIPG LPIPRRIVQF GYDVVH GSL LDLSRAVPTG TFGLVYADLD QVEDAGTDMP AANRAAIAML GTALQMTTAG GVSVLKVNFP TRAFWTQVFN LYATHAT TL HLVKPTIVNS SEVFLVFGGR QSNGALRSTT ALQRALLSLY ARNAAIDRAV THIPFFGVPD DGTSDLGIDA VRLFDPMF S DAVANLPSNA LASLVSRVVP SSIMFTRVPS NGPVSTTIYG KRTFLSNRRR ARLRDVPMLI TTTLVHQRRF TTPPTFTLF SSEAVPVTTL VAAGYNSFIS EQTRNPNLAH LLDLGTGPEC RILSLIPPTL QVTMSDSRPC AELMASFDPA LTAYVQGDYS TAAFWNGIR CDSATAIFTI GAAAAAAGTD LIAFVQQLIP RIVAAGGTRM WLQLNTPLYE VSSLPDLIEI DLRDHVYRFN G GERVEPYA DPVPLQQAIA ALLPAAALSW HTLSPTCDWL PYIIGVGSPL NLSDINTAIS YSRLTPILHI DTTTPPLRVN PV PTPLNQQ CAIRITSLDP AAVLSVQHNG VEVIGGTPGN VISVAGAAAL QYILANQEFL LQFTPTLPGI FDVFLTTLGQ PPV PRGSFT ITPPPTTVAL NMPPPRQLDF TDVGNDARIT CDPYYQLAVC IFKDGQYVRV NPEKASVVTN APNRDLHFVL DLAD NHVLL YLCDVTPSGL GDRIAFPIVD IYRIAFPRNT PVRASLPYTG GGAHLTSGGN PFMSLTTPPA VLPAGVALAA LSTSV ATQY PTYTLPAGVY EYVIE UniProtKB: VP1 |
-分子 #6: VP3
| 分子 | 名称: VP3 / タイプ: protein_or_peptide / ID: 6 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Grass carp reovirus (ウイルス) Grass carp reovirus (ウイルス) |
| 分子量 | 理論値: 132.203312 KDa |
| 組換発現 | 生物種:  Ctenopharyngodon idella (ソウギョ) Ctenopharyngodon idella (ソウギョ) |
| 配列 | 文字列: MPRRSARKAQ SAIASPADTN VVPAKDAPTT NSPPSTTSPN QAAADANQQQ AGIVSSQSGP NAVGDSAPSS SVNNDGDIIT RPTSDSIAA VANATKPAAV VSDPQSMKVT PIVNPSSYVC NVCNARFSTM SALSEHLRSD HRDDASTLLA TPMINNAIRS F LTAWDDIR ...文字列: MPRRSARKAQ SAIASPADTN VVPAKDAPTT NSPPSTTSPN QAAADANQQQ AGIVSSQSGP NAVGDSAPSS SVNNDGDIIT RPTSDSIAA VANATKPAAV VSDPQSMKVT PIVNPSSYVC NVCNARFSTM SALSEHLRSD HRDDASTLLA TPMINNAIRS F LTAWDDIR ILSPDVSSKS LSAYLDSAVA NGPELIIEDT GLCTSFMLLD NIPSAHLTKE LIGFTWFMQM YQMTPPLPEG AV NRIVCMT NWASLGDEGR GLEVRLPPPT DSSVHAYKTV LSRGYIDNAQ FNPLALRSNV LLMLLQFTLS NLKINKSSTF TSD VTTITS GRMIRAFEGR PELLALAYPG RAVLPTQTKN AQFLSTAIAD RIGRLDRANL IGGEVSAMVE CMELCDALTL HIRE TYIML LRSMHQDPTQ IVQIVNECAN NLLNSTIPIS LRPTILCPWF ASSEDLRLQQ VMHLVNISSN TAAALPLVEA LSTLL RSVT PLVLDPTVLT NAITTISEST TQTISPISEI LRLLQPMGND YAAFWKCIAS WAYNGLVTTV LSEDAFPDSS QSITHL PSM WKCLFLTLAG PMTSDPHSPV KVFMALANLL AQPEPIAIGV PGMHQTTPAS QFSHPGVWPP GFLNPQLINP QQAPLLR AF AEHIRANWPQ PSEFGYGSTL QGSANLFIPS NRMVYPWPNQ PLPRLTVAPT YDSAMSNWIS TTIAFFIRVV NSVNMTAT V NDLTRRTMTG VMTAMRQVKT MTPFYIQHMC PTELSVLASV TVTPPFQVPF TRLVQNDVIT NVLVARVDPA QRGDAAVDI RATHATFAAA LPVDPAAIVV AMLCGQTETN LIPSHHYGKA FAPLFASNAM FTRNQRAVIT REAFVCARSA VAQCQDAGFL VPRPLDALR QFDVTSAAAA EIMHAVNDAF KTAFDLDGAL LDGLALYGDP RIADLSAAYL QYGGNVVREH VPPGPSHIHR A LQQVESTF MAEMNLFNVA RGNLYLVQTA TNGNWSPMAP VAAPPFVRGG PNVRVVGRFG TIVPRPNGLE PQLIDDGNVP RD IAGDWVY PSDVLQVSVA VFRDYVWPMV KAGRTRVLVE LGHYVYTLHY YDPQISLDEA PILEEWLSKI NPAGIPPVPF CIP IPQVYP CITARRVHYA FTSENNNDSL FSTNAASIDT AFGENAAVSP LRWPGLVDPN YRVGTNDLPN RITLYNSLYR YNFT YPTLD GIMYVRSAT UniProtKB: RNA helicase |
-分子 #7: MYRISTIC ACID
| 分子 | 名称: MYRISTIC ACID / タイプ: ligand / ID: 7 / コピー数: 10 / 式: MYR |
|---|---|
| 分子量 | 理論値: 228.371 Da |
| Chemical component information |  ChemComp-MYR: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 平均電子線量: 25.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)