+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6370 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 3D-Structure of negatively stained Schistosome myosin filament obtained by low-dose electron microscopy | |||||||||
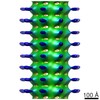
 マップデータ マップデータ | Map of Schistosome thick filaments. Initial view is from the Z-line perspective. If the map is rotated by 90 degrees in x direction, the J motif of the interacting heads is featured and the backbone subfilaments can be seen clearly. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Schistosoma mansoni / rigid docking / single particle reconstruction / Iterative Helical Real Space Reconstruction (IHRSR) / negative stain / thick filament / smooth muscle | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報myosin filament / myosin complex / myosin II complex / structural constituent of muscle / sarcomere organization / microfilament motor activity / myofibril / cytoskeletal motor activity / muscle contraction / actin filament organization ...myosin filament / myosin complex / myosin II complex / structural constituent of muscle / sarcomere organization / microfilament motor activity / myofibril / cytoskeletal motor activity / muscle contraction / actin filament organization / actin filament binding / calcium ion binding / ATP binding / membrane / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | らせん対称体再構成法 / ネガティブ染色法 / 解像度: 23.0 Å | |||||||||
 データ登録者 データ登録者 | Sulbaran G / Alamo L / Pinto A / Marquez G / Mendez F / Padron R / Craig R | |||||||||
 引用 引用 |  ジャーナル: PROC.NAT.ACAD.SCI.USA / 年: 2015 ジャーナル: PROC.NAT.ACAD.SCI.USA / 年: 2015タイトル: An invertebrate smooth muscle with striated muscle myosin filaments. 著者: Sulbaran G / Alamo L / Pinto A / Marquez G / Mendez F / Padron R / Craig R #1:  ジャーナル: BIOPHYS.J. / 年: 2014 ジャーナル: BIOPHYS.J. / 年: 2014タイトル: Schistosome Muscles Contain Striated Muscle-Like Myosin Filaments in a Smooth Muscle-Like Architecture 著者: Sulbaran G / Alamo L / Pinto A / Marquez G / Mendez F / Padron R / Craig R | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6370.map.gz emd_6370.map.gz | 1.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6370-v30.xml emd-6370-v30.xml emd-6370.xml emd-6370.xml | 15.5 KB 15.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_6370_fsc.xml emd_6370_fsc.xml | 5.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_6370.tif emd_6370.tif emd_6370_1.tif emd_6370_1.tif emd_6370_2.tif emd_6370_2.tif | 267.7 KB 285.8 KB 237.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6370 http://ftp.pdbj.org/pub/emdb/structures/EMD-6370 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6370 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6370 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6370_validation.pdf.gz emd_6370_validation.pdf.gz | 333.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6370_full_validation.pdf.gz emd_6370_full_validation.pdf.gz | 332.8 KB | 表示 | |
| XML形式データ |  emd_6370_validation.xml.gz emd_6370_validation.xml.gz | 8.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6370 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6370 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6370 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6370 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6370.map.gz / 形式: CCP4 / 大きさ: 8.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6370.map.gz / 形式: CCP4 / 大きさ: 8.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map of Schistosome thick filaments. Initial view is from the Z-line perspective. If the map is rotated by 90 degrees in x direction, the J motif of the interacting heads is featured and the backbone subfilaments can be seen clearly. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 5.7 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Myosin thick filaments from Schistosoma mansoni smooth muscle
| 全体 | 名称: Myosin thick filaments from Schistosoma mansoni smooth muscle |
|---|---|
| 要素 |
|
-超分子 #1000: Myosin thick filaments from Schistosoma mansoni smooth muscle
| 超分子 | 名称: Myosin thick filaments from Schistosoma mansoni smooth muscle タイプ: sample / ID: 1000 詳細: S. mansoni relaxed myosin thick filaments were isolated by permeabilizing and homogenizing whole animals in relaxing solution, centrifuged, and resuspended in blebbistatin. 集合状態: polymer of myosin II molecules helically assembled over a paramyosin core Number unique components: 1 |
|---|
-分子 #1: Myosin II
| 分子 | 名称: Myosin II / タイプ: protein_or_peptide / ID: 1 / Name.synonym: Myosin Type II 詳細: Myosin II is a protein complex formed by two heavy chains and two associated light chains (for each myosin head), plus additional proteins such as paramyosin. Using ATP hydrolysis, myosin ...詳細: Myosin II is a protein complex formed by two heavy chains and two associated light chains (for each myosin head), plus additional proteins such as paramyosin. Using ATP hydrolysis, myosin functions as a molecular motor, producing movement by causing actin filaments to slide. コピー数: 1 集合状態: polymer of myosin II molecules helically assembled over a paramyosin core 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:  |
| 配列 | UniProtKB: Paramyosin GO: myosin complex, cytoskeletal motor activity, ATP binding InterPro: Myosin, N-terminal, SH3-like, P-loop containing nucleoside triphosphate hydrolase, Myosin head, motor domain, INTERPRO: IPR027401, Myosin tail, IQ motif, EF-hand binding site |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7 詳細: 100 mM NaCl, 3 mM MgCl2, 1 mM EGTA, 5 mM PIPES, 1mM NaN3, 5 mM MgATP, 0.01 mM blebbistatin, protease inhibitor cocktail (Sigma P-8465) |
|---|---|
| 染色 | タイプ: NEGATIVE 詳細: One drop of filament suspension was placed on grids and negatively stained with 1% uranyl acetate. |
| グリッド | 詳細: 400-mesh holey carbon grids. Specimens were imaged on thin carbon extending over the holes. |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM120T |
|---|---|
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 240,000 times magnification |
| 詳細 | 1.5 post-magnification, low-dose conditions |
| 日付 | 2013年3月1日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: TVIPS TEMCAM-F224 (2k x 2k) 実像数: 263 / 平均電子線量: 10 e/Å2 詳細: Images were acquired with a 2K x 2K CCD TVIPS camera model F224HD at 5.7 A/pixel. ビット/ピクセル: 16 |
| 電子線 | 加速電圧: 80 kV / 電子線源: LAB6 |
| 電子光学系 | 倍率(補正後): 42000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 0.6 µm / 倍率(公称値): 42000 |
| 試料ステージ | 試料ホルダー: Room temperature holder / 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
- 画像解析
画像解析
| 詳細 | 820 thick filament halves were selected from micrographs and stored in SPIDER format. 131 x 131 pixel segments were cut from these filaments, corresponding to a window of 74.7 nm (~five 14.5 nm-spaced crowns of heads). |
|---|---|
| 最終 再構成 | 想定した対称性 - らせんパラメータ - Δz: 145 Å 想定した対称性 - らせんパラメータ - ΔΦ: 30 ° 想定した対称性 - らせんパラメータ - 軸対称性: C4 (4回回転対称) アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 23.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: SPIDER, EMAN2 詳細: For each iteration of reconstruction (30 cycles), filament segment projections were compared with different projections of the reference reconstruction as follows: seven 2.3 nm axial shifts, ...詳細: For each iteration of reconstruction (30 cycles), filament segment projections were compared with different projections of the reference reconstruction as follows: seven 2.3 nm axial shifts, 2 degree intervals of rotation about the filament axis up to 90 degrees, and 2 degree intervals of out-of-plane tilting from -10 degrees to +10 degrees. The total number of projections was 7 x 45 x 11 = 3465. For the final 19 cycles of the reconstruction, we used only the best-ordered 420 filament halves (those in which >30% of the segments were found good enough to be used by the reconstruction script in the back-projection in previous cycles). From ~17,000 segments, ~9,500 (56%) were included in the final reconstruction. This final 3D-reconstruction was the average of the last 19 reconstructions between cycles 12 - 30. Its resolution, according to the 0.5 Fourier Shell Correlation (FSC) criterion, was 2.3 nm. |
-原子モデル構築 1
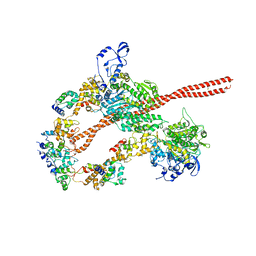
| 初期モデル | PDB ID: Chain - #0 - Chain ID: A / Chain - #1 - Chain ID: B / Chain - #2 - Chain ID: C / Chain - #3 - Chain ID: D / Chain - #4 - Chain ID: E / Chain - #5 - Chain ID: F |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | 3DTP was fitted as a rigid body using the "Fit in Map" tool of UCSF Chimera. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
| 得られたモデル | 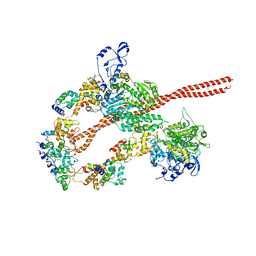 PDB-3jax: |
 ムービー
ムービー コントローラー
コントローラー