[English] 日本語
 Yorodumi
Yorodumi- EMDB-6319: Structure of bacterial chemotaxis signaling CheA2-trimer core com... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: EMDB / ID: EMD-6319 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Structure of bacterial chemotaxis signaling CheA2-trimer core complex by cryo-electron tomography and subvolume averaging | |||||||||
 Map data Map data | Structure of bacterial chemotaxis signaling CheA2-trimer core complex by cryo-electron tomography and subvolume averaging | |||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | Bacterial chemotaxis / Signal transduction / cryo-Electron Tomography / Molecular dynamics simulation / all-atom | |||||||||
| Function / homology |  Function and homology information Function and homology informationnegative regulation of protein modification process / detection of chemical stimulus / methyl accepting chemotaxis protein complex / positive regulation of post-translational protein modification / protein histidine kinase binding / bacterial-type flagellum-dependent swimming motility / regulation of bacterial-type flagellum-dependent cell motility / aerotaxis / protein histidine kinase activity / cell tip ...negative regulation of protein modification process / detection of chemical stimulus / methyl accepting chemotaxis protein complex / positive regulation of post-translational protein modification / protein histidine kinase binding / bacterial-type flagellum-dependent swimming motility / regulation of bacterial-type flagellum-dependent cell motility / aerotaxis / protein histidine kinase activity / cell tip / regulation of chemotaxis / thermotaxis / signal complex assembly / phosphorelay sensor kinase activity / histidine kinase / phosphorelay signal transduction system / establishment of localization in cell / cellular response to amino acid stimulus / protein homooligomerization / chemotaxis / transmembrane signaling receptor activity / protein domain specific binding / signal transduction / protein homodimerization activity / ATP binding / identical protein binding / plasma membrane / cytoplasm / cytosol Similarity search - Function | |||||||||
| Biological species |  | |||||||||
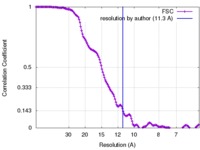
| Method | subtomogram averaging / cryo EM / Resolution: 11.3 Å | |||||||||
 Authors Authors | Cassidy CK / Himes BA / Alvarez FJ / Ma J / Zhou G / Perilla JR / Schulten K / Zhang P | |||||||||
 Citation Citation |  Journal: Elife / Year: 2015 Journal: Elife / Year: 2015Title: CryoEM and computer simulations reveal a novel kinase conformational switch in bacterial chemotaxis signaling. Authors: C Keith Cassidy / Benjamin A Himes / Frances J Alvarez / Jun Ma / Gongpu Zhao / Juan R Perilla / Klaus Schulten / Peijun Zhang /  Abstract: Chemotactic responses in bacteria require large, highly ordered arrays of sensory proteins to mediate the signal transduction that ultimately controls cell motility. A mechanistic understanding of ...Chemotactic responses in bacteria require large, highly ordered arrays of sensory proteins to mediate the signal transduction that ultimately controls cell motility. A mechanistic understanding of the molecular events underlying signaling, however, has been hampered by the lack of a high-resolution structural description of the extended array. Here, we report a novel reconstitution of the array, involving the receptor signaling domain, histidine kinase CheA, and adaptor protein CheW, as well as a density map of the core-signaling unit at 11.3 Å resolution, obtained by cryo-electron tomography and sub-tomogram averaging. Extracting key structural constraints from our density map, we computationally construct and refine an atomic model of the core array structure, exposing novel interfaces between the component proteins. Using all-atom molecular dynamics simulations, we further reveal a distinctive conformational change in CheA. Mutagenesis and chemical cross-linking experiments confirm the importance of the conformational dynamics of CheA for chemotactic function. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Movie |
 Movie viewer Movie viewer |
|---|---|
| Structure viewer | EM map:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| Supplemental images |
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_6319.map.gz emd_6319.map.gz | 2.6 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-6319-v30.xml emd-6319-v30.xml emd-6319.xml emd-6319.xml | 13.7 KB 13.7 KB | Display Display |  EMDB header EMDB header |
| FSC (resolution estimation) |  emd_6319_fsc.xml emd_6319_fsc.xml | 13.2 KB | Display |  FSC data file FSC data file |
| Images |  emd_6319.jpg emd_6319.jpg emd_6319.png emd_6319.png | 29.8 KB 29.8 KB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6319 http://ftp.pdbj.org/pub/emdb/structures/EMD-6319 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6319 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6319 | HTTPS FTP |
-Related structure data
| Related structure data |  3ja6MC  3234C  6320C M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| Related items in Molecule of the Month |
- Map
Map
| File |  Download / File: emd_6319.map.gz / Format: CCP4 / Size: 5.3 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_6319.map.gz / Format: CCP4 / Size: 5.3 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Structure of bacterial chemotaxis signaling CheA2-trimer core complex by cryo-electron tomography and subvolume averaging | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Projections & slices | Image control
Images are generated by Spider. generated in cubic-lattice coordinate | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 3.01 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
CCP4 map header:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-Supplemental data
- Sample components
Sample components
-Entire : tarCF CheA CheW
| Entire | Name: tarCF CheA CheW |
|---|---|
| Components |
|
-Supramolecule #1000: tarCF CheA CheW
| Supramolecule | Name: tarCF CheA CheW / type: sample / ID: 1000 Oligomeric state: Trimer of (CheA dimer, dimer of tarCF trimers of dimers, 2 CheW subunits) Number unique components: 3 |
|---|---|
| Molecular weight | Theoretical: 1.66 MDa |
-Macromolecule #1: Chemotaxis protein CheA
| Macromolecule | Name: Chemotaxis protein CheA / type: protein_or_peptide / ID: 1 / Name.synonym: Bacterial Chemotaxis Histidine Kinase CheA / Number of copies: 6 / Oligomeric state: Dimer / Recombinant expression: Yes |
|---|---|
| Source (natural) | Organism:  |
| Molecular weight | Theoretical: 71.384 KDa |
| Recombinant expression | Organism:  |
| Sequence | UniProtKB: Chemotaxis protein CheA |
-Macromolecule #2: Chemotaxis protein CheW
| Macromolecule | Name: Chemotaxis protein CheW / type: protein_or_peptide / ID: 2 / Number of copies: 2 / Oligomeric state: monomeric / Recombinant expression: Yes |
|---|---|
| Source (natural) | Organism:  |
| Molecular weight | Theoretical: 18.083 KDa |
| Recombinant expression | Organism:  |
| Sequence | UniProtKB: Chemotaxis protein CheW |
-Macromolecule #3: Methyl-accepting chemotaxis protein II
| Macromolecule | Name: Methyl-accepting chemotaxis protein II / type: protein_or_peptide / ID: 3 / Name.synonym: tarCF Details: Cytoplasmic fragment of wild-type aspartate receptor Number of copies: 6 / Oligomeric state: trimer of dimers / Recombinant expression: Yes |
|---|---|
| Source (natural) | Organism:  |
| Molecular weight | Theoretical: 31.209 KDa |
| Recombinant expression | Organism:  |
| Sequence | UniProtKB: Methyl-accepting chemotaxis protein II |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | subtomogram averaging |
| Aggregation state | 2D array |
- Sample preparation
Sample preparation
| Buffer | pH: 7.4 / Details: 75 mM Tris-HCl, 100 mM KCl, 5 mM MgCl2 |
|---|---|
| Grid | Details: Perforated R2/2 Quantifoil grids precoated with 10 nm fiducial gold beads on the backside of the grid |
| Vitrification | Cryogen name: ETHANE / Instrument: HOMEMADE PLUNGER Method: Single-sided blotting to avoid disruption of the monolayer |
| Details | Pseudo-crystalline 2D monolayer reconstituted on a lipid monolayer |
- Electron microscopy
Electron microscopy
| Microscope | FEI POLARA 300 |
|---|---|
| Date | Jan 7, 2009 |
| Image recording | Category: CCD / Film or detector model: GATAN ULTRASCAN 4000 (4k x 4k) / Number real images: 60 / Average electron dose: 60 e/Å2 / Bits/pixel: 16 |
| Electron beam | Acceleration voltage: 200 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Calibrated magnification: 49834 / Illumination mode: SPOT SCAN / Imaging mode: BRIGHT FIELD / Cs: 2.0 mm / Nominal defocus max: 8.0 µm / Nominal defocus min: 4.0 µm / Nominal magnification: 39000 |
| Sample stage | Specimen holder model: OTHER / Tilt series - Axis1 - Min angle: -70 ° / Tilt series - Axis1 - Max angle: 70 ° |
| Experimental equipment |  Model: Tecnai Polara / Image courtesy: FEI Company |
 Movie
Movie Controller
Controller














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)