+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6291 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 16 Angstrom cryo-EM reconstruction of the alpha, beta, gamma TF55 chaperonin | |||||||||
 マップデータ マップデータ | Cryo-EM reconstruction of the Sulfolobus solfataricus TF55 alpha/beta/gamma chaperonin | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | TF55 chaperonin / protein folding / Sulfolobus solfataricus / cryo-EM | |||||||||
| 生物種 |   Sulfolobus solfataricus (古細菌) Sulfolobus solfataricus (古細菌) | |||||||||
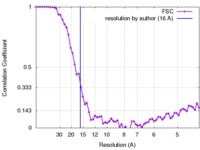
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 16.0 Å | |||||||||
 データ登録者 データ登録者 | Chaston JJ / Stewart AG / Smits C / Aragao D / Struwe W / Benesch J / Xwong A / Ling M / Ashsan B / Sandin S ...Chaston JJ / Stewart AG / Smits C / Aragao D / Struwe W / Benesch J / Xwong A / Ling M / Ashsan B / Sandin S / Rhodes D / Molugu SK / Bernal RA / Stock D | |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2016 ジャーナル: Structure / 年: 2016タイトル: Structural and Functional Insights into the Evolution and Stress Adaptation of Type II Chaperonins. 著者: Jessica J Chaston / Callum Smits / David Aragão / Andrew S W Wong / Bilal Ahsan / Sara Sandin / Sudheer K Molugu / Sanjay K Molugu / Ricardo A Bernal / Daniela Stock / Alastair G Stewart /    要旨: Chaperonins are essential biological complexes assisting protein folding in all kingdoms of life. Whereas homooligomeric bacterial GroEL binds hydrophobic substrates non-specifically, the ...Chaperonins are essential biological complexes assisting protein folding in all kingdoms of life. Whereas homooligomeric bacterial GroEL binds hydrophobic substrates non-specifically, the heterooligomeric eukaryotic CCT binds specifically to distinct classes of substrates. Sulfolobales, which survive in a wide range of temperatures, have evolved three different chaperonin subunits (α, β, γ) that form three distinct complexes tailored for different substrate classes at cold, normal, and elevated temperatures. The larger octadecameric β complexes cater for substrates under heat stress, whereas smaller hexadecameric αβ complexes prevail under normal conditions. The cold-shock complex contains all three subunits, consistent with greater substrate specificity. Structural analysis using crystallography and electron microscopy reveals the geometry of these complexes and shows a novel arrangement of the α and β subunits in the hexadecamer enabling incorporation of the γ subunit. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6291.map.gz emd_6291.map.gz | 3.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6291-v30.xml emd-6291-v30.xml emd-6291.xml emd-6291.xml | 12.1 KB 12.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_6291_fsc.xml emd_6291_fsc.xml | 6.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_6291.jpg emd_6291.jpg | 824.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6291 http://ftp.pdbj.org/pub/emdb/structures/EMD-6291 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6291 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6291 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6291_validation.pdf.gz emd_6291_validation.pdf.gz | 79.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6291_full_validation.pdf.gz emd_6291_full_validation.pdf.gz | 78.2 KB | 表示 | |
| XML形式データ |  emd_6291_validation.xml.gz emd_6291_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6291 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6291 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6291 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6291 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6291.map.gz / 形式: CCP4 / 大きさ: 15.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6291.map.gz / 形式: CCP4 / 大きさ: 15.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
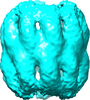
| 注釈 | Cryo-EM reconstruction of the Sulfolobus solfataricus TF55 alpha/beta/gamma chaperonin | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.174 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
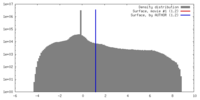
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM reconstruction of the TF55 chaperonin containing the alph...
| 全体 | 名称: Cryo-EM reconstruction of the TF55 chaperonin containing the alpha, beta and gamma subunits |
|---|---|
| 要素 |
|
-超分子 #1000: Cryo-EM reconstruction of the TF55 chaperonin containing the alph...
| 超分子 | 名称: Cryo-EM reconstruction of the TF55 chaperonin containing the alpha, beta and gamma subunits タイプ: sample / ID: 1000 集合状態: Octadecamer composed of 6 alpha, 6 beta, and 6 gamma subunits per complex Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 1.08 MDa / 手法: sequence |
-分子 #1: TF55 chaperonin
| 分子 | 名称: TF55 chaperonin / タイプ: protein_or_peptide / ID: 1 詳細: The TF55 chaperonin is composed of three proteins (alpha, beta, and gamma) arranged in an alternating fashion to form a 9-subunit ring (3 of each protein) which then stacks back-to-back with ...詳細: The TF55 chaperonin is composed of three proteins (alpha, beta, and gamma) arranged in an alternating fashion to form a 9-subunit ring (3 of each protein) which then stacks back-to-back with a second ring, forming an 18-subunit complex. コピー数: 18 / 集合状態: octadecamer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Sulfolobus solfataricus (古細菌) / 株: DSM1617 Sulfolobus solfataricus (古細菌) / 株: DSM1617 |
| 分子量 | 理論値: 1 MDa |
| 組換発現 | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 8 詳細: 20 mM Tris-Cl, pH 8.0, 2 mM MgCl2, 1 mM EDTA, 50 mM NaCl |
| グリッド | 詳細: 400-mesh copper grid with holey carbon film support (Quantifoil R2/2) |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 80 % / チャンバー内温度: 108 K / 装置: HOMEMADE PLUNGER 手法: 3 microliters of sample were blotted off the Quantifoil R2/2 grid for 3 seconds before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 3200FS |
|---|---|
| 温度 | 最低: 83 K / 最高: 103 K / 平均: 93 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 250,000 times magnification. |
| 特殊光学系 | エネルギーフィルター - 名称: JEOL in-column エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 20.0 eV |
| 日付 | 2014年6月20日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) デジタル化 - サンプリング間隔: 15 µm / 実像数: 16534 / 平均電子線量: 25 e/Å2 / ビット/ピクセル: 8 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 69000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 60000 |
| 試料ステージ | 試料ホルダー: 626 Gatan holder / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)