+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6233 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Negative stain single particle electron microscopy of Marburg Virus Glycoproteins bound to antibodies from a human survivor | |||||||||
 マップデータ マップデータ | Reconstruction of Marburg Virus GPdMuc bound to antibody MR78 from a human survivor | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Marburg Virus / Antibodies / Neutralization / Glycoprotein | |||||||||
| 生物種 |  Marburg marburgvirus (ウイルス) / Marburg marburgvirus (ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 22.0 Å | |||||||||
 データ登録者 データ登録者 | Flyak A / Ilinykh PA / Murin CD / Garron T / Shen X / Fusco ML / Hashiguchi T / Bornholdt ZA / Slaughter C / Sapparapu G ...Flyak A / Ilinykh PA / Murin CD / Garron T / Shen X / Fusco ML / Hashiguchi T / Bornholdt ZA / Slaughter C / Sapparapu G / Ksiazek TG / Ward AB / Saphire EO / Bukreyev A / Crowe JE | |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2015 ジャーナル: Cell / 年: 2015タイトル: Mechanism of human antibody-mediated neutralization of Marburg virus. 著者: Andrew I Flyak / Philipp A Ilinykh / Charles D Murin / Tania Garron / Xiaoli Shen / Marnie L Fusco / Takao Hashiguchi / Zachary A Bornholdt / James C Slaughter / Gopal Sapparapu / Curtis ...著者: Andrew I Flyak / Philipp A Ilinykh / Charles D Murin / Tania Garron / Xiaoli Shen / Marnie L Fusco / Takao Hashiguchi / Zachary A Bornholdt / James C Slaughter / Gopal Sapparapu / Curtis Klages / Thomas G Ksiazek / Andrew B Ward / Erica Ollmann Saphire / Alexander Bukreyev / James E Crowe /  要旨: The mechanisms by which neutralizing antibodies inhibit Marburg virus (MARV) are not known. We isolated a panel of neutralizing antibodies from a human MARV survivor that bind to MARV glycoprotein ...The mechanisms by which neutralizing antibodies inhibit Marburg virus (MARV) are not known. We isolated a panel of neutralizing antibodies from a human MARV survivor that bind to MARV glycoprotein (GP) and compete for binding to a single major antigenic site. Remarkably, several of the antibodies also bind to Ebola virus (EBOV) GP. Single-particle EM structures of antibody-GP complexes reveal that all of the neutralizing antibodies bind to MARV GP at or near the predicted region of the receptor-binding site. The presence of the glycan cap or mucin-like domain blocks binding of neutralizing antibodies to EBOV GP, but not to MARV GP. The data suggest that MARV-neutralizing antibodies inhibit virus by binding to infectious virions at the exposed MARV receptor-binding site, revealing a mechanism of filovirus inhibition. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6233.map.gz emd_6233.map.gz | 2.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6233-v30.xml emd-6233-v30.xml emd-6233.xml emd-6233.xml | 11 KB 11 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_6233.jpg emd_6233.jpg | 571.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6233 http://ftp.pdbj.org/pub/emdb/structures/EMD-6233 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6233 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6233 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6233_validation.pdf.gz emd_6233_validation.pdf.gz | 78.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6233_full_validation.pdf.gz emd_6233_full_validation.pdf.gz | 78 KB | 表示 | |
| XML形式データ |  emd_6233_validation.xml.gz emd_6233_validation.xml.gz | 493 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6233 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6233 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6233 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6233 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6233.map.gz / 形式: CCP4 / 大きさ: 3.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6233.map.gz / 形式: CCP4 / 大きさ: 3.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of Marburg Virus GPdMuc bound to antibody MR78 from a human survivor | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.65 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Fab Fragment of MR78 human IgG1 monoclonal antibody to Marburg Vi...
| 全体 | 名称: Fab Fragment of MR78 human IgG1 monoclonal antibody to Marburg Virus GP |
|---|---|
| 要素 |
|
-超分子 #1000: Fab Fragment of MR78 human IgG1 monoclonal antibody to Marburg Vi...
| 超分子 | 名称: Fab Fragment of MR78 human IgG1 monoclonal antibody to Marburg Virus GP タイプ: sample / ID: 1000 / 集合状態: trimer / Number unique components: 2 |
|---|---|
| 分子量 | 理論値: 310 KDa |
-分子 #1: GP, mucin-deleted
| 分子 | 名称: GP, mucin-deleted / タイプ: protein_or_peptide / ID: 1 / Name.synonym: glycoprotein 詳細: Recombinant Marburg Virus GP; mucin-like domains were not included in the construct. コピー数: 1 / 集合状態: trimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Marburg marburgvirus (ウイルス) / 別称: Marburg virus Marburg marburgvirus (ウイルス) / 別称: Marburg virus |
| 分子量 | 理論値: 160 MDa |
| 組換発現 | 生物種:  組換細胞: Schneider S2 cells / 組換プラスミド: pMT-puro |
-分子 #2: Human Monoclonal Antibody IgG1
| 分子 | 名称: Human Monoclonal Antibody IgG1 / タイプ: protein_or_peptide / ID: 2 / Name.synonym: IgG1, monoclonal mAb / 詳細: Human monoclonal antibody IgG1, MR78 / コピー数: 1 / 集合状態: monomer / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human / 組織: Blood / 細胞: Peripheral blood mononuclear cells Homo sapiens (ヒト) / 別称: Human / 組織: Blood / 細胞: Peripheral blood mononuclear cells |
| 分子量 | 理論値: 150 KDa |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.03 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: 20 mM Tris, 150 mM NaCl |
| 染色 | タイプ: NEGATIVE 詳細: Grids with absorbed protein stained with 2% uranyl formate for 30 seconds |
| グリッド | 詳細: 400 mesh copper grid with carbon support, plasma cleaned 20 seconds (Ar/O2 mix, Gatan) |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI SPIRIT |
|---|---|
| 日付 | 2013年11月12日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: OTHER / 実像数: 200 |
| 電子線 | 加速電圧: 120 kV / 電子線源: TUNGSTEN HAIRPIN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 52000 |
| 試料ステージ | 試料ホルダーモデル: OTHER |
| 実験機器 |  モデル: Tecnai Spirit / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | Particles were selected using DoG picker. |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 22.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: EMAN2 / 使用した粒子像数: 12896 |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera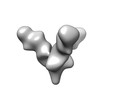









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















