[日本語] English
 万見
万見- EMDB-6016: Dynein motor domain in complex with Lis1 in the presence of ATP and Vi -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6016 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
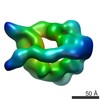
| タイトル | Dynein motor domain in complex with Lis1 in the presence of ATP and Vi | |||||||||
 マップデータ マップデータ | Reconstruction of dynein motor domain in complex with Lis1 in the presence of ATP and Vi | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | dynein / lis1 / regulation mechanism | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報microtubule sliding / karyogamy / microtubule organizing center organization / nuclear migration along microtubule / astral microtubule / establishment of mitotic spindle localization / microtubule plus-end binding / spindle pole body / vesicle transport along microtubule / minus-end-directed microtubule motor activity ...microtubule sliding / karyogamy / microtubule organizing center organization / nuclear migration along microtubule / astral microtubule / establishment of mitotic spindle localization / microtubule plus-end binding / spindle pole body / vesicle transport along microtubule / minus-end-directed microtubule motor activity / microtubule associated complex / cytoplasmic dynein complex / dynein light intermediate chain binding / nuclear migration / dynein complex binding / dynein intermediate chain binding / establishment of mitotic spindle orientation / mitotic sister chromatid segregation / Antigen processing: Ubiquitination & Proteasome degradation / cytoplasmic microtubule / cytoplasmic microtubule organization / Neutrophil degranulation / mitotic spindle organization / kinetochore / spindle pole / nuclear envelope / cell cortex / cell division / ATP hydrolysis activity / ATP binding / identical protein binding / nucleus / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / ネガティブ染色法 / 解像度: 23.1 Å | |||||||||
 データ登録者 データ登録者 | Toropova K / Zou S / Roberts AJ / Redwine WB / Goodman BS / Reck-Peterson SL / Leschziner AE | |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2014 ジャーナル: Elife / 年: 2014タイトル: Lis1 regulates dynein by sterically blocking its mechanochemical cycle. 著者: Katerina Toropova / Sirui Zou / Anthony J Roberts / William B Redwine / Brian S Goodman / Samara L Reck-Peterson / Andres E Leschziner /  要旨: Regulation of cytoplasmic dynein's motor activity is essential for diverse eukaryotic functions, including cell division, intracellular transport, and brain development. The dynein regulator Lis1 is ...Regulation of cytoplasmic dynein's motor activity is essential for diverse eukaryotic functions, including cell division, intracellular transport, and brain development. The dynein regulator Lis1 is known to keep dynein bound to microtubules; however, how this is accomplished mechanistically remains unknown. We have used three-dimensional electron microscopy, single-molecule imaging, biochemistry, and in vivo assays to help establish this mechanism. The three-dimensional structure of the dynein-Lis1 complex shows that binding of Lis1 to dynein's AAA+ ring sterically prevents dynein's main mechanical element, the 'linker', from completing its normal conformational cycle. Single-molecule experiments show that eliminating this block by shortening the linker to a point where it can physically bypass Lis1 renders single dynein motors insensitive to regulation by Lis1. Our data reveal that Lis1 keeps dynein in a persistent microtubule-bound state by directly blocking the progression of its mechanochemical cycle. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6016.map.gz emd_6016.map.gz | 17.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6016-v30.xml emd-6016-v30.xml emd-6016.xml emd-6016.xml | 12.1 KB 12.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  400_6016.gif 400_6016.gif 80_6016.gif 80_6016.gif | 43.6 KB 3.7 KB | ||
| その他 |  emd_6016_half_map_1.map.gz emd_6016_half_map_1.map.gz emd_6016_half_map_2.map.gz emd_6016_half_map_2.map.gz | 17 MB 17 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6016 http://ftp.pdbj.org/pub/emdb/structures/EMD-6016 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6016 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6016 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6016_validation.pdf.gz emd_6016_validation.pdf.gz | 79 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6016_full_validation.pdf.gz emd_6016_full_validation.pdf.gz | 78.1 KB | 表示 | |
| XML形式データ |  emd_6016_validation.xml.gz emd_6016_validation.xml.gz | 495 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6016 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6016 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6016 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6016 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6016.map.gz / 形式: CCP4 / 大きさ: 21.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6016.map.gz / 形式: CCP4 / 大きさ: 21.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of dynein motor domain in complex with Lis1 in the presence of ATP and Vi | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
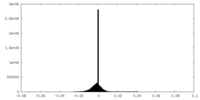
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.73 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-添付マップデータ: emd 6016 half map 1.map
| ファイル | emd_6016_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
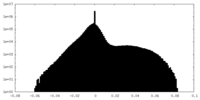
| 密度ヒストグラム |
-添付マップデータ: emd 6016 half map 2.map
| ファイル | emd_6016_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
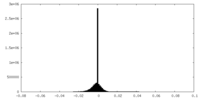
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Dynein motor domain in complex with Lis1 in 500 uM ATP/NaVO4
| 全体 | 名称: Dynein motor domain in complex with Lis1 in 500 uM ATP/NaVO4 |
|---|---|
| 要素 |
|
-超分子 #1000: Dynein motor domain in complex with Lis1 in 500 uM ATP/NaVO4
| 超分子 | 名称: Dynein motor domain in complex with Lis1 in 500 uM ATP/NaVO4 タイプ: sample / ID: 1000 / 集合状態: One motor domain to one Lis1 propeller domain / Number unique components: 2 |
|---|---|
| 分子量 | 実験値: 445 KDa / 理論値: 445 KDa |
-分子 #1: Dynein heavy chain
| 分子 | 名称: Dynein heavy chain / タイプ: protein_or_peptide / ID: 1 / Name.synonym: DYN1 / コピー数: 1 / 集合状態: Monomer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 331 KDa / 理論値: 331 KDa |
| 組換発現 | 生物種:  |
| 配列 | UniProtKB: Dynein heavy chain, cytoplasmic |
-分子 #2: Lis1
| 分子 | 名称: Lis1 / タイプ: protein_or_peptide / ID: 2 / Name.synonym: Pac1 / 詳細: Only a single copy of Lis1 is resolved in the map. / コピー数: 2 / 集合状態: Dimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 57 KDa / 理論値: 57 KDa |
| 組換発現 | 生物種:  |
| 配列 | UniProtKB: Nuclear distribution protein PAC1 |
-実験情報
-構造解析
| 手法 | ネガティブ染色法, クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.03 mg/mL |
|---|---|
| 緩衝液 | pH: 8 詳細: 50 mM Tris-HCl, 150 mM potassium acetate, 2 mM magnesium acetate, 1 mM EGTA, 1 mM DTT, 500 uM Mg-ATP/NaVO4 |
| 染色 | タイプ: NEGATIVE 詳細: Grids with adsorbed protein were floated on 2% w/v uranyl formate, then sandwiched with a thin layer of carbon, blotted, and frozen in liquid nitrogen. |
| グリッド | 詳細: 200 mesh C-flat grid with thin carbon support |
| 凍結 | 凍結剤: NITROGEN / 装置: OTHER 手法: Manually blot, wait 10-20 seconds, then manually plunge into liquid nitrogen. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 20 |
|---|---|
| 日付 | 2013年3月19日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) デジタル化 - サンプリング間隔: 15 µm / 実像数: 103 / 平均電子線量: 25 e/Å2 |
| 電子線 | 加速電圧: 120 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 86800 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 最大 デフォーカス(公称値): 1.2 µm / 最小 デフォーカス(公称値): 0.51 µm / 倍率(公称値): 62000 |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| CTF補正 | 詳細: phase flip |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 23.1 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: RELION 詳細: The dataset was first 3D-classified in RELION. Particles in this map displayed a resolved linker domain at lower contour levels. 使用した粒子像数: 1072 |
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー


























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)