+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-6012 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Electron microscopy of Calsyntenin-3 tetramer conformation II | |||||||||
 マップデータ マップデータ | Cstn-3 High Molecular Weight (HMW) tetramer conformation II | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Synaptic organizer / trans-synaptic bridge | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 16.5 Å | |||||||||
 データ登録者 データ登録者 | Lu Z / Wang Y / Chen F / Tong F / Reddy MVVVS / Luo L / Seshadrinathan S / Zhang L / Holthauzen LMF / Craig AM ...Lu Z / Wang Y / Chen F / Tong F / Reddy MVVVS / Luo L / Seshadrinathan S / Zhang L / Holthauzen LMF / Craig AM / Ren G / Rudenko G | |||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2014 ジャーナル: J Biol Chem / 年: 2014タイトル: Calsyntenin-3 molecular architecture and interaction with neurexin 1α. 著者: Zhuoyang Lu / Yun Wang / Fang Chen / Huimin Tong / M V V V Sekhar Reddy / Lin Luo / Suchithra Seshadrinathan / Lei Zhang / Luis Marcelo F Holthauzen / Ann Marie Craig / Gang Ren / Gabby Rudenko /    要旨: Calsyntenin 3 (Cstn3 or Clstn3), a recently identified synaptic organizer, promotes the development of synapses. Cstn3 localizes to the postsynaptic membrane and triggers presynaptic differentiation. ...Calsyntenin 3 (Cstn3 or Clstn3), a recently identified synaptic organizer, promotes the development of synapses. Cstn3 localizes to the postsynaptic membrane and triggers presynaptic differentiation. Calsyntenin members play an evolutionarily conserved role in memory and learning. Cstn3 was recently shown in cell-based assays to interact with neurexin 1α (n1α), a synaptic organizer that is implicated in neuropsychiatric disease. Interaction would permit Cstn3 and n1α to form a trans-synaptic complex and promote synaptic differentiation. However, it is contentious whether Cstn3 binds n1α directly. To understand the structure and function of Cstn3, we determined its architecture by electron microscopy and delineated the interaction between Cstn3 and n1α biochemically and biophysically. We show that Cstn3 ectodomains form monomers as well as tetramers that are stabilized by disulfide bonds and Ca(2+), and both are probably flexible in solution. We show further that the extracellular domains of Cstn3 and n1α interact directly and that both Cstn3 monomers and tetramers bind n1α with nanomolar affinity. The interaction is promoted by Ca(2+) and requires minimally the LNS domain of Cstn3. Furthermore, Cstn3 uses a fundamentally different mechanism to bind n1α compared with other neurexin partners, such as the synaptic organizer neuroligin 2, because Cstn3 does not strictly require the sixth LNS domain of n1α. Our structural data suggest how Cstn3 as a synaptic organizer on the postsynaptic membrane, particularly in tetrameric form, may assemble radially symmetric trans-synaptic bridges with the presynaptic synaptic organizer n1α to recruit and spatially organize proteins into networks essential for synaptic function. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_6012.map.gz emd_6012.map.gz | 5.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-6012-v30.xml emd-6012-v30.xml emd-6012.xml emd-6012.xml | 10.3 KB 10.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  400_6012.gif 400_6012.gif 80_6012.gif 80_6012.gif | 15.4 KB 2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-6012 http://ftp.pdbj.org/pub/emdb/structures/EMD-6012 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6012 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-6012 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_6012_validation.pdf.gz emd_6012_validation.pdf.gz | 79.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_6012_full_validation.pdf.gz emd_6012_full_validation.pdf.gz | 78.3 KB | 表示 | |
| XML形式データ |  emd_6012_validation.xml.gz emd_6012_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6012 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6012 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6012 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-6012 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_6012.map.gz / 形式: CCP4 / 大きさ: 7.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_6012.map.gz / 形式: CCP4 / 大きさ: 7.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
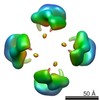
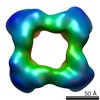
| 注釈 | Cstn-3 High Molecular Weight (HMW) tetramer conformation II | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.88 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
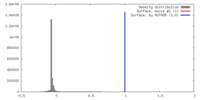
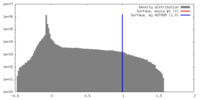
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Cstn-3 Tetramer (High Molecular Weight)
| 全体 | 名称: Cstn-3 Tetramer (High Molecular Weight) |
|---|---|
| 要素 |
|
-超分子 #1000: Cstn-3 Tetramer (High Molecular Weight)
| 超分子 | 名称: Cstn-3 Tetramer (High Molecular Weight) / タイプ: sample / ID: 1000 / 集合状態: Tetramer / Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 352 KDa 手法: Calculated from amino acid content without N-linked glycosylation |
-分子 #1: Calsyntenin-3
| 分子 | 名称: Calsyntenin-3 / タイプ: protein_or_peptide / ID: 1 / Name.synonym: Cstn-3 / コピー数: 4 / 集合状態: Tetramer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human / 組織: neuron / 細胞中の位置: Synapse Homo sapiens (ヒト) / 別称: Human / 組織: neuron / 細胞中の位置: Synapse |
| 分子量 | 理論値: 88 KDa |
| 組換発現 | 生物種:  Trichoplusia ni (イラクサキンウワバ) / 組換株: BTI-TN-5B1-4 / 組換細胞: High Five Trichoplusia ni (イラクサキンウワバ) / 組換株: BTI-TN-5B1-4 / 組換細胞: High Five |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.05 mg/mL |
|---|---|
| 緩衝液 | pH: 8 / 詳細: 25 mM Tris, pH 8.0, 120 mM NaCl, 5 mM CaCl2 |
| 染色 | タイプ: NEGATIVE 詳細: An aliquot (~4 uL) of sample was placed on a grid. After ~1 min incubation, excess solution was blotted with filter paper, and the grid stained for ~1 min by submersion in one drop (~35 uL) ...詳細: An aliquot (~4 uL) of sample was placed on a grid. After ~1 min incubation, excess solution was blotted with filter paper, and the grid stained for ~1 min by submersion in one drop (~35 uL) of 1% w/v uranyl formate (UF) on Parafilm before being nitrogen-air-dried at room temperature. |
| グリッド | 詳細: thin-carbon-coated 200 mesh copper grid (CF200-Cu, EMS), glow-discharged 15 seconds |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | ZEISS LIBRA120PLUS |
|---|---|
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 125,000 times magnification. |
| 日付 | 2012年12月11日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 実像数: 639 / 平均電子線量: 200 e/Å2 |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.2 mm / 最大 デフォーカス(公称値): 0.6 µm / 最小 デフォーカス(公称値): 0.1 µm / 倍率(公称値): 125000 |
| 試料ステージ | 試料ホルダーモデル: OTHER |
- 画像解析
画像解析
| CTF補正 | 詳細: ctffind3 |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 16.5 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: EMAN, IPET / 使用した粒子像数: 6699 |
| 最終 2次元分類 | クラス数: 150 |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera





 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)