+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5864 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Single-particle reconstruction of conformation IV of ligand-free sGC | |||||||||
 マップデータ マップデータ | Single-particle reconstruction of conformation IV of ligand-free sGC | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | soluble guanylate cyclase / conformational heterogeneity | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 30.0 Å | |||||||||
 データ登録者 データ登録者 | Campbell MG / Underbakke ES / Potter CS / Carragher B / Marletta MA | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2014 ジャーナル: Proc Natl Acad Sci U S A / 年: 2014タイトル: Single-particle EM reveals the higher-order domain architecture of soluble guanylate cyclase. 著者: Melody G Campbell / Eric S Underbakke / Clinton S Potter / Bridget Carragher / Michael A Marletta /  要旨: Soluble guanylate cyclase (sGC) is the primary nitric oxide (NO) receptor in mammals and a central component of the NO-signaling pathway. The NO-signaling pathways mediate diverse physiological ...Soluble guanylate cyclase (sGC) is the primary nitric oxide (NO) receptor in mammals and a central component of the NO-signaling pathway. The NO-signaling pathways mediate diverse physiological processes, including vasodilation, neurotransmission, and myocardial functions. sGC is a heterodimer assembled from two homologous subunits, each comprised of four domains. Although crystal structures of isolated domains have been reported, no structure is available for full-length sGC. We used single-particle electron microscopy to obtain the structure of the complete sGC heterodimer and determine its higher-order domain architecture. Overall, the protein is formed of two rigid modules: the catalytic dimer and the clustered Per/Art/Sim and heme-NO/O2-binding domains, connected by a parallel coiled coil at two hinge points. The quaternary assembly demonstrates a very high degree of flexibility. We captured hundreds of individual conformational snapshots of free sGC, NO-bound sGC, and guanosine-5'-[(α,β)-methylene]triphosphate-bound sGC. The molecular architecture and pronounced flexibility observed provides a significant step forward in understanding the mechanism of NO signaling. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5864.map.gz emd_5864.map.gz | 15.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5864-v30.xml emd-5864-v30.xml emd-5864.xml emd-5864.xml | 14.7 KB 14.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5864.jpg emd_5864.jpg | 52.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5864 http://ftp.pdbj.org/pub/emdb/structures/EMD-5864 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5864 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5864 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_5864_validation.pdf.gz emd_5864_validation.pdf.gz | 77.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_5864_full_validation.pdf.gz emd_5864_full_validation.pdf.gz | 76.6 KB | 表示 | |
| XML形式データ |  emd_5864_validation.xml.gz emd_5864_validation.xml.gz | 495 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5864 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5864 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5864 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5864 | HTTPS FTP |
-関連構造データ
| 関連構造データ | 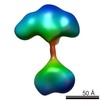 5861C  5862C  5863C  5865C  5866C  5867C  5868C 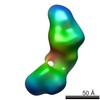 5869C 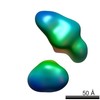 5870C  5871C  5872C  5873C  5874C  5875C 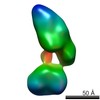 5876C  5877C  5878C 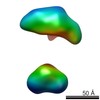 5879C 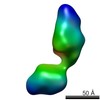 5880C 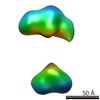 5881C  5882C  5883C  5884C C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5864.map.gz / 形式: CCP4 / 大きさ: 41.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5864.map.gz / 形式: CCP4 / 大きさ: 41.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Single-particle reconstruction of conformation IV of ligand-free sGC | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Soluble Guanylate Cyclase, ligand-free
| 全体 | 名称: Soluble Guanylate Cyclase, ligand-free |
|---|---|
| 要素 |
|
-超分子 #1000: Soluble Guanylate Cyclase, ligand-free
| 超分子 | 名称: Soluble Guanylate Cyclase, ligand-free / タイプ: sample / ID: 1000 / 集合状態: Heterodimer / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 150 KDa / 理論値: 150 KDa |
-分子 #1: Soluble Guanylate Cyclase
| 分子 | 名称: Soluble Guanylate Cyclase / タイプ: protein_or_peptide / ID: 1 / Name.synonym: sGC / コピー数: 1 / 集合状態: Heterodimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 150 KDa / 理論値: 150 KDa |
| 組換発現 | 生物種:  組換細胞: Sf9 組換プラスミド: pFastBac1/sGCALPHA1 and pFastBac1/sGCBETA1 |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 / 詳細: 50 mM TEA, 150 mM NaCl, 5 mM DTT |
|---|---|
| 染色 | タイプ: NEGATIVE 詳細: 3 microliters of sample were applied to grid. The specimen was stained twice with 2% uranyl formate, then allowed to air-dry. |
| グリッド | 詳細: Glow discharged C-flat grid with 2-micron-diameter holes overlaid by thin 1.5 nm continuous carbon |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 温度 | 平均: 298 K |
| 日付 | 2013年1月26日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: TVIPS TEMCAM-F416 (4k x 4k) 実像数: 2204 / 平均電子線量: 35 e/Å2 |
| Tilt angle max | 0 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 1.2 µm / 倍率(公称値): 80000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC / Tilt angle min: -55 |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | See publication |
|---|---|
| CTF補正 | 詳細: Each micrograph |
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 30.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: SPIDER / 使用した粒子像数: 263 |
| 最終 2次元分類 | クラス数: 1 |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)