+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5532 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Single particle tomography of TRiC chaperonin with internalized substrate | |||||||||
 マップデータ マップデータ | Single Particle Tomography average of TRiC chaperonin incubated with mhttQ51 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Mutant huntingtin / TRiC chaperonin / Single Particle Tomography / cryo electron microscopy / amyloid | |||||||||
| 生物種 |  | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 50.0 Å | |||||||||
 データ登録者 データ登録者 | Shahmoradian SH / Galaz JG / Schmid MF / Cong Y / Ma B / Spiess C / Frydman J / Ludtke SJ / Chiu W | |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2013 ジャーナル: Elife / 年: 2013タイトル: TRiC's tricks inhibit huntingtin aggregation. 著者: Sarah H Shahmoradian / Jesus G Galaz-Montoya / Michael F Schmid / Yao Cong / Boxue Ma / Christoph Spiess / Judith Frydman / Steven J Ludtke / Wah Chiu /  要旨: In Huntington's disease, a mutated version of the huntingtin protein leads to cell death. Mutant huntingtin is known to aggregate, a process that can be inhibited by the eukaryotic chaperonin TRiC ...In Huntington's disease, a mutated version of the huntingtin protein leads to cell death. Mutant huntingtin is known to aggregate, a process that can be inhibited by the eukaryotic chaperonin TRiC (TCP1-ring complex) in vitro and in vivo. A structural understanding of the genesis of aggregates and their modulation by cellular chaperones could facilitate the development of therapies but has been hindered by the heterogeneity of amyloid aggregates. Using cryo-electron microscopy (cryoEM) and single particle cryo-electron tomography (SPT) we characterize the growth of fibrillar aggregates of mutant huntingtin exon 1 containing an expanded polyglutamine tract with 51 residues (mhttQ51), and resolve 3-D structures of the chaperonin TRiC interacting with mhttQ51. We find that TRiC caps mhttQ51 fibril tips via the apical domains of its subunits, and also encapsulates smaller mhtt oligomers within its chamber. These two complementary mechanisms provide a structural description for TRiC's inhibition of mhttQ51 aggregation in vitro. DOI:http://dx.doi.org/10.7554/eLife.00710.001. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5532.map.gz emd_5532.map.gz | 3.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5532-v30.xml emd-5532-v30.xml emd-5532.xml emd-5532.xml | 9.9 KB 9.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5532.png emd_5532.png emd_5532_1.png emd_5532_1.png | 148.1 KB 126.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5532 http://ftp.pdbj.org/pub/emdb/structures/EMD-5532 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5532 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5532 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_5532_validation.pdf.gz emd_5532_validation.pdf.gz | 78.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_5532_full_validation.pdf.gz emd_5532_full_validation.pdf.gz | 77.5 KB | 表示 | |
| XML形式データ |  emd_5532_validation.xml.gz emd_5532_validation.xml.gz | 492 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5532 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5532 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5532 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5532 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5532.map.gz / 形式: CCP4 / 大きさ: 3.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5532.map.gz / 形式: CCP4 / 大きさ: 3.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Single Particle Tomography average of TRiC chaperonin incubated with mhttQ51 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.401 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
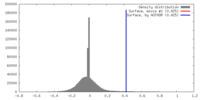
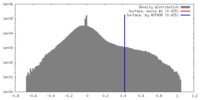
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : TRiC chaperonin + mhttQ51
| 全体 | 名称: TRiC chaperonin + mhttQ51 |
|---|---|
| 要素 |
|
-超分子 #1000: TRiC chaperonin + mhttQ51
| 超分子 | 名称: TRiC chaperonin + mhttQ51 / タイプ: sample / ID: 1000 / Number unique components: 2 |
|---|---|
| 分子量 | 理論値: 1.01 MDa |
-分子 #1: TCP-1 Ring Complex with mutant huntingtin Q51 exon-1
| 分子 | 名称: TCP-1 Ring Complex with mutant huntingtin Q51 exon-1 タイプ: protein_or_peptide / ID: 1 / Name.synonym: TRiC with mhtt / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 1.01 MDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| グリッド | 詳細: 200 mesh copper Quantifoil grid, glow discharged |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 2100 |
|---|---|
| 詳細 | Serial EM software |
| 日付 | 2009年3月19日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: GENERIC CCD / デジタル化 - サンプリング間隔: 2 µm / 実像数: 121 / 平均電子線量: 62 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 5.0 µm / 倍率(公称値): 25000 |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN / Tilt series - Axis1 - Min angle: -60 ° / Tilt series - Axis1 - Max angle: 60 ° |
- 画像解析
画像解析
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 50.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: EMAN2 / 使用したサブトモグラム数: 7 |
|---|---|
| 最終 3次元分類 | クラス数: 1 |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)