+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5525 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Electron cryo-microscopy of ABC BmrA in 12-fold symmetry rings | |||||||||
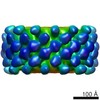
 マップデータ マップデータ | Reconstruction of BmrA in a lipidic environment. Reconstruction is D12 symmetrized. 24 homodimers of BmrA are inserted into a lipid bilayer. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ABC transporter / exporter / bmra / yvcc / transmembrane protein / reconstitution into lipids / membrane protein | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 23.0 Å | |||||||||
 データ登録者 データ登録者 | Fribourg PF / Chami M / Sorzano CO / Gubellini F / Marabini R / Marco S / Jault JM / Levy D | |||||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2014 ジャーナル: J Mol Biol / 年: 2014タイトル: 3D cryo-electron reconstruction of BmrA, a bacterial multidrug ABC transporter in an inward-facing conformation and in a lipidic environment. 著者: Pierre Frederic Fribourg / Mohamed Chami / Carlos Oscar S Sorzano / Francesca Gubellini / Roberto Marabini / Sergio Marco / Jean-Michel Jault / Daniel Lévy /    要旨: ABC (ATP-binding cassette) membrane exporters are efflux transporters of a wide diversity of molecule across the membrane at the expense of ATP. A key issue regarding their catalytic cycle is whether ...ABC (ATP-binding cassette) membrane exporters are efflux transporters of a wide diversity of molecule across the membrane at the expense of ATP. A key issue regarding their catalytic cycle is whether or not their nucleotide-binding domains (NBDs) are physically disengaged in the resting state. To settle this controversy, we obtained structural data on BmrA, a bacterial multidrug homodimeric ABC transporter, in a membrane-embedded state. BmrA in the apostate was reconstituted in lipid bilayers forming a mixture of ring-shaped structures of 24 or 39 homodimers. Three-dimensional models of the ring-shaped structures of 24 or 39 homodimers were calculated at 2.3 nm and 2.5 nm resolution from cryo-electron microscopy, respectively. In these structures, BmrA adopts an inward-facing open conformation similar to that found in mouse P-glycoprotein structure with the NBDs separated by 3 nm. Both lipidic leaflets delimiting the transmembrane domains of BmrA were clearly resolved. In planar membrane sheets, the NBDs were even more separated. BmrA in an ATP-bound conformation was determined from two-dimensional crystals grown in the presence of ATP and vanadate. A projection map calculated at 1.6 nm resolution shows an open outward-facing conformation. Overall, the data are consistent with a mechanism of drug transport involving large conformational changes of BmrA and show that a bacterial ABC exporter can adopt at least two open inward conformations in lipid membrane. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5525.map.gz emd_5525.map.gz | 8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5525-v30.xml emd-5525-v30.xml emd-5525.xml emd-5525.xml | 10.9 KB 10.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5525.tif emd_5525.tif | 732.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5525 http://ftp.pdbj.org/pub/emdb/structures/EMD-5525 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5525 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5525 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_5525_validation.pdf.gz emd_5525_validation.pdf.gz | 78.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_5525_full_validation.pdf.gz emd_5525_full_validation.pdf.gz | 77.6 KB | 表示 | |
| XML形式データ |  emd_5525_validation.xml.gz emd_5525_validation.xml.gz | 492 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5525 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5525 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5525 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5525 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5525.map.gz / 形式: CCP4 / 大きさ: 29.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5525.map.gz / 形式: CCP4 / 大きさ: 29.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of BmrA in a lipidic environment. Reconstruction is D12 symmetrized. 24 homodimers of BmrA are inserted into a lipid bilayer. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.5 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : BmrA reconstituted at high density into lipid bilayer.
| 全体 | 名称: BmrA reconstituted at high density into lipid bilayer. |
|---|---|
| 要素 |
|
-超分子 #1000: BmrA reconstituted at high density into lipid bilayer.
| 超分子 | 名称: BmrA reconstituted at high density into lipid bilayer. タイプ: sample / ID: 1000 集合状態: 24 Homodimers of BmrA inserted into lipid bilayer Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 3.12 MDa / 理論値: 3.12 MDa |
-分子 #1: BmrA
| 分子 | 名称: BmrA / タイプ: protein_or_peptide / ID: 1 / Name.synonym: YvcC / 詳細: 24 homodimers of inserted into lipid bilayer / コピー数: 24 / 集合状態: Dimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 3.12 MDa / 理論値: 3.12 MDa |
| 組換発現 | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.8 / 詳細: 50mM HEPES, 100mM NaCl |
| グリッド | 詳細: Ted Pella inc. holey formvar grid |
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER 詳細: specimen linked to a Ni++-NTA-DOGS/DOPC lipid monolayer 手法: Blot for 3 seconds before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS CM200FEG |
|---|---|
| 日付 | 2003年1月1日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: NIKON COOLSCAN / 実像数: 102 / 平均電子線量: 15 e/Å2 / ビット/ピクセル: 8 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 50000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.26 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 2.6 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| 詳細 | Xmipp CL2D classification and Projection matching refinement with Invert Fourier 3D reconstruction |
|---|---|
| CTF補正 | 詳細: Micrograph based |
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 23.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: Xmipp / 使用した粒子像数: 1188 |
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | Protocol: Rigid body |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: Cross correlation factor |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)






















