+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5495 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM reconstruction of Sputnik virus | |||||||||
 マップデータ マップデータ | Reconstruction of Sputnik virus full particles at 3.5A resolution | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Jelly roll structure / major capsid protein / penton protein / minor capsid protein | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Sputnik virophage (スプートニクヴィロファージ) Sputnik virophage (スプートニクヴィロファージ) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Zhang XZ / Sun SY / Xiang Y / Wong J / Klose T / Raoult D / Rossmann MG | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2012 ジャーナル: Proc Natl Acad Sci U S A / 年: 2012タイトル: Structure of Sputnik, a virophage, at 3.5-Å resolution. 著者: Xinzheng Zhang / Siyang Sun / Ye Xiang / Jimson Wong / Thomas Klose / Didier Raoult / Michael G Rossmann /  要旨: "Sputnik" is a dsDNA virus, referred to as a virophage, that is coassembled with Mimivirus in the host amoeba. We have used cryo-EM to produce an electron density map of the icosahedral Sputnik virus ..."Sputnik" is a dsDNA virus, referred to as a virophage, that is coassembled with Mimivirus in the host amoeba. We have used cryo-EM to produce an electron density map of the icosahedral Sputnik virus at 3.5-Å resolution, sufficient to verify the identity of most amino acids in the capsid proteins and to establish the identity of the pentameric protein forming the fivefold vertices. It was also shown that the virus lacks an internal membrane. The capsid is organized into a T = 27 lattice in which there are 260 trimeric capsomers and 12 pentameric capsomers. The trimeric capsomers consist of three double "jelly-roll" major capsid proteins creating pseudohexameric capsomer symmetry. The pentameric capsomers consist of five single jelly-roll proteins. The release of the genome by displacing one or more of the pentameric capsomers may be the result of a low-pH environment. These results suggest a mechanism of Sputnik DNA ejection that probably also occurs in other big icosahedral double jelly-roll viruses such as Adenovirus. In this study, the near-atomic resolution structure of a virus has been established where crystallization for X-ray crystallography was not feasible. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5495.map.gz emd_5495.map.gz | 302.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5495-v30.xml emd-5495-v30.xml emd-5495.xml emd-5495.xml | 9.5 KB 9.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5495_1.jpg emd_5495_1.jpg | 438.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5495 http://ftp.pdbj.org/pub/emdb/structures/EMD-5495 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5495 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5495 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_5495_validation.pdf.gz emd_5495_validation.pdf.gz | 353 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_5495_full_validation.pdf.gz emd_5495_full_validation.pdf.gz | 352.6 KB | 表示 | |
| XML形式データ |  emd_5495_validation.xml.gz emd_5495_validation.xml.gz | 4.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5495 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5495 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5495 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5495 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5495.map.gz / 形式: CCP4 / 大きさ: 843.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5495.map.gz / 形式: CCP4 / 大きさ: 843.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of Sputnik virus full particles at 3.5A resolution | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
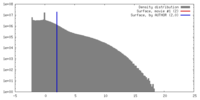
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Sputnik
| 全体 | 名称: Sputnik |
|---|---|
| 要素 |
|
-超分子 #1000: Sputnik
| 超分子 | 名称: Sputnik / タイプ: sample / ID: 1000 / Number unique components: 1 |
|---|
-超分子 #1: Sputnik virophage
| 超分子 | 名称: Sputnik virophage / タイプ: virus / ID: 1 / 詳細: Sputnik virus full particle / NCBI-ID: 543939 / 生物種: Sputnik virophage / データベース: NCBI / ウイルスタイプ: SATELLITE / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Acanthamoeba (真核生物) / 別称: PROTOZOA Acanthamoeba (真核生物) / 別称: PROTOZOA |
| ウイルス殻 | Shell ID: 1 / 名称: Vp20 / 直径: 750 Å / T番号(三角分割数): 27 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7 / 詳細: PBS |
| グリッド | 詳細: 400 mesh Cu grid (c-flat), 1.2 um hole, glow discharged |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 100 K / 装置: GATAN CRYOPLUNGE 3 / 手法: blot 6 seconds |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| アライメント法 | Legacy - 非点収差: corrected at 250,000 magnification |
| 日付 | 2011年1月1日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM デジタル化 - スキャナー: NIKON SUPER COOLSCAN 9000 デジタル化 - サンプリング間隔: 6.35 µm / 平均電子線量: 22 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 1.1 µm / 倍率(公称値): 59000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | Using EMAN |
|---|---|
| CTF補正 | 詳細: each particle |
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 3.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: EMAN, Frealign / 使用した粒子像数: 12000 |
| 最終 角度割当 | 詳細: EMAN |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)