+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5448 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-tomography and subtomogram averaging of the Newcastle disease virus matrix protein array | |||||||||
 マップデータ マップデータ | Subtomogram average of Newcastle disease virus matrix protein | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | virus assembly / matrix protein / pleomorphic virus structure / paramyxovirus / viral membrane / cryo-tomography | |||||||||
| 生物種 |  Newcastle disease virus (ウイルス) Newcastle disease virus (ウイルス) | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 45.6 Å | |||||||||
 データ登録者 データ登録者 | Battisti AJ / Meng G / Winkler DC / McGinnes LW / Plevka P / Steven AC / Morrison TG / Rossmann MG | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2012 ジャーナル: Proc Natl Acad Sci U S A / 年: 2012タイトル: Structure and assembly of a paramyxovirus matrix protein. 著者: Anthony J Battisti / Geng Meng / Dennis C Winkler / Lori W McGinnes / Pavel Plevka / Alasdair C Steven / Trudy G Morrison / Michael G Rossmann /  要旨: Many pleomorphic, lipid-enveloped viruses encode matrix proteins that direct their assembly and budding, but the mechanism of this process is unclear. We have combined X-ray crystallography and ...Many pleomorphic, lipid-enveloped viruses encode matrix proteins that direct their assembly and budding, but the mechanism of this process is unclear. We have combined X-ray crystallography and cryoelectron tomography to show that the matrix protein of Newcastle disease virus, a paramyxovirus and relative of measles virus, forms dimers that assemble into pseudotetrameric arrays that generate the membrane curvature necessary for virus budding. We show that the glycoproteins are anchored in the gaps between the matrix proteins and that the helical nucleocapsids are associated in register with the matrix arrays. About 90% of virions lack matrix arrays, suggesting that, in agreement with previous biological observations, the matrix protein needs to dissociate from the viral membrane during maturation, as is required for fusion and release of the nucleocapsid into the host's cytoplasm. Structure and sequence conservation imply that other paramyxovirus matrix proteins function similarly. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5448.map.gz emd_5448.map.gz | 169.8 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5448-v30.xml emd-5448-v30.xml emd-5448.xml emd-5448.xml | 11.4 KB 11.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5448.tif emd_5448.tif | 161 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5448 http://ftp.pdbj.org/pub/emdb/structures/EMD-5448 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5448 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5448 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_5448_validation.pdf.gz emd_5448_validation.pdf.gz | 78.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_5448_full_validation.pdf.gz emd_5448_full_validation.pdf.gz | 77.4 KB | 表示 | |
| XML形式データ |  emd_5448_validation.xml.gz emd_5448_validation.xml.gz | 495 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5448 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5448 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5448 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5448 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5448.map.gz / 形式: CCP4 / 大きさ: 1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5448.map.gz / 形式: CCP4 / 大きさ: 1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Subtomogram average of Newcastle disease virus matrix protein | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 7.5 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Newcastle disease virus matrix protein array from subtomogram ave...
| 全体 | 名称: Newcastle disease virus matrix protein array from subtomogram averaging |
|---|---|
| 要素 |
|
-超分子 #1000: Newcastle disease virus matrix protein array from subtomogram ave...
| 超分子 | 名称: Newcastle disease virus matrix protein array from subtomogram averaging タイプ: sample / ID: 1000 / 集合状態: an array of dimers / Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 76 KDa |
-超分子 #1: Newcastle disease virus
| 超分子 | 名称: Newcastle disease virus / タイプ: virus / ID: 1 / 詳細: Newcastle disease virus strain B1 / NCBI-ID: 11176 / 生物種: Newcastle disease virus / データベース: NCBI / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
- 試料調製
試料調製
| 緩衝液 | pH: 8 / 詳細: 0.02 M Tris, 0.12 M NaCl, 0.001 M EDTA |
|---|---|
| グリッド | 詳細: 200 mesh holey carbon copper grids (Quantifoil Micro Tools, GmbH, Germany) |
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 19,500 times magnification |
| 特殊光学系 | エネルギーフィルター - 名称: Tridiem GIF 863 エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 30.0 eV |
| 日付 | 2010年10月19日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 1000 (2k x 2k) デジタル化 - サンプリング間隔: 15.0 µm / 実像数: 87 / 平均電子線量: 163 e/Å2 / ビット/ピクセル: 16 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 20000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 8.0 µm / 最小 デフォーカス(公称値): 8.0 µm / 倍率(公称値): 19500 |
| 試料ステージ | 試料ホルダーモデル: OTHER / Tilt series - Axis1 - Min angle: -64.5 ° / Tilt series - Axis1 - Max angle: 64.5 ° |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | Images aligned using colloidal gold fiducial markers and reconstructed using the weighted back-projection method as implemented in EMAN. Average number of tilts used in the 3D reconstructions: 87. Average tomographic tilt angle increment: 1.5. |
|---|---|
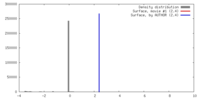
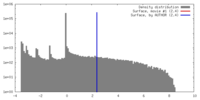
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 45.6 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: IMOD, Uppsala_Software_Factory 詳細: Tomographic reconstruction showed an array of matrix protein subunits, which were averaged to reduce noise. Two-fold symmetry enforced for individual subunits. Membrane density masked out. |
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - #0 - Chain ID: A / Chain - #1 - Chain ID: B |
|---|---|
| ソフトウェア | 名称: EMfit |
| 詳細 | Protocol: Rigid body. 10 matrix protein dimers were simultaneously fitted into the matrix array density using EMfit |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: sumF, clash |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)