+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5433 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | dATP-inhibited class Ia ribonucleotide reductase from E. coli: alpha4beta4 open conformation 3 | |||||||||
 マップデータ マップデータ | alpha4beta4 open conformation 3 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ribonucleotide reductase / allostery / inhibition / nucleotide metabolism | |||||||||
| 機能・相同性 | ribonucleoside-diphosphate reductase complex 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 31.5 Å | |||||||||
 データ登録者 データ登録者 | Zimanyi CM / Ando N / Brignole E / Asturias FJ / Stubbe J / Drennan CL | |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2012 ジャーナル: Structure / 年: 2012タイトル: Tangled up in knots: structures of inactivated forms of E. coli class Ia ribonucleotide reductase. 著者: Christina M Zimanyi / Nozomi Ando / Edward J Brignole / Francisco J Asturias / Joanne Stubbe / Catherine L Drennan /  要旨: Ribonucleotide reductases (RNRs) provide the precursors for DNA biosynthesis and repair and are successful targets for anticancer drugs such as clofarabine and gemcitabine. Recently, we reported that ...Ribonucleotide reductases (RNRs) provide the precursors for DNA biosynthesis and repair and are successful targets for anticancer drugs such as clofarabine and gemcitabine. Recently, we reported that dATP inhibits E. coli class Ia RNR by driving formation of RNR subunits into α4β4 rings. Here, we present the first X-ray structure of a gemcitabine-inhibited E. coli RNR and show that the previously described α4β4 rings can interlock to form an unprecedented (α4β4)2 megacomplex. This complex is also seen in a higher-resolution dATP-inhibited RNR structure presented here, which employs a distinct crystal lattice from that observed in the gemcitabine-inhibited case. With few reported examples of protein catenanes, we use data from small-angle X-ray scattering and electron microscopy to both understand the solution conditions that contribute to concatenation in RNRs as well as present a mechanism for the formation of these unusual structures. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5433.map.gz emd_5433.map.gz | 1.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5433-v30.xml emd-5433-v30.xml emd-5433.xml emd-5433.xml | 11.7 KB 11.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5433_1.png emd_5433_1.png | 82.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5433 http://ftp.pdbj.org/pub/emdb/structures/EMD-5433 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5433 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5433 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_5433_validation.pdf.gz emd_5433_validation.pdf.gz | 78.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_5433_full_validation.pdf.gz emd_5433_full_validation.pdf.gz | 77.6 KB | 表示 | |
| XML形式データ |  emd_5433_validation.xml.gz emd_5433_validation.xml.gz | 493 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5433 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5433 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5433 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5433 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5433.map.gz / 形式: CCP4 / 大きさ: 1.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5433.map.gz / 形式: CCP4 / 大きさ: 1.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
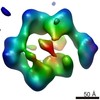
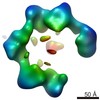
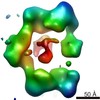
| 注釈 | alpha4beta4 open conformation 3 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X: 4.36 Å / Y: 4.36 Å / Z: 6.54 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : E. coli Class Ia ribonucleotide reductase
| 全体 | 名称: E. coli Class Ia ribonucleotide reductase |
|---|---|
| 要素 |
|
-超分子 #1000: E. coli Class Ia ribonucleotide reductase
| 超分子 | 名称: E. coli Class Ia ribonucleotide reductase / タイプ: sample / ID: 1000 集合状態: two alpha2 subunits in complex with two beta2 subunits Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 517 KDa / 理論値: 517 KDa / 手法: Calculated from amino acid sequence of subunits |
-分子 #1: E. coli Class Ia ribonucleotide reductase
| 分子 | 名称: E. coli Class Ia ribonucleotide reductase / タイプ: protein_or_peptide / ID: 1 / Name.synonym: RNR / 詳細: alpha4beta4 open conformation / コピー数: 1 / 集合状態: hetero-octamer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 517 KDa / 理論値: 517 KDa |
| 組換発現 | 生物種:  |
| 配列 | GO: ribonucleoside-diphosphate reductase complex |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.6 詳細: 50 mM HEPES, 15 mM MgCl2, 1 mM EDTA, 1 mM CDP, 0.05 mM dATP |
|---|---|
| 染色 | タイプ: NEGATIVE 詳細: 5ul protein, washed immediately 3x [2% uranyl acetate, 0.2% trehalose], carbon sandwich |
| グリッド | 詳細: thin carbon support on 300 mesh Cu/Rh grid, glow discharge in amylamine |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 日付 | 2010年7月31日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 実像数: 89 / 平均電子線量: 25 e/Å2 / 詳細: images acquired as tilt-pairs / ビット/ピクセル: 16 |
| 電子線 | 加速電圧: 120 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 1.801 µm / 最小 デフォーカス(公称値): 0.275 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: room temperature / 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC / Tilt angle min: -59.1 / Tilt angle max: -54.3 |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | Semi-automated particle selection from untilted images with EMAN2. Particles matched in tilted images using modified TiltPicker. Processed in SPIDER. |
|---|---|
| CTF補正 | 詳細: each particle |
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 31.5 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: SPIDER / 使用した粒子像数: 389 |
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - #0 - Chain ID: A / Chain - #1 - Chain ID: B / Chain - #2 - Chain ID: D / Chain - #3 - Chain ID: E |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | Protocol: rigid body. Subunits iteratively fit with Chimera |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: correlation |
-原子モデル構築 2
| 初期モデル | PDB ID: Chain - #0 - Chain ID: A / Chain - #1 - Chain ID: B |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | Protocol: rigid body. Subunits iteratively fit with Chimera |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: correlation |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)