+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5429 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Negative stain 3D EM of MCM2-7 from Encephalitozoon cuniculi | |||||||||
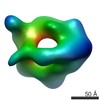
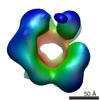
 マップデータ マップデータ | Single-particle 3D reconstruction of EcuMCM2-7 with ATPgammaS | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | DNA replication / replicative helicase / MCM2-7 / ATPase / hexameric motor | |||||||||
| 生物種 |  Encephalitozoon cuniculi (ウサギエンケファリトゾーン) Encephalitozoon cuniculi (ウサギエンケファリトゾーン) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 24.0 Å | |||||||||
 データ登録者 データ登録者 | Lyubimov AY / Costa A / Bleichert F / Botchan MR / Berger JM | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2012 ジャーナル: Proc Natl Acad Sci U S A / 年: 2012タイトル: ATP-dependent conformational dynamics underlie the functional asymmetry of the replicative helicase from a minimalist eukaryote. 著者: Artem Y Lyubimov / Alessandro Costa / Franziska Bleichert / Michael R Botchan / James M Berger /  要旨: The heterohexameric minichromosome maintenance (MCM2-7) complex is an ATPase that serves as the central replicative helicase in eukaryotes. During initiation, the ring-shaped MCM2-7 particle is ...The heterohexameric minichromosome maintenance (MCM2-7) complex is an ATPase that serves as the central replicative helicase in eukaryotes. During initiation, the ring-shaped MCM2-7 particle is thought to open to facilitate loading onto DNA. The conformational state accessed during ring opening, the interplay between ATP binding and MCM2-7 architecture, and the use of these events in the regulation of DNA unwinding are poorly understood. To address these issues in isolation from the regulatory complexity of existing eukaryotic model systems, we investigated the structure/function relationships of a naturally minimized MCM2-7 complex from the microsporidian parasite Encephalitozoon cuniculi. Electron microscopy and small-angle X-ray scattering studies show that, in the absence of ATP, MCM2-7 spontaneously adopts a left-handed, open-ring structure. Nucleotide binding does not promote ring closure but does cause the particle to constrict in a two-step process that correlates with the filling of high- and low-affinity ATPase sites. Our findings support the idea that an open ring forms the default conformational state of the isolated MCM2-7 complex, and they provide a structural framework for understanding the multiphasic ATPase kinetics observed in different MCM2-7 systems. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5429.map.gz emd_5429.map.gz | 869 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5429-v30.xml emd-5429-v30.xml emd-5429.xml emd-5429.xml | 14.3 KB 14.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5429.png emd_5429.png | 94.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5429 http://ftp.pdbj.org/pub/emdb/structures/EMD-5429 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5429 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5429 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_5429_validation.pdf.gz emd_5429_validation.pdf.gz | 78.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_5429_full_validation.pdf.gz emd_5429_full_validation.pdf.gz | 77.7 KB | 表示 | |
| XML形式データ |  emd_5429_validation.xml.gz emd_5429_validation.xml.gz | 493 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5429 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5429 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5429 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5429 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5429.map.gz / 形式: CCP4 / 大きさ: 1001 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5429.map.gz / 形式: CCP4 / 大きさ: 1001 KB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Single-particle 3D reconstruction of EcuMCM2-7 with ATPgammaS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 3.56 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Encephalitozoon cuniculi MCM2-7 complex bound to ATPgammaS
| 全体 | 名称: Encephalitozoon cuniculi MCM2-7 complex bound to ATPgammaS |
|---|---|
| 要素 |
|
-超分子 #1000: Encephalitozoon cuniculi MCM2-7 complex bound to ATPgammaS
| 超分子 | 名称: Encephalitozoon cuniculi MCM2-7 complex bound to ATPgammaS タイプ: sample / ID: 1000 / 集合状態: MCM2-7 heterohexamer / Number unique components: 6 |
|---|---|
| 分子量 | 理論値: 480 KDa |
-分子 #1: Minichromosome maintenance protein 2
| 分子 | 名称: Minichromosome maintenance protein 2 / タイプ: protein_or_peptide / ID: 1 / Name.synonym: Mcm2 / コピー数: 1 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Encephalitozoon cuniculi (ウサギエンケファリトゾーン) Encephalitozoon cuniculi (ウサギエンケファリトゾーン) |
| 分子量 | 理論値: 88 KDa |
| 組換発現 | 生物種:  組換プラスミド: pFastBac |
-分子 #2: Minichromosome maintenance protein 3
| 分子 | 名称: Minichromosome maintenance protein 3 / タイプ: protein_or_peptide / ID: 2 / Name.synonym: Mcm3 / コピー数: 1 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Encephalitozoon cuniculi (ウサギエンケファリトゾーン) Encephalitozoon cuniculi (ウサギエンケファリトゾーン) |
| 分子量 | 理論値: 77 KDa |
| 組換発現 | 生物種:  組換プラスミド: pFastBac |
-分子 #3: Minichromosome maintenance protein 4
| 分子 | 名称: Minichromosome maintenance protein 4 / タイプ: protein_or_peptide / ID: 3 / Name.synonym: Mcm4 / コピー数: 1 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Encephalitozoon cuniculi (ウサギエンケファリトゾーン) Encephalitozoon cuniculi (ウサギエンケファリトゾーン) |
| 分子量 | 理論値: 80 KDa |
| 組換発現 | 生物種:  組換プラスミド: pFastBac |
-分子 #4: Minichromosome maintenance protein 5
| 分子 | 名称: Minichromosome maintenance protein 5 / タイプ: protein_or_peptide / ID: 4 / Name.synonym: Mcm5 / コピー数: 1 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Encephalitozoon cuniculi (ウサギエンケファリトゾーン) Encephalitozoon cuniculi (ウサギエンケファリトゾーン) |
| 分子量 | 理論値: 79 KDa |
| 組換発現 | 生物種:  組換プラスミド: pFastBac |
-分子 #5: Minichromosome maintenance protein 6
| 分子 | 名称: Minichromosome maintenance protein 6 / タイプ: protein_or_peptide / ID: 5 / Name.synonym: Mcm6 / コピー数: 1 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Encephalitozoon cuniculi (ウサギエンケファリトゾーン) Encephalitozoon cuniculi (ウサギエンケファリトゾーン) |
| 分子量 | 理論値: 82 KDa |
| 組換発現 | 生物種:  組換プラスミド: pFastBac |
-分子 #6: Minichromosome maintenance protein 7
| 分子 | 名称: Minichromosome maintenance protein 7 / タイプ: protein_or_peptide / ID: 6 / Name.synonym: Mcm7 / コピー数: 1 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Encephalitozoon cuniculi (ウサギエンケファリトゾーン) Encephalitozoon cuniculi (ウサギエンケファリトゾーン) |
| 分子量 | 理論値: 77 KDa |
| 組換発現 | 生物種:  組換プラスミド: pFastBac |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.0 mg/mL |
|---|---|
| 緩衝液 | pH: 8 詳細: 300 mM NaOAc, 50 mM imidazole, pH 8.0, 5 mM Mg(OAc)2, 0.2 mM TCEP, 10% glycerol, 10 mM ATPgammaS |
| 染色 | タイプ: NEGATIVE 詳細: Grids with adsorbed protein (diluted to 0.03 mg/mL) floated on 2% uranyl formate for 10 sec (x 5 times) |
| グリッド | 詳細: 400 mesh copper grids, nitrocellulose amyl acetate support, glow discharged, holey |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 12 |
|---|---|
| 日付 | 2011年1月5日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: GENERIC GATAN / 実像数: 16532 / 平均電子線量: 20 e/Å2 |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 30000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
- 画像解析
画像解析
| 詳細 | Particles selected automatically using DoG Picker and Batchboxer in the APPION environment |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 24.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: EMAN2, SPARX 詳細: Nine iterative rounds were performed, with the output of each round used as starting model (after filtering) for the subsequent round until convergence. Convergence was determined by lack of ...詳細: Nine iterative rounds were performed, with the output of each round used as starting model (after filtering) for the subsequent round until convergence. Convergence was determined by lack of further changes in volume features and distortion of FSC at higher resolution. 使用した粒子像数: 16532 |
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | Protocol: Rigid body. The 6 protomers were fit using sequential volume-to-volume fitting in Chimera |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)






















