+ Open data
Open data
- Basic information
Basic information
| Entry | Database: EMDB / ID: EMD-5379 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
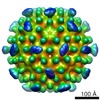
| Title | Cryo-EM 3D reconstruction of naturally empty Triatoma Virus | |||||||||
 Map data Map data | 3D reconstruction of naturally empty TrV particle | |||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | Triatoma virus / Dicistrovirus / cryo-electron microscopy / 3D reconstruction | |||||||||
| Biological species |  Triatoma virus Triatoma virus | |||||||||
| Method | single particle reconstruction / cryo EM / Resolution: 19.0 Å | |||||||||
 Authors Authors | Neumann E / Agirre J / Goret G / LeGoff M / Marti GA / Navaza J / Guerin DMA | |||||||||
 Citation Citation |  Journal: J Gen Virol / Year: 2013 Journal: J Gen Virol / Year: 2013Title: Cryo-electron microscopy reconstructions of triatoma virus particles: a clue to unravel genome delivery and capsid disassembly. Authors: J Agirre / G Goret / M LeGoff / R Sánchez-Eugenia / G A Marti / J Navaza / D M A Guérin / E Neumann /    Abstract: Triatoma virus (TrV) is a member of the insect virus family Dicistroviridae and consists of a small, non-enveloped capsid that encloses its positive-sense ssRNA genome. Using cryo-transmission ...Triatoma virus (TrV) is a member of the insect virus family Dicistroviridae and consists of a small, non-enveloped capsid that encloses its positive-sense ssRNA genome. Using cryo-transmission electron microscopy and three-dimensional reconstruction techniques combined with fitting of the available crystallographic models, this study analysed the capsids corresponding to mature and several RNA-empty TrV particles. After genome release, the resulting reconstruction of the empty capsids displayed no prominent conformational changes with respect to the full virion capsid. The results showed that RNA delivery led to empty capsids with an apparent overall intact protein shell and suggested that, in a subsequent step, empty capsids disassemble into small symmetrical particles. Contrary to what is observed upon genome release in mammalian picornaviruses, the empty TrV capsid maintained a protein shell thickness and size identical to that in full virions. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Movie |
 Movie viewer Movie viewer |
|---|---|
| Structure viewer | EM map:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| Supplemental images |
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_5379.map.gz emd_5379.map.gz | 11.6 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-5379-v30.xml emd-5379-v30.xml emd-5379.xml emd-5379.xml | 8.6 KB 8.6 KB | Display Display |  EMDB header EMDB header |
| Images |  emd_5379_1.tif emd_5379_1.tif | 166.2 KB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5379 http://ftp.pdbj.org/pub/emdb/structures/EMD-5379 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5379 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5379 | HTTPS FTP |
-Related structure data
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- Map
Map
| File |  Download / File: emd_5379.map.gz / Format: CCP4 / Size: 61.8 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_5379.map.gz / Format: CCP4 / Size: 61.8 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | 3D reconstruction of naturally empty TrV particle | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
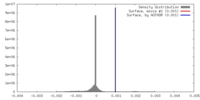
| Voxel size | X=Y=Z: 1.77 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
CCP4 map header:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-Supplemental data
- Sample components
Sample components
-Entire : Naturally empty Triatoma Virus
| Entire | Name: Naturally empty Triatoma Virus |
|---|---|
| Components |
|
-Supramolecule #1000: Naturally empty Triatoma Virus
| Supramolecule | Name: Naturally empty Triatoma Virus / type: sample / ID: 1000 / Number unique components: 1 |
|---|
-Supramolecule #1: Triatoma virus
| Supramolecule | Name: Triatoma virus / type: virus / ID: 1 / Name.synonym: TrV / NCBI-ID: 103442 / Sci species name: Triatoma virus / Database: NCBI / Virus type: OTHER / Virus isolate: OTHER / Virus enveloped: No / Virus empty: Yes / Syn species name: TrV |
|---|---|
| Host (natural) | synonym: INVERTEBRATES |
| Virus shell | Shell ID: 1 / T number (triangulation number): 1 |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Buffer | pH: 7.4 / Details: NMT (10 mM NaCl, 1 mM MgCl2, 50 mM Tris-HCl) |
|---|---|
| Grid | Details: 400 mesh quantifoil grid |
| Vitrification | Cryogen name: ETHANE / Instrument: OTHER / Details: Vitrification instrument: Vitrobot FEI |
- Electron microscopy
Electron microscopy
| Microscope | FEI/PHILIPS CM200T |
|---|---|
| Image recording | Category: FILM / Film or detector model: KODAK SO-163 FILM / Digitization - Scanner: ZEISS SCAI / Digitization - Sampling interval: 7 µm / Number real images: 15 |
| Electron beam | Acceleration voltage: 200 kV / Electron source: LAB6 |
| Electron optics | Calibrated magnification: 39500 / Illumination mode: OTHER / Imaging mode: BRIGHT FIELD / Cs: 2 mm / Nominal defocus max: 4.0 µm / Nominal defocus min: 1.2 µm / Nominal magnification: 38000 |
| Sample stage | Specimen holder: Side entry liquid nitrogen-cooled cryo specimen holder Specimen holder model: GATAN LIQUID NITROGEN |
- Image processing
Image processing
| Final reconstruction | Resolution.type: BY AUTHOR / Resolution: 19.0 Å / Resolution method: FSC 0.5 CUT-OFF / Software - Name: EM3DR2 / Number images used: 695 |
|---|
 Movie
Movie Controller
Controller



 UCSF Chimera
UCSF Chimera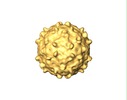











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)