+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5274 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Molecular Structure of Unliganded Native CP-MAC gp120 trimer: Spike region | |||||||||
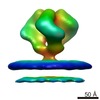
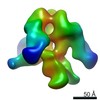
 マップデータ マップデータ | 3D Average of Envelope Glycoproteins from SIV CP-MAC - structure (20 Angstrom) determined by cryo-electron tomography combined with 3D averaging | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Antigens / CD4 / cryo-electron microscopy / HIV / SIV / envelope glycoprotein / gp120 / gp41 / HIV-1 / Immunoglobulin Fab Fragments | |||||||||
| 生物種 | unidentified (未定義) | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 20.0 Å | |||||||||
 データ登録者 データ登録者 | White TA / Bartesaghi A / Borgnia M / Subramaniam S | |||||||||
 引用 引用 |  ジャーナル: PLoS Pathog / 年: 2010 ジャーナル: PLoS Pathog / 年: 2010タイトル: Molecular architectures of trimeric SIV and HIV-1 envelope glycoproteins on intact viruses: strain-dependent variation in quaternary structure. 著者: Tommi A White / Alberto Bartesaghi / Mario J Borgnia / Joel R Meyerson / M Jason V de la Cruz / Julian W Bess / Rachna Nandwani / James A Hoxie / Jeffrey D Lifson / Jacqueline L S Milne / Sriram Subramaniam /  要旨: The initial step in target cell infection by human, and the closely related simian immunodeficiency viruses (HIV and SIV, respectively) occurs with the binding of trimeric envelope glycoproteins (Env) ...The initial step in target cell infection by human, and the closely related simian immunodeficiency viruses (HIV and SIV, respectively) occurs with the binding of trimeric envelope glycoproteins (Env), composed of heterodimers of the viral transmembrane glycoprotein (gp41) and surface glycoprotein (gp120) to target T-cells. Knowledge of the molecular structure of trimeric Env on intact viruses is important both for understanding the molecular mechanisms underlying virus-cell interactions and for the design of effective immunogen-based vaccines to combat HIV/AIDS. Previous analyses of intact HIV-1 BaL virions have already resulted in structures of trimeric Env in unliganded and CD4-liganded states at ~20 Å resolution. Here, we show that the molecular architectures of trimeric Env from SIVmneE11S, SIVmac239 and HIV-1 R3A strains are closely comparable to that previously determined for HIV-1 BaL, with the V1 and V2 variable loops located at the apex of the spike, close to the contact zone between virus and cell. The location of the V1/V2 loops in trimeric Env was definitively confirmed by structural analysis of HIV-1 R3A virions engineered to express Env with deletion of these loops. Strikingly, in SIV CP-MAC, a CD4-independent strain, trimeric Env is in a constitutively "open" conformation with gp120 trimers splayed out in a conformation similar to that seen for HIV-1 BaL Env when it is complexed with sCD4 and the CD4i antibody 17b. Our findings suggest a structural explanation for the molecular mechanism of CD4-independent viral entry and further establish that cryo-electron tomography can be used to discover distinct, functionally relevant quaternary structures of Env displayed on intact viruses. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5274.map.gz emd_5274.map.gz | 1.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5274-v30.xml emd-5274-v30.xml emd-5274.xml emd-5274.xml | 11.1 KB 11.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5274_1.jpg emd_5274_1.jpg | 79.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5274 http://ftp.pdbj.org/pub/emdb/structures/EMD-5274 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5274 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5274 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_5274_validation.pdf.gz emd_5274_validation.pdf.gz | 77.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_5274_full_validation.pdf.gz emd_5274_full_validation.pdf.gz | 76.5 KB | 表示 | |
| XML形式データ |  emd_5274_validation.xml.gz emd_5274_validation.xml.gz | 493 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5274 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5274 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5274 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5274 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5274.map.gz / 形式: CCP4 / 大きさ: 3.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5274.map.gz / 形式: CCP4 / 大きさ: 3.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3D Average of Envelope Glycoproteins from SIV CP-MAC - structure (20 Angstrom) determined by cryo-electron tomography combined with 3D averaging | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : SIV CP-MAC virus
| 全体 | 名称: SIV CP-MAC virus |
|---|---|
| 要素 |
|
-超分子 #1000: SIV CP-MAC virus
| 超分子 | 名称: SIV CP-MAC virus / タイプ: sample / ID: 1000 詳細: Structures determined by cryo-electron tomography combined with 3D averaging 集合状態: trimer / Number unique components: 1 |
|---|
-分子 #1: envelope glycoprotein from Simian Immunodeficiency virus CP-MAC
| 分子 | 名称: envelope glycoprotein from Simian Immunodeficiency virus CP-MAC タイプ: protein_or_peptide / ID: 1 / Name.synonym: Env 詳細: Envelope glycoproteins present on the surface of intact virions. 集合状態: trimer / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) / 株: SIV CP-MAC / 別称: Simian Immunodeficiency Virus / 細胞: SupT1 / 細胞中の位置: viral membrane |
| 分子量 | 理論値: 480 KDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 / 詳細: TNE Buffer (10 mM Tris, 150 mM NaCl, 1 mM EDTA) |
|---|---|
| グリッド | 詳細: 200 mesh Quantifoil Multi A |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 77 K / 装置: FEI VITROBOT MARK III 詳細: Vitrification instrument: Mark III Vitrobot (FEI, Netherlands) 手法: blot for 6 seconds, at 25 C, 100 percent humidity, blot offset of -2, into an ethane slurry cooled by liquid nitrogen |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 温度 | 最低: 81 K / 最高: 82 K / 平均: 81 K |
| 特殊光学系 | エネルギーフィルター - 名称: GATAN GIF エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 20.0 eV |
| 詳細 | Areas were selected for proper ice thickness (about 200 nm). |
| 日付 | 2009年7月21日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: GENERIC GATAN / 平均電子線量: 150 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 2.5 µm / 倍率(公称値): 34000 |
| 試料ステージ | 試料ホルダー: Cartridge / 試料ホルダーモデル: GATAN LIQUID NITROGEN / Tilt series - Axis1 - Min angle: -70 ° / Tilt series - Axis1 - Max angle: 70 ° |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | Average tomographic tilt angle increment: 1. |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 20.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称:  IMOD IMOD詳細: Please see A. Bartesaghi, et al. Journal of Structural Biology, 2008 |
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称:  UCSF Chimera UCSF Chimera |
| 詳細 | Protocol: Rigid body. Automated fitting procedures |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: correlation coefficient |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)






















