+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Sporosarcina pasteurii urease | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Urease / enzyme / Nickel / urea / HYDROLASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報urease complex / urease / urease activity / urea catabolic process / nickel cation binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Sporosarcina pasteurii (バクテリア) Sporosarcina pasteurii (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.12 Å | |||||||||
 データ登録者 データ登録者 | Mazzei L / Tria G / Ciurli S / Cianci M | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Int J Biol Macromol / 年: 2024 ジャーナル: Int J Biol Macromol / 年: 2024タイトル: Exploring the conformational space of the mobile flap in Sporosarcina pasteurii urease by cryo-electron microscopy. 著者: Luca Mazzei / Giancarlo Tria / Stefano Ciurli / Michele Cianci /  要旨: To fully understand enzymatic dynamics, it is essential to explore the complete conformational space of a biological catalyst. The catalytic mechanism of the nickel-dependent urease, the most ...To fully understand enzymatic dynamics, it is essential to explore the complete conformational space of a biological catalyst. The catalytic mechanism of the nickel-dependent urease, the most efficient enzyme known, holds significant relevance for medical, pharmaceutical, and agro-environmental applications. A critical aspect of urease function is the conformational change of a helix-turn-helix motif that covers the active site cavity, known as the mobile flap. This motif has been observed in either an open or a closed conformation through X-ray crystallography studies and has been proposed to stabilize the coordination of a urea molecule to the essential dinuclear Ni(II) cluster in the active site, a requisite for subsequent substrate hydrolysis. This study employs cryo-electron microscopy (cryo-EM) to investigate the transient states within the conformational space of the mobile flap, devoid of the possible constraints of crystallization conditions and solid-state effects. By comparing two cryo-EM structures of Sporosarcina pasteurii urease, one in its native form and the other inhibited by N-(n-butyl) phosphoric triamide (NBPTO), we have unprecedently identified an intermediate state between the open and the catalytically efficient closed conformation of the helix-turn-helix motif, suggesting a role of its tip region in this transition between the two states. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_51450.map.gz emd_51450.map.gz | 10.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-51450-v30.xml emd-51450-v30.xml emd-51450.xml emd-51450.xml | 20 KB 20 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_51450_fsc.xml emd_51450_fsc.xml | 8.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_51450.png emd_51450.png | 77.4 KB | ||
| Filedesc metadata |  emd-51450.cif.gz emd-51450.cif.gz | 6 KB | ||
| その他 |  emd_51450_additional_1.map.gz emd_51450_additional_1.map.gz emd_51450_half_map_1.map.gz emd_51450_half_map_1.map.gz emd_51450_half_map_2.map.gz emd_51450_half_map_2.map.gz | 428.7 KB 59.4 MB 59.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-51450 http://ftp.pdbj.org/pub/emdb/structures/EMD-51450 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-51450 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-51450 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_51450_validation.pdf.gz emd_51450_validation.pdf.gz | 784.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_51450_full_validation.pdf.gz emd_51450_full_validation.pdf.gz | 783.8 KB | 表示 | |
| XML形式データ |  emd_51450_validation.xml.gz emd_51450_validation.xml.gz | 16 KB | 表示 | |
| CIF形式データ |  emd_51450_validation.cif.gz emd_51450_validation.cif.gz | 21.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-51450 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-51450 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-51450 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-51450 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9gmlMC  9gnrC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_51450.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_51450.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.96 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_51450_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_51450_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_51450_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : native Sporosarcina pasteurii urease
| 全体 | 名称: native Sporosarcina pasteurii urease |
|---|---|
| 要素 |
|
-超分子 #1: native Sporosarcina pasteurii urease
| 超分子 | 名称: native Sporosarcina pasteurii urease / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  Sporosarcina pasteurii (バクテリア) Sporosarcina pasteurii (バクテリア) |
| 分子量 | 理論値: 250 KDa |
-分子 #1: Urease subunit gamma
| 分子 | 名称: Urease subunit gamma / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: urease |
|---|---|
| 由来(天然) | 生物種:  Sporosarcina pasteurii (バクテリア) Sporosarcina pasteurii (バクテリア) |
| 分子量 | 理論値: 11.134895 KDa |
| 配列 | 文字列: (CXM)HLNPAEKEK LQIFLASELA LKRKARGLKL NYPEAVAIIT SFIMEGARDG KTVAMLMEEG KHVLTRDDVM EGVPEM IDD IQAEATFPDG TKLVTVHNPI S UniProtKB: Urease subunit gamma |
-分子 #2: Urease subunit beta
| 分子 | 名称: Urease subunit beta / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号: urease |
|---|---|
| 由来(天然) | 生物種:  Sporosarcina pasteurii (バクテリア) Sporosarcina pasteurii (バクテリア) |
| 分子量 | 理論値: 13.975539 KDa |
| 配列 | 文字列: MSNNNYIVPG EYRVAEGEIE INAGREKTTI RVSNTGDRPI QVGSHIHFVE VNKELLFDRA EGIGRRLNIP SGTAARFEPG EEMEVELTE LGGNREVFGI SDLTNGSVDN KELILQRAKE LGYKGVE UniProtKB: Urease subunit beta |
-分子 #3: Urease subunit alpha
| 分子 | 名称: Urease subunit alpha / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO / EC番号: urease |
|---|---|
| 由来(天然) | 生物種:  Sporosarcina pasteurii (バクテリア) Sporosarcina pasteurii (バクテリア) |
| 分子量 | 理論値: 61.575648 KDa |
| 配列 | 文字列: MKINRQQYAE SYGPTVGDQV RLADTDLWIE VEKDYTTYGD EANFGGGKVL REGMGENGTY TRTENVLDLL LTNALILDYT GIYKADIGV KDGYIVGIGK GGNPDIMDGV TPNMIVGTAT EVIAAEGKIV TAGGIDTHVH FINPDQVDVA LANGITTLFG G GTGPAEGS ...文字列: MKINRQQYAE SYGPTVGDQV RLADTDLWIE VEKDYTTYGD EANFGGGKVL REGMGENGTY TRTENVLDLL LTNALILDYT GIYKADIGV KDGYIVGIGK GGNPDIMDGV TPNMIVGTAT EVIAAEGKIV TAGGIDTHVH FINPDQVDVA LANGITTLFG G GTGPAEGS KATTVTPGPW NIEKMLKSTE GLPINVGILG KGHGSSIAPI MEQIDAGAAG L(KCX)IHEDWGAT PASIDRSL T VADEADVQVA IHSDTLNEAG FLEDTLRAIN GRVIHSFHVE GAGGGHAPDI MAMAGHPNVL PSSTNPTRPF TVNTIDEHL DMLMVCHHLK QNIPEDVAFA DSRIRPETIA AEDILHDLGI ISMMSTDALA MGRAGEMVLR TWQTADKMKK QRGPLAEEKN GSDNFRAKR YVSKYTINPA IAQGIAHEVG SIEEGKFADL VLWEPKFFGV KADRVIKGGI IAYAQIGDPS ASIPTPQPVM G RRMYGTVG DLIHDTNITF MSKSSIQQGV PAKLGLKRRI GTVKNCRNIG KKDMKWNDVT TDIDINPETY EVKVDGEVLT CE PVKELPM AQRYFLF UniProtKB: Urease subunit alpha |
-分子 #4: NICKEL (II) ION
| 分子 | 名称: NICKEL (II) ION / タイプ: ligand / ID: 4 / コピー数: 2 / 式: NI |
|---|---|
| 分子量 | 理論値: 58.693 Da |
| Chemical component information |  ChemComp-NI: |
-分子 #5: HYDROXIDE ION
| 分子 | 名称: HYDROXIDE ION / タイプ: ligand / ID: 5 / コピー数: 1 / 式: OH |
|---|---|
| 分子量 | 理論値: 17.007 Da |
| Chemical component information | 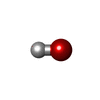 ChemComp-OH: |
-分子 #6: water
| 分子 | 名称: water / タイプ: ligand / ID: 6 / コピー数: 2 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 50 mM HEPES buffer, at pH 7.5 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 283 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 平均露光時間: 25.0 sec. / 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.2 µm / 最小 デフォーカス(公称値): 0.8 µm |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-9gml: |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)













































