+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-5133 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 17 angstrom of wild Rabbit Hemorrhagic DiseaseVirus core-like particle cryomicroscopy structure | |||||||||
 マップデータ マップデータ | This is the cryo-electron microscopy reconstruction of the wild rabbit hemorrhagic disease virus core-like particles. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | RHDV / vp60 / vp10 / core-like particles | |||||||||
| 生物種 |  rabbit hemorrhagic disease virus core like particles (ウイルス) rabbit hemorrhagic disease virus core like particles (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 17.0 Å | |||||||||
 データ登録者 データ登録者 | Hu Z / Tian X / Zhai Y / Xu W / Zheng D / Sun F | |||||||||
 引用 引用 |  ジャーナル: Protein Cell / 年: 2010 ジャーナル: Protein Cell / 年: 2010タイトル: Cryo-electron microscopy reconstructions of two types of wild rabbit hemorrhagic disease viruses characterized the structural features of Lagovirus. 著者: Zhongjun Hu / Xiaojuan Tian / Yujia Zhai / Wei Xu / Dong Zheng / Fei Sun /  要旨: Rabbit hemorrhagic disease was described in China in 1984 and can cause hemorrhagic necrosis of the liver within two or three days after infection. The etiological agent, rabbit hemorrhagic disease ...Rabbit hemorrhagic disease was described in China in 1984 and can cause hemorrhagic necrosis of the liver within two or three days after infection. The etiological agent, rabbit hemorrhagic disease virus (RHDV), belongs to the Lagovirus genus in the Caliciviridae family. Compared to other calicivirus, such as rNV and SMSV, the structure of Lagovirus members is not well characterized. In this report, structures of two types of wild RHDV particles, the intact virion and the core-like particle (CLP), were reconstructed by cryo-electron microscopy at 11 &0A and 17 &0A, respectively. This is the first time the 3D structure of wild caliciviruses CLP has been provided, and the 3D structure of intact RHDV virion is the highest resolution structure in Lagovirus. Comparison of the intact virion and CLP structures clearly indicated that CLP was produced from the intact virion with the protrusion dissociated. In contrast with the crystal structures of recombinant Norovirus and San Miguel sea lion virus, the capsomers of RHDV virion exhibited unique structural features and assembly modes. Both P1 and P2 subdomains have interactions inside the AB capsomer, while only P2 subdomains have interaction inside CC capsomer. The pseudo atomic models of RHDV capsomers were constructed by homology modeling and density map fitting, and the rotation of RHDV VP60 P domain with respect to its S domain, compared with SMSV, was observed. Collectively, our cryo-electron microscopic studies of RHDV provide close insight into the structure of Lagovirus, which is important for functional analysis and better vaccine development in the future. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_5133.map.gz emd_5133.map.gz | 11.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-5133-v30.xml emd-5133-v30.xml emd-5133.xml emd-5133.xml | 10.2 KB 10.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_5133_1.jpg emd_5133_1.jpg | 51.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-5133 http://ftp.pdbj.org/pub/emdb/structures/EMD-5133 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5133 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-5133 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_5133_validation.pdf.gz emd_5133_validation.pdf.gz | 78.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_5133_full_validation.pdf.gz emd_5133_full_validation.pdf.gz | 78 KB | 表示 | |
| XML形式データ |  emd_5133_validation.xml.gz emd_5133_validation.xml.gz | 493 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5133 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5133 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5133 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-5133 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_5133.map.gz / 形式: CCP4 / 大きさ: 15.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_5133.map.gz / 形式: CCP4 / 大きさ: 15.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is the cryo-electron microscopy reconstruction of the wild rabbit hemorrhagic disease virus core-like particles. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.54 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
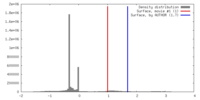
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : wild Rabbit Hemorrhagic Disease Viruses core-like particles
| 全体 | 名称: wild Rabbit Hemorrhagic Disease Viruses core-like particles |
|---|---|
| 要素 |
|
-超分子 #1000: wild Rabbit Hemorrhagic Disease Viruses core-like particles
| 超分子 | 名称: wild Rabbit Hemorrhagic Disease Viruses core-like particles タイプ: sample / ID: 1000 / 集合状態: icosahedral / Number unique components: 3 |
|---|---|
| 分子量 | 理論値: 7.2 MDa |
-超分子 #1: rabbit hemorrhagic disease virus core like particles
| 超分子 | 名称: rabbit hemorrhagic disease virus core like particles タイプ: virus / ID: 1 Name.synonym: rabbit hemorrhagic disease virus core like particles 生物種: rabbit hemorrhagic disease virus core like particles データベース: NCBI / ウイルスタイプ: VIROID / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: No Syn species name: rabbit hemorrhagic disease virus core like particles |
|---|---|
| 宿主 | 生物種:  |
| 分子量 | 理論値: 7.2 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: S / 直径: 320 Å / T番号(三角分割数): 3 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| グリッド | 詳細: 300 mesh holygrid |
|---|---|
| 凍結 | 凍結剤: ETHANE / 装置: OTHER 手法: Embeded in thin layer of vitreous ice on freshly carbon-coated home-made holey EM grid by manually blotting the grids with filter paper and then plunging into liquid ethane cooled by liquid nitrogen |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 20 |
|---|---|
| 温度 | 平均: 105 K |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - サンプリング間隔: 12.7 µm / 実像数: 230 / 平均電子線量: 20 e/Å2 / ビット/ピクセル: 16 |
| 電子線 | 加速電圧: 200 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 2.0 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダー: Eucentric / 試料ホルダーモデル: GATAN HELIUM |
- 画像解析
画像解析
| CTF補正 | 詳細: Each particle |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 17.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: Eman spider / 使用した粒子像数: 895 |
| 最終 2次元分類 | クラス数: 21 |
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称:  chimera chimera |
| 詳細 | Protocol: Rigid Body |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: correlation coefficient |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)