+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Density for a protein trimer unit bound to RNA from a single-layered nucleoprotein-RNA helical assembly from Marburg virus | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | RNA-binding protein / VIRAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報viral RNA genome packaging / helical viral capsid / viral budding via host ESCRT complex / viral nucleocapsid / host cell cytoplasm / ribonucleoprotein complex / RNA binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Marburg virus - Musoke, Kenya, 1980 (ウイルス) / Marburg virus - Musoke, Kenya, 1980 (ウイルス) /  Marburg virus - Musoke (ウイルス) Marburg virus - Musoke (ウイルス) | |||||||||
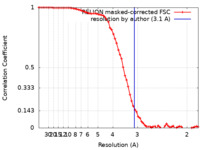
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Zinzula L / Beck F / Camasta M / Bohn S / Liu C / Morado D / Bracher A / Plitzko JM / Baumeister W | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Cryo-EM structure of single-layered nucleoprotein-RNA complex from Marburg virus. 著者: Luca Zinzula / Florian Beck / Marianna Camasta / Stefan Bohn / Chuan Liu / Dustin Morado / Andreas Bracher / Juergen M Plitzko / Wolfgang Baumeister /    要旨: Marburg virus (MARV) causes lethal hemorrhagic fever in humans, posing a threat to global health. We determined by cryogenic electron microscopy (cryo-EM) the MARV helical ribonucleoprotein (RNP) ...Marburg virus (MARV) causes lethal hemorrhagic fever in humans, posing a threat to global health. We determined by cryogenic electron microscopy (cryo-EM) the MARV helical ribonucleoprotein (RNP) complex structure in single-layered conformation, which differs from the previously reported structure of a double-layered helix. Our findings illuminate novel RNP interactions and expand knowledge on MARV genome packaging and nucleocapsid assembly, both processes representing attractive targets for the development of antiviral therapeutics against MARV disease. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_50804.map.gz emd_50804.map.gz | 20.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-50804-v30.xml emd-50804-v30.xml emd-50804.xml emd-50804.xml | 17.1 KB 17.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_50804_fsc.xml emd_50804_fsc.xml | 6.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_50804.png emd_50804.png | 71.5 KB | ||
| マスクデータ |  emd_50804_msk_1.map emd_50804_msk_1.map | 27 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-50804.cif.gz emd-50804.cif.gz | 5.5 KB | ||
| その他 |  emd_50804_half_map_1.map.gz emd_50804_half_map_1.map.gz emd_50804_half_map_2.map.gz emd_50804_half_map_2.map.gz | 20.7 MB 20.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-50804 http://ftp.pdbj.org/pub/emdb/structures/EMD-50804 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50804 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50804 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_50804_validation.pdf.gz emd_50804_validation.pdf.gz | 890.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_50804_full_validation.pdf.gz emd_50804_full_validation.pdf.gz | 890.2 KB | 表示 | |
| XML形式データ |  emd_50804_validation.xml.gz emd_50804_validation.xml.gz | 12.3 KB | 表示 | |
| CIF形式データ |  emd_50804_validation.cif.gz emd_50804_validation.cif.gz | 17 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50804 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50804 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50804 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50804 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9fvdC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_50804.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_50804.map.gz / 形式: CCP4 / 大きさ: 27 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.93 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_50804_msk_1.map emd_50804_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_50804_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_50804_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Helical assembly of nucleoprotein-RNA complex from Marburg virus
| 全体 | 名称: Helical assembly of nucleoprotein-RNA complex from Marburg virus |
|---|---|
| 要素 |
|
-超分子 #1: Helical assembly of nucleoprotein-RNA complex from Marburg virus
| 超分子 | 名称: Helical assembly of nucleoprotein-RNA complex from Marburg virus タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Marburg virus - Musoke, Kenya, 1980 (ウイルス) Marburg virus - Musoke, Kenya, 1980 (ウイルス) |
-分子 #1: N protein from Marburg virus
| 分子 | 名称: N protein from Marburg virus / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Marburg virus - Musoke (ウイルス) Marburg virus - Musoke (ウイルス) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GMDLHSLLEL GTKPTAPHVR NKKVILFDTN HQVSICNQII DAINSGIDLG DLLEGGLLTL CVEHYYNSDK DKFNTSPIAK YLRDAGYEFD VIKNADATRF LDVIPNEPHY SPLILALKTL ESTESQRGRI GLFLSFCSLF LPKLVVGDRA SIEKALRQVT VHQEQGIVTY ...文字列: GMDLHSLLEL GTKPTAPHVR NKKVILFDTN HQVSICNQII DAINSGIDLG DLLEGGLLTL CVEHYYNSDK DKFNTSPIAK YLRDAGYEFD VIKNADATRF LDVIPNEPHY SPLILALKTL ESTESQRGRI GLFLSFCSLF LPKLVVGDRA SIEKALRQVT VHQEQGIVTY PNHWLTTGHM KVIFGILRSS FILKFVLIHQ GVNLVTGHDA YDSIISNSVG QTRFSGLLIV KTVLEFILQK TDSGVTLHPL VRTSKVKNEV ASFKQALSNL ARHGEYAPFA RVLNLSGINN LEHGLYPQLS AIALGVATAH GSTLAGVNVG EQYQQLREAA HDAEVKLQRR HEHQEIQAIA EDDEERKILE QFHLQKTEIT HSQTLAVLSQ KREKLARLAA EIENNIVEDQ GFKQSQNRVS QSFLNDPTPV EVTVQARPMN RVEHHHHHHH H UniProtKB: Nucleoprotein |
-分子 #2: RNA (5'-R(*AP*GP*AP*CP*AP*CP*AP*CP*AP*AP*AP*AP*AP*CP*AP*AP*GP*A)-3')
| 分子 | 名称: RNA (5'-R(*AP*GP*AP*CP*AP*CP*AP*CP*AP*AP*AP*AP*AP*CP*AP*AP*GP*A)-3') タイプ: rna / ID: 2 |
|---|---|
| 由来(天然) | 生物種:  Marburg virus - Musoke, Kenya, 1980 (ウイルス) Marburg virus - Musoke, Kenya, 1980 (ウイルス) |
| 配列 | 文字列: AGACACACAA AAACAAGA |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 濃度 | 1.25 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||
| 凍結 | 凍結剤: ETHANE-PROPANE / チャンバー内湿度: 95 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 実像数: 5518 / 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: AlphaFold / Chain - Initial model type: in silico model |
|---|---|
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)