+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the reduced cytochrome bd oxidase from M. tuberculosis | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Ubiquinone / Demethylmenaquinone / Cryo-EM / Respiration / Substrate specificity / Disulfide regulation / OXIDOREDUCTASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cytochrome complex / aerobic electron transport chain / oxidoreductase activity, acting on diphenols and related substances as donors, oxygen as acceptor / electron transfer activity / heme binding / metal ion binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Mycobacterium tuberculosis H37Rv (結核菌) Mycobacterium tuberculosis H37Rv (結核菌) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.96 Å | |||||||||
 データ登録者 データ登録者 | Kayastha K / Bruenle S | |||||||||
| 資金援助 |  オランダ, 1件 オランダ, 1件
| |||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2025 ジャーナル: J Biol Chem / 年: 2025タイトル: Menaquinone-specific turnover by Mycobacterium tuberculosis cytochrome bd is redox regulated by the Q-loop disulfide bond. 著者: Tijn T van der Velden / Kanwal Kayastha / Caspar Y J Waterham / Steffen Brünle / Lars J C Jeuken /  要旨: Cytochrome bd from Mycobacterium tuberculosis (Mtbd) is a menaquinol oxidase that has gained interest as an antibiotic target because of its importance in survival under infectious conditions. Mtbd ...Cytochrome bd from Mycobacterium tuberculosis (Mtbd) is a menaquinol oxidase that has gained interest as an antibiotic target because of its importance in survival under infectious conditions. Mtbd contains a characteristic disulfide bond that has been hypothesized to allow for Mtbd activity regulation at the enzymatic level, possibly helping M. tuberculosis to rapidly adapt to the hostile environment of the phagosome. Here, the role of the disulfide bond and quinone specificity have been determined by reconstitution of a minimal respiratory chain and the single-particle cryo-EM structure in the disulfide-reduced form. Mtbd was shown to be specific for menaquinone, while regulation by reduction of the Q-loop disulfide bond decreased oxidase activity up to 90%. Structural analysis shows that a salt bridge unique to Mtbd keeps the Q-loop partially structured in its disulfide-reduced form, which could facilitate the rapid activation of Mtbd upon exposure to reactive oxygen species. We signify Mtbd as the first redox sensory terminal oxidase and propose that this helps M. tuberculosis in the defense against reactive oxygen species encountered during infection. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_50520.map.gz emd_50520.map.gz | 31.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-50520-v30.xml emd-50520-v30.xml emd-50520.xml emd-50520.xml | 22.1 KB 22.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_50520_fsc.xml emd_50520_fsc.xml | 11.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_50520.png emd_50520.png | 148.2 KB | ||
| マスクデータ |  emd_50520_msk_1.map emd_50520_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-50520.cif.gz emd-50520.cif.gz | 7.5 KB | ||
| その他 |  emd_50520_half_map_1.map.gz emd_50520_half_map_1.map.gz emd_50520_half_map_2.map.gz emd_50520_half_map_2.map.gz | 59.5 MB 59.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-50520 http://ftp.pdbj.org/pub/emdb/structures/EMD-50520 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50520 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50520 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_50520_validation.pdf.gz emd_50520_validation.pdf.gz | 731.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_50520_full_validation.pdf.gz emd_50520_full_validation.pdf.gz | 730.8 KB | 表示 | |
| XML形式データ |  emd_50520_validation.xml.gz emd_50520_validation.xml.gz | 15.9 KB | 表示 | |
| CIF形式データ |  emd_50520_validation.cif.gz emd_50520_validation.cif.gz | 21.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50520 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50520 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50520 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-50520 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  9fkaMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_50520.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_50520.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.836 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_50520_msk_1.map emd_50520_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_50520_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_50520_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cytochrome BD-I oxidase
| 全体 | 名称: Cytochrome BD-I oxidase |
|---|---|
| 要素 |
|
-超分子 #1: Cytochrome BD-I oxidase
| 超分子 | 名称: Cytochrome BD-I oxidase / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 詳細: Cytochrome BD-I oxidase bound with Heme B, cis-Heme D, Menaquinone-9, Cardiolipins, PE lipid |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 942 KDa |
-分子 #1: Probable integral membrane cytochrome D ubiquinol oxidase (Subuni...
| 分子 | 名称: Probable integral membrane cytochrome D ubiquinol oxidase (Subunit I) CydA (Cytochrome BD-I oxidase subunit I) タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mycobacterium tuberculosis H37Rv (結核菌) Mycobacterium tuberculosis H37Rv (結核菌) |
| 分子量 | 理論値: 53.863098 KDa |
| 組換発現 | 生物種:  Mycolicibacterium smegmatis (バクテリア) Mycolicibacterium smegmatis (バクテリア) |
| 配列 | 文字列: MNVVDISRWQ FGITTVYHFI FVPLTIGLAP LIAVMQTLWV VTDNPAWYRL TKFFGKLFLI NFAIGVATGI VQEFQFGMNW SEYSRFVGD VFGAPLAMEG LAAFFFESTF IGLWIFGWNR LPRLVHLACI WIVAIAVNVS AFFIIAANSF MQHPVGAHYN P TTGRAELS ...文字列: MNVVDISRWQ FGITTVYHFI FVPLTIGLAP LIAVMQTLWV VTDNPAWYRL TKFFGKLFLI NFAIGVATGI VQEFQFGMNW SEYSRFVGD VFGAPLAMEG LAAFFFESTF IGLWIFGWNR LPRLVHLACI WIVAIAVNVS AFFIIAANSF MQHPVGAHYN P TTGRAELS SIVVLLTNNT AQAAFTHTVS GALLTAGTFV AAVSAWWLVR SSTTHADSDT QAMYRPATIL GCWVALAATA GL LFTGDHQ GKLMFQQQPM KMASAESLCD TQTDPNFSVL TVGRQNNCDS LTRVIEVPYV LPFLAEGRIS GVTLQGIRDL QQE YQQRFG PNDYRPNLFV TYWSFRMMIG LMAIPVLFAL IALWLTRGGQ IPNQRWFSWL ALLTMPAPFL ANSAGWVFTE MGRQ PWVVV PNPTGDQLVR LTVKAGVSDH SATVVATSLL MFTLVYAVLA VIWCWLLKRY IVEGPLEHDA EPAAHGAPRD DEVAP LSFA Y UniProtKB: Probable integral membrane cytochrome D ubiquinol oxidase (Subunit I) CydA (Cytochrome BD-I oxidase subunit I) |
-分子 #2: Probable integral membrane cytochrome D ubiquinol oxidase (Subuni...
| 分子 | 名称: Probable integral membrane cytochrome D ubiquinol oxidase (Subunit II) CydB (Cytochrome BD-I oxidase subunit II) タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Mycobacterium tuberculosis H37Rv (結核菌) Mycobacterium tuberculosis H37Rv (結核菌) |
| 分子量 | 理論値: 37.650957 KDa |
| 組換発現 | 生物種:  Mycolicibacterium smegmatis (バクテリア) Mycolicibacterium smegmatis (バクテリア) |
| 配列 | 文字列: MVLQELWFGV IAALFLGFFI LEGFDFGVGM LMAPFAHVGM GDPETHRRTA LNTIGPVWDG NEVWLITAGA AIFAAFPGWY ATVFSALYL PLLAILFGMI LRAVAIEWRG KIDDPKWRTG ADFGIAAGSW LPALLWGVAF AILVRGLPVD ANGHVALSIP D VLNAYTLL ...文字列: MVLQELWFGV IAALFLGFFI LEGFDFGVGM LMAPFAHVGM GDPETHRRTA LNTIGPVWDG NEVWLITAGA AIFAAFPGWY ATVFSALYL PLLAILFGMI LRAVAIEWRG KIDDPKWRTG ADFGIAAGSW LPALLWGVAF AILVRGLPVD ANGHVALSIP D VLNAYTLL GGLATAGLFS LYGAVFIALK TSGPIRDDAY RFAVWLSLPV AGLVAGFGLW TQLAYGKDWT WLVLAVAGCA QA AATVLVW RRVSDGWAFM CTLIVVAAVV VLLFGALYPN LVPSTLNPQW SLTIHNASST PYTLKIMTWV TAFFAPLTVA YQT WTYWVF RQRISAERIP PPTGLARRAP UniProtKB: Probable integral membrane cytochrome D ubiquinol oxidase (Subunit II) CydB (Cytochrome BD-I oxidase subunit II) |
-分子 #3: PHOSPHATIDYLETHANOLAMINE
| 分子 | 名称: PHOSPHATIDYLETHANOLAMINE / タイプ: ligand / ID: 3 / コピー数: 1 / 式: PTY |
|---|---|
| 分子量 | 理論値: 734.039 Da |
| Chemical component information |  ChemComp-PTY: |
-分子 #4: MENAQUINONE-9
| 分子 | 名称: MENAQUINONE-9 / タイプ: ligand / ID: 4 / コピー数: 1 / 式: MQ9 |
|---|---|
| 分子量 | 理論値: 785.233 Da |
| Chemical component information | 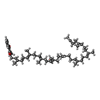 ChemComp-MQ9: |
-分子 #5: PROTOPORPHYRIN IX CONTAINING FE
| 分子 | 名称: PROTOPORPHYRIN IX CONTAINING FE / タイプ: ligand / ID: 5 / コピー数: 2 / 式: HEM |
|---|---|
| 分子量 | 理論値: 616.487 Da |
| Chemical component information |  ChemComp-HEM: |
-分子 #6: CIS-HEME D HYDROXYCHLORIN GAMMA-SPIROLACTONE
| 分子 | 名称: CIS-HEME D HYDROXYCHLORIN GAMMA-SPIROLACTONE / タイプ: ligand / ID: 6 / コピー数: 1 / 式: HDD |
|---|---|
| 分子量 | 理論値: 632.487 Da |
| Chemical component information |  ChemComp-HDD: |
-分子 #7: CARDIOLIPIN
| 分子 | 名称: CARDIOLIPIN / タイプ: ligand / ID: 7 / コピー数: 1 / 式: CDL |
|---|---|
| 分子量 | 理論値: 1.464043 KDa |
| Chemical component information |  ChemComp-CDL: |
-分子 #8: (2R,5R,11R,14R)-5,8,11-trihydroxy-5,11-dioxido-17-oxo-2,14-bis(te...
| 分子 | 名称: (2R,5R,11R,14R)-5,8,11-trihydroxy-5,11-dioxido-17-oxo-2,14-bis(tetradecanoyloxy)-4,6,10,12,16-pentaoxa-5,11-diphosphatriacont-1-yl tetradecanoate タイプ: ligand / ID: 8 / コピー数: 1 / 式: CD4 |
|---|---|
| 分子量 | 理論値: 1.241633 KDa |
| Chemical component information |  ChemComp-CD4: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.5 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
詳細: 50 mM Tris-HCl (pH 7.4), 150 mM NaCl, 0.005% LMNG | ||||||||||||
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 90 sec. / 前処理 - 雰囲気: OTHER / 詳細: 15 mAmp | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV / 詳細: Objective aperture of 100 um |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 2 / 実像数: 14078 / 平均露光時間: 4.04 sec. / 平均電子線量: 100.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)














































