[English] 日本語
 Yorodumi
Yorodumi- EMDB-50176: Poliovirus type 1 (strain Mahoney) stabilised virus-like particle... -
+ Open data
Open data
- Basic information
Basic information
| Entry |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Poliovirus type 1 (strain Mahoney) stabilised virus-like particle (PV1 SC6b) in complex with GPP3 and GSH. | |||||||||
 Map data Map data | ||||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | Capsid protein / virus-like particle / inhibitor / complex / vaccine / VIRUS LIKE PARTICLE | |||||||||
| Function / homology |  Function and homology information Function and homology informationsymbiont-mediated suppression of host translation initiation / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of RIG-I activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MDA-5 activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MAVS activity / picornain 2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane ...symbiont-mediated suppression of host translation initiation / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of RIG-I activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MDA-5 activity / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MAVS activity / picornain 2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / ribonucleoside triphosphate phosphatase activity / nucleoside-triphosphate phosphatase / channel activity / monoatomic ion transmembrane transport / RNA helicase activity / endocytosis involved in viral entry into host cell / symbiont-mediated activation of host autophagy / RNA-directed RNA polymerase / cysteine-type endopeptidase activity / viral RNA genome replication / RNA-directed RNA polymerase activity / DNA-templated transcription / virion attachment to host cell / host cell nucleus / structural molecule activity / proteolysis / RNA binding / zinc ion binding / ATP binding / membrane Similarity search - Function | |||||||||
| Biological species |   Human poliovirus 1 Mahoney Human poliovirus 1 Mahoney | |||||||||
| Method | single particle reconstruction / cryo EM / Resolution: 2.75 Å | |||||||||
 Authors Authors | Bahar MW / Nasta V / Sherry L / Stonehouse NJ / Rowlands DJ / Fry EE / Stuart DI | |||||||||
| Funding support |  United States, 1 items United States, 1 items
| |||||||||
 Citation Citation |  Journal: Nat Commun / Year: 2025 Journal: Nat Commun / Year: 2025Title: Recombinant expression systems for production of stabilised virus-like particles as next-generation polio vaccines. Authors: Lee Sherry / Mohammad W Bahar / Claudine Porta / Helen Fox / Keith Grehan / Veronica Nasta / Helen M E Duyvesteyn / Luigi De Colibus / Johanna Marsian / Inga Murdoch / Daniel Ponndorf / ...Authors: Lee Sherry / Mohammad W Bahar / Claudine Porta / Helen Fox / Keith Grehan / Veronica Nasta / Helen M E Duyvesteyn / Luigi De Colibus / Johanna Marsian / Inga Murdoch / Daniel Ponndorf / Seong-Ryong Kim / Sachin Shah / Sarah Carlyle / Jessica J Swanson / Sue Matthews / Clare Nicol / George P Lomonossoff / Andrew J Macadam / Elizabeth E Fry / David I Stuart / Nicola J Stonehouse / David J Rowlands /   Abstract: Polioviruses have caused crippling disease in humans for centuries, prior to the successful development of vaccines in the mid-1900's, which dramatically reduced disease prevalence. Continued use of ...Polioviruses have caused crippling disease in humans for centuries, prior to the successful development of vaccines in the mid-1900's, which dramatically reduced disease prevalence. Continued use of these vaccines, however, threatens ultimate disease eradication and achievement of a polio-free world. Virus-like particles (VLPs) that lack a viral genome represent a safer potential vaccine, although they require particle stabilization. Using our previously established genetic techniques to stabilize the structural capsid proteins, we demonstrate production of poliovirus VLPs of all three serotypes, from four different recombinant expression systems. We compare the antigenicity, thermostability and immunogenicity of these stabilized VLPs against the current inactivated polio vaccine, demonstrating equivalent or superior immunogenicity in female Wistar rats. Structural analyses of these recombinant VLPs provide a rational understanding of the stabilizing mutations and the role of potential excipients. Collectively, we have established these poliovirus stabilized VLPs as viable next-generation vaccine candidates for the future. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Supplemental images |
|---|
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_50176.map.gz emd_50176.map.gz | 323.8 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-50176-v30.xml emd-50176-v30.xml emd-50176.xml emd-50176.xml | 25.3 KB 25.3 KB | Display Display |  EMDB header EMDB header |
| FSC (resolution estimation) |  emd_50176_fsc.xml emd_50176_fsc.xml | 14.7 KB | Display |  FSC data file FSC data file |
| Images |  emd_50176.png emd_50176.png | 210.7 KB | ||
| Filedesc metadata |  emd-50176.cif.gz emd-50176.cif.gz | 8.2 KB | ||
| Others |  emd_50176_half_map_1.map.gz emd_50176_half_map_1.map.gz emd_50176_half_map_2.map.gz emd_50176_half_map_2.map.gz | 314.5 MB 314.5 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-50176 http://ftp.pdbj.org/pub/emdb/structures/EMD-50176 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50176 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-50176 | HTTPS FTP |
-Related structure data
| Related structure data |  9f3qMC  9eyyC  9ez0C  9f0kC  9f59C  9f5pC M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| Related items in Molecule of the Month |
- Map
Map
| File |  Download / File: emd_50176.map.gz / Format: CCP4 / Size: 343 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_50176.map.gz / Format: CCP4 / Size: 343 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 1.08 Å | ||||||||||||||||||||||||||||||||||||
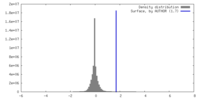
| Density |
| ||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
|
-Supplemental data
-Half map: #2
| File | emd_50176_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
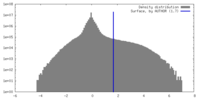
| Density Histograms |
-Half map: #1
| File | emd_50176_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
- Sample components
Sample components
-Entire : Human poliovirus 1 Mahoney
| Entire | Name:   Human poliovirus 1 Mahoney Human poliovirus 1 Mahoney |
|---|---|
| Components |
|
-Supramolecule #1: Human poliovirus 1 Mahoney
| Supramolecule | Name: Human poliovirus 1 Mahoney / type: virus / ID: 1 / Parent: 0 / Macromolecule list: #1-#3 Details: Recombinantly expressed virus-like particle of poliovirus type 1 (Mahoney strain). NCBI-ID: 12081 / Sci species name: Human poliovirus 1 Mahoney / Sci species strain: Mahoney / Virus type: VIRUS-LIKE PARTICLE / Virus isolate: SEROTYPE / Virus enveloped: No / Virus empty: Yes |
|---|---|
| Host (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Molecular weight | Theoretical: 5.84 MDa |
| Virus shell | Shell ID: 1 / Name: Virus shell 1 / Diameter: 310.0 Å / T number (triangulation number): 1 |
-Macromolecule #1: Capsid protein VP1
| Macromolecule | Name: Capsid protein VP1 / type: protein_or_peptide / ID: 1 / Number of copies: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:   Human poliovirus 1 Mahoney Human poliovirus 1 Mahoney |
| Molecular weight | Theoretical: 33.447586 KDa |
| Recombinant expression | Organism:  Komagataella pastoris (fungus) Komagataella pastoris (fungus) |
| Sequence | String: GLGQMLESMI DNTVRETVGA ATSRDALPNT EASGPTHSKE IPALTAVETG ATNPLVPSDT VQTRHVVQHR SRSESSIESF FARGACVTI MTVDNPASTT NKDKLFAVWK ITYKDTVQLR RKLEFFTYSR FDMELTFVVT ANFTETNNGH ALNQVYQIMY V PPGAPVPE ...String: GLGQMLESMI DNTVRETVGA ATSRDALPNT EASGPTHSKE IPALTAVETG ATNPLVPSDT VQTRHVVQHR SRSESSIESF FARGACVTI MTVDNPASTT NKDKLFAVWK ITYKDTVQLR RKLEFFTYSR FDMELTFVVT ANFTETNNGH ALNQVYQIMY V PPGAPVPE KWDDYTWQTS SNPSIFYTYG TAPARISVPY VGISNAYSHF YDGFSKVPLK DQSAALGDSL YGAASLNDFG IL AVRVVND PNPTKVTSKI RVYLKPKHIR VWCPRPPRAV AYYGPGVDYK DGTLTPLSTK DLTTY UniProtKB: Genome polyprotein |
-Macromolecule #2: Capsid protein VP0
| Macromolecule | Name: Capsid protein VP0 / type: protein_or_peptide / ID: 2 / Number of copies: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:   Human poliovirus 1 Mahoney Human poliovirus 1 Mahoney |
| Molecular weight | Theoretical: 37.465812 KDa |
| Recombinant expression | Organism:  Komagataella pastoris (fungus) Komagataella pastoris (fungus) |
| Sequence | String: MGAQVSSQKV GAHENSNGAY GGSTINYTTI NYYRDSASNA ASKQDFSQDP SKFTEPIKDV LIKTAPMLNS PNIEACGYSD RVLQLTLGN STITAQEAAN SVVAYGRWPE YLRDSEANPV DQPTEPEVAA CRFYTLDTVS WTKESRGWWW KLPDALRDMG L FGQNMYYH ...String: MGAQVSSQKV GAHENSNGAY GGSTINYTTI NYYRDSASNA ASKQDFSQDP SKFTEPIKDV LIKTAPMLNS PNIEACGYSD RVLQLTLGN STITAQEAAN SVVAYGRWPE YLRDSEANPV DQPTEPEVAA CRFYTLDTVS WTKESRGWWW KLPDALRDMG L FGQNMYYH YLGRSGYTVH VQCNASKFHQ GALGVFAVPE MCLAGDSNTT TMHTSYQNAN PGEKGGTFTG TFTPDNNQTS PA RRFCPVD YLLGNGTLLG NAFVFPHQII NLRTNNCATL VLPYVNSLSI DSMVKHNNWG IAILPLAPLN FASESSPEIP ITL TIAPMC CEFNGLRNIT LPRLQ UniProtKB: Genome polyprotein |
-Macromolecule #3: Capsid protein VP3
| Macromolecule | Name: Capsid protein VP3 / type: protein_or_peptide / ID: 3 / Number of copies: 1 / Enantiomer: LEVO |
|---|---|
| Source (natural) | Organism:   Human poliovirus 1 Mahoney Human poliovirus 1 Mahoney |
| Molecular weight | Theoretical: 26.550549 KDa |
| Recombinant expression | Organism:  Komagataella pastoris (fungus) Komagataella pastoris (fungus) |
| Sequence | String: GLPVMNTPGS NQYLTADNFQ SPCALPEFDV TPPIDIPGEV KNMMELAEID TMIPFDLSAT KKNTMEMYRV RLSDKPHTDD PILCLSLSP ASDPRLSHTM LGEILNYYTH WAGSLKFTFM FCGSMMATGK LLVSYAPPGA DPPKKRKEAM LGTHVIWDIG L QSSCTMVV ...String: GLPVMNTPGS NQYLTADNFQ SPCALPEFDV TPPIDIPGEV KNMMELAEID TMIPFDLSAT KKNTMEMYRV RLSDKPHTDD PILCLSLSP ASDPRLSHTM LGEILNYYTH WAGSLKFTFM FCGSMMATGK LLVSYAPPGA DPPKKRKEAM LGTHVIWDIG L QSSCTMVV PWISNTTYRL TIDDSFTEGG YISVFYQTRI VVPLSTPREM DILGFVSACN DFSVRLLRDT THIEQKALAQ UniProtKB: Genome polyprotein |
-Macromolecule #4: 1-[(3S)-5-[4-[(E)-ETHOXYIMINOMETHYL]PHENOXY]-3-METHYL-PENTYL]-3-P...
| Macromolecule | Name: 1-[(3S)-5-[4-[(E)-ETHOXYIMINOMETHYL]PHENOXY]-3-METHYL-PENTYL]-3-PYRIDIN-4-YL-IMIDAZOLIDIN-2-ONE type: ligand / ID: 4 / Number of copies: 1 / Formula: YM2 |
|---|---|
| Molecular weight | Theoretical: 410.509 Da |
| Chemical component information | 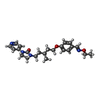 ChemComp-YM2: |
-Macromolecule #5: GLUTATHIONE
| Macromolecule | Name: GLUTATHIONE / type: ligand / ID: 5 / Number of copies: 1 / Formula: GSH |
|---|---|
| Molecular weight | Theoretical: 307.323 Da |
| Chemical component information |  ChemComp-GSH: |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Concentration | 0.1 mg/mL | ||||||
|---|---|---|---|---|---|---|---|
| Buffer | pH: 7 / Component:
| ||||||
| Grid | Model: EMS Lacey Carbon / Mesh: 400 / Support film - Material: CARBON / Support film - topology: LACEY / Support film - Film thickness: 3 / Pretreatment - Type: GLOW DISCHARGE / Pretreatment - Time: 30 sec. / Pretreatment - Atmosphere: AIR Details: The exact grid type used was Ultra-thin carbon support film, 3nm - on lacey carbon (product AGS187-4, Agar Scientific). | ||||||
| Vitrification | Cryogen name: ETHANE-PROPANE / Chamber humidity: 100 % / Chamber temperature: 277.15 K / Instrument: FEI VITROBOT MARK IV Details: 3-4 ul of sample blotted for 3.5 seconds with -15 blot force on FEI Vitrobot mark IV.. | ||||||
| Details | Sample purified by sucrose density gradient ultracentrifugation. Concentrated sample mixed with GPP3 and GSH (10mM final) to form complex. |
- Electron microscopy
Electron microscopy
| Microscope | FEI TITAN KRIOS |
|---|---|
| Details | Calibrated pixel size was 1.08 A/pix. |
| Image recording | Film or detector model: FEI FALCON III (4k x 4k) / Detector mode: INTEGRATING / Digitization - Frames/image: 1-30 / Number grids imaged: 1 / Number real images: 4467 / Average exposure time: 0.77 sec. / Average electron dose: 35.49 e/Å2 Details: Pixel sampling was 1.08 A/pixel. Images collected in linear mode on Falcon 3. |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | C2 aperture diameter: 100.0 µm / Calibrated magnification: 129629 / Illumination mode: FLOOD BEAM / Imaging mode: DARK FIELD / Cs: 2.7 mm / Nominal defocus max: 2.9 µm / Nominal defocus min: 0.8 µm / Nominal magnification: 75000 |
| Sample stage | Specimen holder model: FEI TITAN KRIOS AUTOGRID HOLDER / Cooling holder cryogen: NITROGEN |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
+ Image processing
Image processing
-Atomic model buiding 1
| Initial model | PDB ID: Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| Software | Name:  UCSF Chimera (ver. 1.16) UCSF Chimera (ver. 1.16) |
| Details | Initial model was rigid body fitted using UCSF chimera and Coot. Global minimization and B-factor refinement was performed in real space using phenix_real.space.refine. |
| Refinement | Space: REAL / Protocol: RIGID BODY FIT / Target criteria: Cross-correlation coefficient |
| Output model |  PDB-9f3q: |
 Movie
Movie Controller
Controller










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)