+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4901 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of St1Cas9-sgRNA-tDNA20-AcrIIA6 dimeric assembly. | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CRISPR-Cas9 / anti-CRISPR protein / bacteriophages / Streptococcus thermophilus Cas9 / St1Cas9 / hydrolase | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont-mediated suppression of host CRISPR-cas system / maintenance of CRISPR repeat elements / endonuclease activity / defense response to virus / 加水分解酵素; エステル加水分解酵素 / DNA binding / RNA binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Streptococcus phage D1811 (ファージ) / Streptococcus phage D1811 (ファージ) /  Streptococcus thermophilus (strain ATCC BAA-491 / LMD-9) (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) / Streptococcus thermophilus (strain ATCC BAA-491 / LMD-9) (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) /  Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) / Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) /  Streptococcus virus 2972 (ウイルス) Streptococcus virus 2972 (ウイルス) | |||||||||
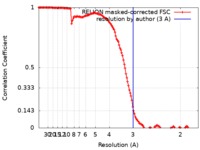
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||
 データ登録者 データ登録者 | Swuec P | |||||||||
| 資金援助 |  フランス, 1件 フランス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2019 ジャーナル: Mol Cell / 年: 2019タイトル: Cas9 Allosteric Inhibition by the Anti-CRISPR Protein AcrIIA6. 著者: Olivier Fuchsbauer / Paolo Swuec / Claire Zimberger / Béatrice Amigues / Sébastien Levesque / Daniel Agudelo / Alexis Duringer / Antonio Chaves-Sanjuan / Silvia Spinelli / Geneviève M ...著者: Olivier Fuchsbauer / Paolo Swuec / Claire Zimberger / Béatrice Amigues / Sébastien Levesque / Daniel Agudelo / Alexis Duringer / Antonio Chaves-Sanjuan / Silvia Spinelli / Geneviève M Rousseau / Minja Velimirovic / Martino Bolognesi / Alain Roussel / Christian Cambillau / Sylvain Moineau / Yannick Doyon / Adeline Goulet /    要旨: In the arms race against bacteria, bacteriophages have evolved diverse anti-CRISPR proteins (Acrs) that block CRISPR-Cas immunity. Acrs play key roles in the molecular coevolution of bacteria with ...In the arms race against bacteria, bacteriophages have evolved diverse anti-CRISPR proteins (Acrs) that block CRISPR-Cas immunity. Acrs play key roles in the molecular coevolution of bacteria with their predators, use a variety of mechanisms of action, and provide tools to regulate Cas-based genome manipulation. Here, we present structural and functional analyses of AcrIIA6, an Acr from virulent phages, exploring its unique anti-CRISPR action. Our cryo-EM structures and functional data of AcrIIA6 binding to Streptococcus thermophilus Cas9 (St1Cas9) show that AcrIIA6 acts as an allosteric inhibitor and induces St1Cas9 dimerization. AcrIIA6 reduces St1Cas9 binding affinity for DNA and prevents DNA binding within cells. The PAM and AcrIIA6 recognition sites are structurally close and allosterically linked. Mechanistically, AcrIIA6 affects the St1Cas9 conformational dynamics associated with PAM binding. Finally, we identify a natural St1Cas9 variant resistant to AcrIIA6 illustrating Acr-driven mutational escape and molecular diversification of Cas9 proteins. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4901.map.gz emd_4901.map.gz | 190 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4901-v30.xml emd-4901-v30.xml emd-4901.xml emd-4901.xml | 19.4 KB 19.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4901_fsc.xml emd_4901_fsc.xml | 13.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4901.png emd_4901.png | 137 KB | ||
| マスクデータ |  emd_4901_msk_1.map emd_4901_msk_1.map | 202.8 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-4901.cif.gz emd-4901.cif.gz | 7.2 KB | ||
| その他 |  emd_4901_additional.map.gz emd_4901_additional.map.gz | 159.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4901 http://ftp.pdbj.org/pub/emdb/structures/EMD-4901 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4901 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4901 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4901_validation.pdf.gz emd_4901_validation.pdf.gz | 665.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4901_full_validation.pdf.gz emd_4901_full_validation.pdf.gz | 665.4 KB | 表示 | |
| XML形式データ |  emd_4901_validation.xml.gz emd_4901_validation.xml.gz | 13.2 KB | 表示 | |
| CIF形式データ |  emd_4901_validation.cif.gz emd_4901_validation.cif.gz | 17.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4901 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4901 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4901 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4901 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4901.map.gz / 形式: CCP4 / 大きさ: 202.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4901.map.gz / 形式: CCP4 / 大きさ: 202.8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.889 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
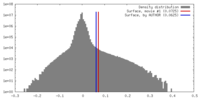
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_4901_msk_1.map emd_4901_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
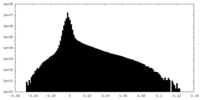
| 密度ヒストグラム |
-追加マップ: #1
| ファイル | emd_4901_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM structure of St1Cas9-sgRNA-tDNA20-AcrIIA6 dimeric assembly.
| 全体 | 名称: Cryo-EM structure of St1Cas9-sgRNA-tDNA20-AcrIIA6 dimeric assembly. |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM structure of St1Cas9-sgRNA-tDNA20-AcrIIA6 dimeric assembly.
| 超分子 | 名称: Cryo-EM structure of St1Cas9-sgRNA-tDNA20-AcrIIA6 dimeric assembly. タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|
-超分子 #2: AcrIIA6
| 超分子 | 名称: AcrIIA6 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Streptococcus phage D1811 (ファージ) Streptococcus phage D1811 (ファージ) |
-超分子 #3: CRISPR-associated endonuclease Cas9
| 超分子 | 名称: CRISPR-associated endonuclease Cas9 / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  Streptococcus thermophilus (strain ATCC BAA-491 / LMD-9) (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus (strain ATCC BAA-491 / LMD-9) (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) |
-超分子 #4: RNA (78-MER)
| 超分子 | 名称: RNA (78-MER) / タイプ: complex / ID: 4 / 親要素: 1 / 含まれる分子: #3 |
|---|---|
| 由来(天然) | 生物種:  Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) |
-超分子 #5: tDNA20
| 超分子 | 名称: tDNA20 / タイプ: complex / ID: 5 / 親要素: 1 / 含まれる分子: #4 |
|---|---|
| 由来(天然) | 生物種:  Streptococcus virus 2972 (ウイルス) Streptococcus virus 2972 (ウイルス) |
-分子 #1: AcrIIA6
| 分子 | 名称: AcrIIA6 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Streptococcus phage D1811 (ファージ) Streptococcus phage D1811 (ファージ) |
| 分子量 | 理論値: 21.464391 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKINDDIKEL ILEYMSRYFK FENDFYKLPG IKFTDANWQK FKNGGTDIEK MGAARVNAML DCLFDDFELA MIGKAQTNYY NDNSLKMNM PFYTYYDMFK KQQLLKWLKN NRDDVIGGTG RMYTASGNYI ANAYLEVALE SSSLGSGSYM LQMRFKDYSK G QEPIPSGR QNRLEWIENN LENIR UniProtKB: AcrIIA6 |
-分子 #2: CRISPR-associated endonuclease Cas9 1
| 分子 | 名称: CRISPR-associated endonuclease Cas9 1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO EC番号: 加水分解酵素; エステル加水分解酵素 |
|---|---|
| 由来(天然) | 生物種:  Streptococcus thermophilus (strain ATCC BAA-491 / LMD-9) (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus (strain ATCC BAA-491 / LMD-9) (ストレプトコッカス・サリバリウス 亜種 サーモフィラス)株: ATCC BAA-491 / LMD-9 |
| 分子量 | 理論値: 129.700961 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSDLVLGLDI GIGSVGVGIL NKVTGEIIHK NSRIFPAAQA ENNLVRRTNR QGRRLTRRKK HRRVRLNRLF EESGLITDFT KISINLNPY QLRVKGLTDE LSNEELFIAL KNMVKHRGIS YLDDASDDGN SSIGDYAQIV KENSKQLETK TPGQIQLERY Q TYGQLRGD ...文字列: MSDLVLGLDI GIGSVGVGIL NKVTGEIIHK NSRIFPAAQA ENNLVRRTNR QGRRLTRRKK HRRVRLNRLF EESGLITDFT KISINLNPY QLRVKGLTDE LSNEELFIAL KNMVKHRGIS YLDDASDDGN SSIGDYAQIV KENSKQLETK TPGQIQLERY Q TYGQLRGD FTVEKDGKKH RLINVFPTSA YRSEALRILQ TQQEFNPQIT DEFINRYLEI LTGKRKYYHG PGNEKSRTDY GR YRTSGET LDNIFGILIG KCTFYPDEFR AAKASYTAQE FNLLNDLNNL TVPTETKKLS KEQKNQIINY VKNEKAMGPA KLF KYIAKL LSCDVADIKG YRIDKSGKAE IHTFEAYRKM KTLETLDIEQ MDRETLDKLA YVLTLNTERE GIQEALEHEF ADGS FSQKQ VDELVQFRKA NSSIFGKGWH NFSVKLMMEL IPELYETSEE QMTILTRLGK QKTTSSSNKT KYIDEKLLTE EIYNP VVAK SVRQAIKIVN AAIKEYGDFD NIVIEMARET NEDDEKKAIQ KIQKANKDEK DAAMLKAANQ YNGKAELPHS VFHGHK QLA TKIRLWHQQG ERCLYTGKTI SIHDLINNSN QFEVDHILPL SITFDDSLAN KVLVYATANQ EKGQRTPYQA LDSMDDA WS FRELKAFVRE SKTLSNKKKE YLLTEEDISK FDVRKKFIER NLVDTRYASR VVLNALQEHF RAHKIDTKVS VVRGQFTS Q LRRHWGIEKT RDTYHHHAVD ALIIAASSQL NLWKKQKNTL VSYSEDQLLD IETGELISDD EYKESVFKAP YQHFVDTLK SKEFEDSILF SYQVDSKFNR KISDATIYAT RQAKVGKDKA DETYVLGKIK DIYTQDGYDA FMKIYKKDKS KFLMYRHDPQ TFEKVIEPI LENYPNKQIN EKGKEVPCNP FLKYKEEHGY IRKYSKKGNG PEIKSLKYYD SKLGNHIDIT PKDSNNKVVL Q SVSPWRAD VYFNKTTGKY EILGLKYADL QFEKGTGTYK ISQEKYNDIK KKEGVDSDSE FKFTLYKNDL LLVKDTETKE QQ LFRFLSR TMPKQKHYVE LKPYDKQKFE GGEALIKVLG NVANSGQCKK GLGKSNISIY KVRTDVLGNQ HIIKNEGDKP KLD F UniProtKB: CRISPR-associated endonuclease Cas9 1 |
-分子 #3: RNA (78-MER)
| 分子 | 名称: RNA (78-MER) / タイプ: rna / ID: 3 / コピー数: 2 |
|---|---|
| 由来(天然) | 生物種:  Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) Streptococcus thermophilus (ストレプトコッカス・サリバリウス 亜種 サーモフィラス) |
| 分子量 | 理論値: 37.438039 KDa |
| 配列 | 文字列: GUUGCGUUGA UAAAAGUAUU GUUUUUGUAC UCUCAAGAUU CAAUAAUCUU GCAGAAGCUA CAAAGAUAAG GCUUCAUGCC GAAAUCAAC ACCCUGUCAU UUUAUGGCAG GGUGUUUU |
-分子 #4: tDNA20
| 分子 | 名称: tDNA20 / タイプ: dna / ID: 4 / コピー数: 2 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:  Streptococcus virus 2972 (ウイルス) Streptococcus virus 2972 (ウイルス) |
| 分子量 | 理論値: 6.100993 KDa |
| 配列 | 文字列: (DA)(DA)(DT)(DA)(DC)(DT)(DT)(DT)(DT)(DA) (DT)(DC)(DA)(DA)(DC)(DG)(DC)(DA)(DA)(DG) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.7 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: OTHER |
|---|---|
| 得られたモデル |  PDB-6rja: |
 ムービー
ムービー コントローラー
コントローラー



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)