+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | RB1 Fab bound to 1D4 anti-idiotype Fab | |||||||||
 マップデータ マップデータ | EMReady enhanced sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Fab anti idiotype RSV / IMMUNE SYSTEM | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.79 Å | |||||||||
 データ登録者 データ登録者 | Warren C | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Vaccines (Basel) / 年: 2025 ジャーナル: Vaccines (Basel) / 年: 2025タイトル: Anti-Idiotypic Antibody as a Booster Vaccine Against Respiratory Syncytial Virus. 著者: Shreya Mukhopadhyay / Ioannis Manolaridis / Christopher Warren / Aimin Tang / Gregory O'Donnell / Bin Luo / Ryan P Staupe / Kalpit A Vora / Zhifeng Chen /  要旨: The respiratory syncytial virus (RSV) is a major cause of lower respiratory tract infections in children and adults. With nearly everyone infected by the age of five, there is an opportunity to ... The respiratory syncytial virus (RSV) is a major cause of lower respiratory tract infections in children and adults. With nearly everyone infected by the age of five, there is an opportunity to develop booster vaccines that enhance B-cell immunity, promoting potent and broadly neutralizing antibodies. One potential approach involves using anti-idiotypic antibodies (anti-IDs) to mimic specific antigenic sites and enhance preexisting immunity in an epitope-specific manner. RB1, a monoclonal antibody (mAb) that binds to site IV of the RSV fusion (RSV F) protein, is a potent and broadly neutralizing against RSV A and B viruses. It is the precursor for MK1654 (clesrovimab), which successfully completed a Phase III clinical trial. In this study, we isolated two anti-IDs, 1A6 and 1D4, targeting RB1 CDR regions, demonstrating that 1A6 competes fully with RSV F in binding to RB1. We resolved the RB1-1A6 and RB1-1D4 Fab-Fab complex structures and proved that 1A6 mimics the RSV F site IV better than 1D4. In an immunogenicity study, mice primed with RSV F and boosted with 1A6 Fab showed a site IV-specific antibody response with a concurrent increase in RSV virus neutralization. These results suggest that anti-IDs could be potentially used as booster vaccines for specific epitopes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
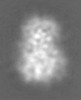
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_48404.map.gz emd_48404.map.gz | 22.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-48404-v30.xml emd-48404-v30.xml emd-48404.xml emd-48404.xml | 20.3 KB 20.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_48404_fsc.xml emd_48404_fsc.xml | 14 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_48404.png emd_48404.png | 63.9 KB | ||
| Filedesc metadata |  emd-48404.cif.gz emd-48404.cif.gz | 6 KB | ||
| その他 |  emd_48404_additional_1.map.gz emd_48404_additional_1.map.gz emd_48404_additional_2.map.gz emd_48404_additional_2.map.gz emd_48404_half_map_1.map.gz emd_48404_half_map_1.map.gz emd_48404_half_map_2.map.gz emd_48404_half_map_2.map.gz | 24.1 MB 24 MB 24.1 MB 24.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-48404 http://ftp.pdbj.org/pub/emdb/structures/EMD-48404 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-48404 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-48404 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_48404_validation.pdf.gz emd_48404_validation.pdf.gz | 558.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_48404_full_validation.pdf.gz emd_48404_full_validation.pdf.gz | 558.3 KB | 表示 | |
| XML形式データ |  emd_48404_validation.xml.gz emd_48404_validation.xml.gz | 4.2 KB | 表示 | |
| CIF形式データ |  emd_48404_validation.cif.gz emd_48404_validation.cif.gz | 4.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-48404 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-48404 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-48404 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-48404 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_48404.map.gz / 形式: CCP4 / 大きさ: 25.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_48404.map.gz / 形式: CCP4 / 大きさ: 25.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | EMReady enhanced sharpened map | ||||||||||||||||||||||||||||||||||||
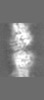
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.5 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: sharpened map
| ファイル | emd_48404_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map | ||||||||||||
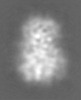
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: unsharpened map
| ファイル | emd_48404_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: halfB
| ファイル | emd_48404_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | halfB | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: halfA
| ファイル | emd_48404_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | halfA | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : RB1 Fab bound to 1D4 anti-idiotype Fab
| 全体 | 名称: RB1 Fab bound to 1D4 anti-idiotype Fab |
|---|---|
| 要素 |
|
-超分子 #1: RB1 Fab bound to 1D4 anti-idiotype Fab
| 超分子 | 名称: RB1 Fab bound to 1D4 anti-idiotype Fab / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: 1D4 heavy chain
| 分子 | 名称: 1D4 heavy chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.029652 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QVQLVESGGG LVQPGGSLRL SCAASGFTFS SSGLHWVRQA PGKGLEWVSG IDSSSSTTYY ADSVKGRFTI SRDNSKNTLY LQMNSLRAE DTAVYYCARY YFDVWGQGTL VTVSSASTKG PSVFPLAPSS KSTSGGTAAL GCLVKDYFPE PVTVSWNSGA L TSGVHTFP ...文字列: QVQLVESGGG LVQPGGSLRL SCAASGFTFS SSGLHWVRQA PGKGLEWVSG IDSSSSTTYY ADSVKGRFTI SRDNSKNTLY LQMNSLRAE DTAVYYCARY YFDVWGQGTL VTVSSASTKG PSVFPLAPSS KSTSGGTAAL GCLVKDYFPE PVTVSWNSGA L TSGVHTFP AVLQSSGLYS LSSVVTVPSS SLGTQTYICN VNHKPSNTKV DKKVEPKSCD |
-分子 #2: 1D4 light chain
| 分子 | 名称: 1D4 light chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 22.444598 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: DIELTQPPSV SVAPGQTARI SCSGDSIGTY AYWYQQKPGQ APVLVIYADD NRPSGIPERF SGSNSGNTAT LTISGTQAED EADYYCSSW ATDNAPVFGG GTKLTVLGQP KAAPSVTLFP PSSEELQANK ATLVCLISDF YPGAVTVAWK ADSSPVKAGV E TTTPSKQS ...文字列: DIELTQPPSV SVAPGQTARI SCSGDSIGTY AYWYQQKPGQ APVLVIYADD NRPSGIPERF SGSNSGNTAT LTISGTQAED EADYYCSSW ATDNAPVFGG GTKLTVLGQP KAAPSVTLFP PSSEELQANK ATLVCLISDF YPGAVTVAWK ADSSPVKAGV E TTTPSKQS NNKYAASSYL SLTPEQWKSH RSYSCQVTHE GSTVEKTVAP TECS |
-分子 #3: RB1 heavy chain
| 分子 | 名称: RB1 heavy chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 24.551439 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EVQLVESGGG LVRPGRSLRL SCTVSGFSFD DSAMSWVRQA PGKGLEWISF IKSKTYGGTK EYAASVKGRF TISRDDSKNI AYLQMNSLK TEDTAVYYCT RGAPYGGNSD YYYGLDVWGQ GTTVTVSSAS TKGPSVFPLA PSSKSTSGGT AALGCLVKDY F PEPVTVSW ...文字列: EVQLVESGGG LVRPGRSLRL SCTVSGFSFD DSAMSWVRQA PGKGLEWISF IKSKTYGGTK EYAASVKGRF TISRDDSKNI AYLQMNSLK TEDTAVYYCT RGAPYGGNSD YYYGLDVWGQ GTTVTVSSAS TKGPSVFPLA PSSKSTSGGT AALGCLVKDY F PEPVTVSW NSGALTSGVH TFPAVLQSSG LYSLSSVVTV PSSSLGTQTY ICNVNHKPSN TKVDKKVEPK SCD |
-分子 #4: RB1 light chain
| 分子 | 名称: RB1 light chain / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.406043 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: DIQMTQSPSS LSASVGDRVT ITCRTSQDVR GALAWYQQKP GKAPKLLIFD ASSLETGVPS RFSGSGSGTV FTLTISSLQP EDFAAYYCQ QFLDFPFTFG QGTRLEIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS ...文字列: DIQMTQSPSS LSASVGDRVT ITCRTSQDVR GALAWYQQKP GKAPKLLIFD ASSLETGVPS RFSGSGSGTV FTLTISSLQP EDFAAYYCQ QFLDFPFTFG QGTRLEIKRT VAAPSVFIFP PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS Q ESVTEQDS KDSTYSLSST LTLSKADYEK HKVYACEVTH QGLSSPVTKS FNRGEC |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 2.3000000000000003 µm 最小 デフォーカス(公称値): 0.6 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)