+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4508 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo em structure of the Listeria stressosome | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Stressosome complex / stress response machine / Bacteria stress sensor / ANTIMICROBIAL PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Listeria monocytogenes EGD-e (バクテリア) Listeria monocytogenes EGD-e (バクテリア) | |||||||||
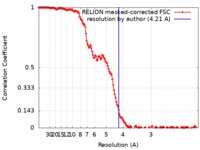
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.21 Å | |||||||||
 データ登録者 データ登録者 | Williams AH / Redzej A | |||||||||
| 資金援助 |  英国, 英国,  フランス, 2件 フランス, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2019 ジャーナル: Nat Commun / 年: 2019タイトル: The cryo-electron microscopy supramolecular structure of the bacterial stressosome unveils its mechanism of activation. 著者: Allison H Williams / Adam Redzej / Nathalie Rolhion / Tiago R D Costa / Aline Rifflet / Gabriel Waksman / Pascale Cossart /   要旨: How the stressosome, the epicenter of the stress response in bacteria, transmits stress signals from the environment has remained elusive. The stressosome consists of multiple copies of three ...How the stressosome, the epicenter of the stress response in bacteria, transmits stress signals from the environment has remained elusive. The stressosome consists of multiple copies of three proteins RsbR, RsbS and RsbT, a kinase that is important for its activation. Using cryo-electron microscopy, we determined the atomic organization of the Listeria monocytogenes stressosome at 3.38 Å resolution. RsbR and RsbS are organized in a 60-protomers truncated icosahedron. A key phosphorylation site on RsbR (T209) is partially hidden by an RsbR flexible loop, whose "open" or "closed" position could modulate stressosome activity. Interaction between three glutamic acids in the N-terminal domain of RsbR and the membrane-bound mini-protein Prli42 is essential for Listeria survival to stress. Together, our data provide the atomic model of the stressosome core and highlight a loop important for stressosome activation, paving the way towards elucidating the mechanism of signal transduction by the stressosome in bacteria. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4508.map.gz emd_4508.map.gz | 21.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4508-v30.xml emd-4508-v30.xml emd-4508.xml emd-4508.xml | 17.8 KB 17.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4508_fsc.xml emd_4508_fsc.xml | 14.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4508.png emd_4508.png | 83.1 KB | ||
| Filedesc metadata |  emd-4508.cif.gz emd-4508.cif.gz | 5.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4508 http://ftp.pdbj.org/pub/emdb/structures/EMD-4508 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4508 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4508 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4508_validation.pdf.gz emd_4508_validation.pdf.gz | 411.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4508_full_validation.pdf.gz emd_4508_full_validation.pdf.gz | 411.1 KB | 表示 | |
| XML形式データ |  emd_4508_validation.xml.gz emd_4508_validation.xml.gz | 13.7 KB | 表示 | |
| CIF形式データ |  emd_4508_validation.cif.gz emd_4508_validation.cif.gz | 18.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4508 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4508 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4508 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4508 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6qcmMC  4510C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4508.map.gz / 形式: CCP4 / 大きさ: 262.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4508.map.gz / 形式: CCP4 / 大きさ: 262.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
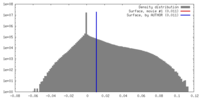
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Listeria Stressosome Core
| 全体 | 名称: Listeria Stressosome Core |
|---|---|
| 要素 |
|
-超分子 #1: Listeria Stressosome Core
| 超分子 | 名称: Listeria Stressosome Core / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: The subcomponents are, RsbR and RsbS |
|---|---|
| 分子量 | 理論値: 1.8 MDa |
-超分子 #2: RsbR
| 超分子 | 名称: RsbR / タイプ: organelle_or_cellular_component / ID: 2 / 親要素: 1 / 含まれる分子: #1-#2, #4-#5 |
|---|---|
| 由来(天然) | 生物種:  Listeria monocytogenes EGD-e (バクテリア) Listeria monocytogenes EGD-e (バクテリア) |
-超分子 #3: RsbS
| 超分子 | 名称: RsbS / タイプ: organelle_or_cellular_component / ID: 3 / 親要素: 1 / 含まれる分子: #3 |
|---|---|
| 由来(天然) | 生物種:  Listeria monocytogenes EGD-e (バクテリア) Listeria monocytogenes EGD-e (バクテリア) |
-分子 #1: RsbR protein
| 分子 | 名称: RsbR protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 30 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Listeria monocytogenes EGD-e (バクテリア) Listeria monocytogenes EGD-e (バクテリア) |
| 分子量 | 理論値: 13.9445 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SALQELSAPL LPIFEKISVM PLIGTIDTER AKLIIENLLI GVVKNRSEVV LIDITGVPVV DTMVAHHIIQ ASEAVRLVGC QAMLVGIRP EIAQTIVNLG IELDQIITTN TMKKGMERAL ALTNREIVE UniProtKB: RsbR protein |
-分子 #2: RsbR protein
| 分子 | 名称: RsbR protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Listeria monocytogenes EGD-e (バクテリア) Listeria monocytogenes EGD-e (バクテリア) |
| 分子量 | 理論値: 14.073681 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: KSALQELSAP LLPIFEKISV MPLIGTIDTE RAKLIIENLL IGVVKNRSEV VLIDITGVPV VDTMVAHHII QASEAVRLVG CQAMLVGIR PEIAQTIVNL GIELDQIITT NTMKKGMERA LALTNREIVE UniProtKB: RsbR protein |
-分子 #3: RsbS protein
| 分子 | 名称: RsbS protein / タイプ: protein_or_peptide / ID: 3 / コピー数: 20 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Listeria monocytogenes EGD-e (バクテリア) Listeria monocytogenes EGD-e (バクテリア) |
| 分子量 | 理論値: 12.606707 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGIPILKLGE CLLISIQSEL DDHTAVEFQE DLLAKIHETS ARGVVIDITS IDFIDSFIAK ILGDVVSMSK LMGAKVVVTG IQPAVAITL IELGITFSGV LSAMDLESGL EKLKQELGE UniProtKB: RsbS protein |
-分子 #4: RsbR protein,RsbR protein
| 分子 | 名称: RsbR protein,RsbR protein / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Listeria monocytogenes EGD-e (バクテリア) Listeria monocytogenes EGD-e (バクテリア) |
| 分子量 | 理論値: 13.479988 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EKTVSIQKSA LQELSAPLLP IFEKISVMPL IGTIDTERAK LIIENLLIGV VKNRSEVVLI DITGVPVVDT MVAHHIIQAS EAVRLVGCQ AMLVGIRPEQ IITTNTMKKG MERALALTNR EIVE UniProtKB: RsbR protein, RsbR protein |
-分子 #5: RsbR protein
| 分子 | 名称: RsbR protein / タイプ: protein_or_peptide / ID: 5 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Listeria monocytogenes EGD-e (バクテリア) Listeria monocytogenes EGD-e (バクテリア) |
| 分子量 | 理論値: 14.860573 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: EKTVSIQKSA LQELSAPLLP IFEKISVMPL IGTIDTERAK LIIENLLIGV VKNRSEVVLI DITGVPVVDT MVAHHIIQAS EAVRLVGCQ AMLVGIRPEI AQTIVNLGIE LDQIITTNTM KKGMERALAL TNREIVE UniProtKB: RsbR protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.02 mg/mL |
|---|---|
| 緩衝液 | pH: 8.5 |
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER/PALLADIUM / メッシュ: 300 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均露光時間: 10.0 sec. / 平均電子線量: 2.25 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: OTHER |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: OTHER |
|---|---|
| 得られたモデル |  PDB-6qcm: |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)