+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of wild type human PSS1 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報L-serine-phosphatidylcholine phosphatidyltransferase activity / L-serine-phosphatidylethanolamine phosphatidyltransferase / L-serine-phosphatidylethanolamine phosphatidyltransferase activity / Synthesis of PS / phosphatidylserine biosynthetic process / transferase activity / endoplasmic reticulum membrane / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||
 データ登録者 データ登録者 | Long T / Li X | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2024 ジャーナル: Cell / 年: 2024タイトル: Molecular insights into human phosphatidylserine synthase 1 reveal its inhibition promotes LDL uptake. 著者: Tao Long / Dongyu Li / Goncalo Vale / Yaoyukun Jiang / Philip Schmiege / Zhongyue J Yang / Jeffrey G McDonald / Xiaochun Li /  要旨: In mammalian cells, two phosphatidylserine (PS) synthases drive PS synthesis. Gain-of-function mutations in the Ptdss1 gene lead to heightened PS production, causing Lenz-Majewski syndrome (LMS). ...In mammalian cells, two phosphatidylserine (PS) synthases drive PS synthesis. Gain-of-function mutations in the Ptdss1 gene lead to heightened PS production, causing Lenz-Majewski syndrome (LMS). Recently, pharmacological inhibition of PSS1 has been shown to suppress tumorigenesis. Here, we report the cryo-EM structures of wild-type human PSS1 (PSS1), the LMS-causing Pro269Ser mutant (PSS1), and PSS1 in complex with its inhibitor DS55980254. PSS1 contains 10 transmembrane helices (TMs), with TMs 4-8 forming a catalytic core in the luminal leaflet. These structures revealed a working mechanism of PSS1 akin to the postulated mechanisms of the membrane-bound O-acyltransferase family. Additionally, we showed that both PS and DS55980254 can allosterically inhibit PSS1 and that inhibition by DS55980254 activates the SREBP pathways, thus enhancing the expression of LDL receptors and increasing cellular LDL uptake. This work uncovers a mechanism of mammalian PS synthesis and suggests that selective PSS1 inhibitors have the potential to lower blood cholesterol levels. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_44178.map.gz emd_44178.map.gz | 97 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-44178-v30.xml emd-44178-v30.xml emd-44178.xml emd-44178.xml | 15.3 KB 15.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_44178.png emd_44178.png | 36.9 KB | ||
| Filedesc metadata |  emd-44178.cif.gz emd-44178.cif.gz | 5.9 KB | ||
| その他 |  emd_44178_half_map_1.map.gz emd_44178_half_map_1.map.gz emd_44178_half_map_2.map.gz emd_44178_half_map_2.map.gz | 95.3 MB 95.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-44178 http://ftp.pdbj.org/pub/emdb/structures/EMD-44178 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44178 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44178 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_44178_validation.pdf.gz emd_44178_validation.pdf.gz | 855.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_44178_full_validation.pdf.gz emd_44178_full_validation.pdf.gz | 855 KB | 表示 | |
| XML形式データ |  emd_44178_validation.xml.gz emd_44178_validation.xml.gz | 13.2 KB | 表示 | |
| CIF形式データ |  emd_44178_validation.cif.gz emd_44178_validation.cif.gz | 15.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44178 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44178 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44178 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-44178 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_44178.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_44178.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8343 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_44178_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_44178_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : PSS1
| 全体 | 名称: PSS1 |
|---|---|
| 要素 |
|
-超分子 #1: PSS1
| 超分子 | 名称: PSS1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Phosphatidylserine synthase 1
| 分子 | 名称: Phosphatidylserine synthase 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO EC番号: L-serine-phosphatidylethanolamine phosphatidyltransferase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 48.359551 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MASCVGSRTL SKDDVNYKMH FRMINEQQVE DITIDFFYRP HTITLLSFTI VSLMYFAFTR DDSVPEDNIW RGILSVIFFF LIISVLAFP NGPFTRPHPA LWRMVFGLSV LYFLFLVFLL FLNFEQVKSL MYWLDPNLRY ATREADVMEY AVNCHVITWE R IISHFDIF ...文字列: MASCVGSRTL SKDDVNYKMH FRMINEQQVE DITIDFFYRP HTITLLSFTI VSLMYFAFTR DDSVPEDNIW RGILSVIFFF LIISVLAFP NGPFTRPHPA LWRMVFGLSV LYFLFLVFLL FLNFEQVKSL MYWLDPNLRY ATREADVMEY AVNCHVITWE R IISHFDIF AFGHFWGWAM KALLIRSYGL CWTISITWEL TELFFMHLLP NFAECWWDQV ILDILLCNGG GIWLGMVVCR FL EMRTYHW ASFKDIHTTT GKIKRAVLQF TPASWTYVRW FDPKSSFQRV AGVYLFMIIW QLTELNTFFL KHIFVFQASH PLS WGRILF IGGITAPTVR QYYAYLTDTQ CKRVGTQCWV FGVIGFLEAI VCIKFGQDLF SKTQILYVVL WLLCVAFTTF LCLY GMIWY AEHY UniProtKB: Phosphatidylserine synthase 1 |
-分子 #2: O-[(R)-{[(2R)-2,3-bis(octadecanoyloxy)propyl]oxy}(hydroxy)phospho...
| 分子 | 名称: O-[(R)-{[(2R)-2,3-bis(octadecanoyloxy)propyl]oxy}(hydroxy)phosphoryl]-L-serine タイプ: ligand / ID: 2 / コピー数: 4 / 式: P5S |
|---|---|
| 分子量 | 理論値: 792.075 Da |
| Chemical component information |  ChemComp-P5S: |
-分子 #3: 1-palmitoyl-2-oleoyl-sn-glycero-3-phosphocholine
| 分子 | 名称: 1-palmitoyl-2-oleoyl-sn-glycero-3-phosphocholine / タイプ: ligand / ID: 3 / コピー数: 4 / 式: LBN |
|---|---|
| 分子量 | 理論値: 760.076 Da |
| Chemical component information |  ChemComp-LBN: |
-分子 #4: (2R)-3-{[(S)-hydroxy{[(1R,2R,3R,4R,5R,6S)-2,3,4,5,6-pentahydroxyc...
| 分子 | 名称: (2R)-3-{[(S)-hydroxy{[(1R,2R,3R,4R,5R,6S)-2,3,4,5,6-pentahydroxycyclohexyl]oxy}phosphoryl]oxy}propane-1,2-diyl dihexadecanoate タイプ: ligand / ID: 4 / コピー数: 2 / 式: A1AIS |
|---|---|
| 分子量 | 理論値: 811.032 Da |
-分子 #5: [(2~{R})-1-[2-azanylethoxy(oxidanyl)phosphoryl]oxy-3-hexadecanoyl...
| 分子 | 名称: [(2~{R})-1-[2-azanylethoxy(oxidanyl)phosphoryl]oxy-3-hexadecanoyloxy-propan-2-yl] (~{Z})-octadec-9-enoate タイプ: ligand / ID: 5 / コピー数: 2 / 式: 6OU |
|---|---|
| 分子量 | 理論値: 717.996 Da |
| Chemical component information | 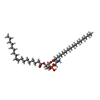 ChemComp-6OU: |
-分子 #6: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 6 / コピー数: 2 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-分子 #7: water
| 分子 | 名称: water / タイプ: ligand / ID: 7 / コピー数: 16 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.7 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 384020 |
| 初期 角度割当 | タイプ: NOT APPLICABLE |
| 最終 角度割当 | タイプ: NOT APPLICABLE |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)