+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4410 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Yeast RPA bound to ssDNA | |||||||||
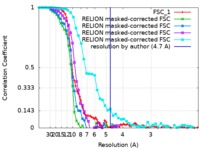
 マップデータ マップデータ | 4.7 Angstrom map (sharpened) corresponding to Yeast RPA Trimerisation Core (Tri-C) bound by ssDNA. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Complex / heterotrimer / DNA binding / OB-fold / DNA BINDING PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報heteroduplex formation / sporulation / DNA replication factor A complex / Gap-filling DNA repair synthesis and ligation in GG-NER / Removal of the Flap Intermediate / telomere maintenance via telomere lengthening / mitotic recombination / Translesion synthesis by REV1 / Translesion synthesis by POLK / Translesion synthesis by POLI ...heteroduplex formation / sporulation / DNA replication factor A complex / Gap-filling DNA repair synthesis and ligation in GG-NER / Removal of the Flap Intermediate / telomere maintenance via telomere lengthening / mitotic recombination / Translesion synthesis by REV1 / Translesion synthesis by POLK / Translesion synthesis by POLI / Translesion Synthesis by POLH / Activation of the pre-replicative complex / single-stranded telomeric DNA binding / Termination of translesion DNA synthesis / Activation of ATR in response to replication stress / telomere maintenance via recombination / reciprocal meiotic recombination / telomeric DNA binding / Gap-filling DNA repair synthesis and ligation in TC-NER / DNA topological change / Dual incision in TC-NER / telomere maintenance via telomerase / telomere maintenance / condensed nuclear chromosome / meiotic cell cycle / nucleotide-excision repair / establishment of protein localization / double-strand break repair via homologous recombination / single-stranded DNA binding / site of double-strand break / double-stranded DNA binding / sequence-specific DNA binding / damaged DNA binding / chromosome, telomeric region / DNA replication / protein ubiquitination / DNA repair / mRNA binding / zinc ion binding / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.7 Å | |||||||||
 データ登録者 データ登録者 | Yates LA / Aramayo RJ | |||||||||
| 資金援助 |  英国, 1件 英国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2018 ジャーナル: Nat Commun / 年: 2018タイトル: A structural and dynamic model for the assembly of Replication Protein A on single-stranded DNA. 著者: Luke A Yates / Ricardo J Aramayo / Nilisha Pokhrel / Colleen C Caldwell / Joshua A Kaplan / Rajika L Perera / Maria Spies / Edwin Antony / Xiaodong Zhang /   要旨: Replication Protein A (RPA), the major eukaryotic single stranded DNA-binding protein, binds to exposed ssDNA to protect it from nucleases, participates in a myriad of nucleic acid transactions and ...Replication Protein A (RPA), the major eukaryotic single stranded DNA-binding protein, binds to exposed ssDNA to protect it from nucleases, participates in a myriad of nucleic acid transactions and coordinates the recruitment of other important players. RPA is a heterotrimer and coats long stretches of single-stranded DNA (ssDNA). The precise molecular architecture of the RPA subunits and its DNA binding domains (DBDs) during assembly is poorly understood. Using cryo electron microscopy we obtained a 3D reconstruction of the RPA trimerisation core bound with ssDNA (∼55 kDa) at ∼4.7 Å resolution and a dimeric RPA assembly on ssDNA. FRET-based solution studies reveal dynamic rearrangements of DBDs during coordinated RPA binding and this activity is regulated by phosphorylation at S178 in RPA70. We present a structural model on how dynamic DBDs promote the cooperative assembly of multiple RPAs on long ssDNA. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4410.map.gz emd_4410.map.gz | 6.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4410-v30.xml emd-4410-v30.xml emd-4410.xml emd-4410.xml | 25.7 KB 25.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4410_fsc_1.xml emd_4410_fsc_1.xml emd_4410_fsc_2.xml emd_4410_fsc_2.xml emd_4410_fsc_3.xml emd_4410_fsc_3.xml emd_4410_fsc_4.xml emd_4410_fsc_4.xml emd_4410_fsc_5.xml emd_4410_fsc_5.xml | 10 KB 3 KB 3 KB 3 KB 4.4 KB | 表示 表示 表示 表示 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4410.png emd_4410.png | 95.6 KB | ||
| Filedesc metadata |  emd-4410.cif.gz emd-4410.cif.gz | 6.8 KB | ||
| その他 |  emd_4410_additional_1.map.gz emd_4410_additional_1.map.gz emd_4410_additional_2.map.gz emd_4410_additional_2.map.gz emd_4410_additional_3.map.gz emd_4410_additional_3.map.gz emd_4410_additional_4.map.gz emd_4410_additional_4.map.gz | 49.4 MB 1.8 MB 1.8 MB 1.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4410 http://ftp.pdbj.org/pub/emdb/structures/EMD-4410 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4410 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4410 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4410_validation.pdf.gz emd_4410_validation.pdf.gz | 489.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4410_full_validation.pdf.gz emd_4410_full_validation.pdf.gz | 489.1 KB | 表示 | |
| XML形式データ |  emd_4410_validation.xml.gz emd_4410_validation.xml.gz | 5.1 KB | 表示 | |
| CIF形式データ |  emd_4410_validation.cif.gz emd_4410_validation.cif.gz | 5.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4410 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4410 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4410 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4410 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4410.map.gz / 形式: CCP4 / 大きさ: 6.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4410.map.gz / 形式: CCP4 / 大きさ: 6.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 4.7 Angstrom map (sharpened) corresponding to Yeast RPA Trimerisation Core (Tri-C) bound by ssDNA. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: 7.5 Angstrom map (unsharpened) corresponding to yeast RPA dimer
| ファイル | emd_4410_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 7.5 Angstrom map (unsharpened) corresponding to yeast RPA dimer | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: 7.7 Angstrom map (sharpened) corresponding to Yeast RPA...
| ファイル | emd_4410_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 7.7 Angstrom map (sharpened) corresponding to Yeast RPA Trimerisation Core (Tri-C) bound by ssDNA with additional DNA-binding domain (DBD-B) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: 8.5 Angstrom map (sharpened) corresponding to Yeast RPA...
| ファイル | emd_4410_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 8.5 Angstrom map (sharpened) corresponding to Yeast RPA Trimerisation Core (Tri-C) bound by ssDNA with additional DNA-binding domain (DBD-B) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: 10 Angstrom map (sharpened) corresponding to Yeast RPA...
| ファイル | emd_4410_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 10 Angstrom map (sharpened) corresponding to Yeast RPA Trimerisation Core (Tri-C) bound by ssDNA with additional DNA-binding domain (DBD-B) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Replication Protein A heterotrimeric complex with ssDNA
| 全体 | 名称: Replication Protein A heterotrimeric complex with ssDNA |
|---|---|
| 要素 |
|
-超分子 #1: Replication Protein A heterotrimeric complex with ssDNA
| 超分子 | 名称: Replication Protein A heterotrimeric complex with ssDNA タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 分子量 | 理論値: 120 KDa |
-超分子 #2: Replication Protein A heterotrimeric complex with ssDNA
| 超分子 | 名称: Replication Protein A heterotrimeric complex with ssDNA タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:  細胞中の位置: nucleus |
-超分子 #3: Replication Protein A heterotrimeric complex with ssDNA
| 超分子 | 名称: Replication Protein A heterotrimeric complex with ssDNA タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #4 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-分子 #1: Replication factor A protein 3
| 分子 | 名称: Replication factor A protein 3 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 13.827723 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MASETPRVDP TEISNVNAPV FRIIAQIKSQ PTESQLILQS PTISSKNGSE VEMITLNNIR VSMNKTFEID SWYEFVCRNN DDGELGFLI LDAVLCKFKE NEDLSLNGVV ALQRLCKKYP EIY UniProtKB: Replication factor A protein 3 |
-分子 #2: Replication factor A protein 2
| 分子 | 名称: Replication factor A protein 2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 14.80987 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: TNTRVNTLTP VTIKQILESK QDIQDGPFVS HNQELHHVCF VGVVRNITDH TANIFLTIED GTGQIEVRKW SEDAAQQFEI GGYVKVFGA LKEFGGKKNI QYAVIKPIDS FNEVLTHHLE VIKCHSIASG MMK UniProtKB: Replication factor A protein 2 |
-分子 #3: Replication factor A protein 1
| 分子 | 名称: Replication factor A protein 1 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 20.643072 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: KFIAQRITIA RAQAENLGRS EKGDFFSVKA AISFLKVDNF AYPACSNENC NKKVLEQPDG TWRCEKCDTN NARPNWRYIL TISIIDETN QLWLTLFDDQ AKQLLGVDAN TLMSLKEEDP NEFTKITQSI QMNEYDFRIR AREDTYNDQS RIRYTVANLH S LNYRAEAD YLADELSKAL UniProtKB: Replication factor A protein 1 |
-分子 #4: DNA (5'-D(P*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP...
| 分子 | 名称: DNA (5'-D(P*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*TP*T)-3') タイプ: dna / ID: 4 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 6.038899 KDa |
| 配列 | 文字列: (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: glow-discharged grid, wait time 30 seconds, blotting time 1 second. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 2.1 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-6i52: |
 ムービー
ムービー コントローラー
コントローラー




















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)