[English] 日本語
 Yorodumi
Yorodumi- EMDB-44068: Cryo-EM structure of human uMtCK1 in complex with transition stat... -
+ Open data
Open data
- Basic information
Basic information
| Entry |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Title | Cryo-EM structure of human uMtCK1 in complex with transition state analog | ||||||||||||
 Map data Map data | |||||||||||||
 Sample Sample |
| ||||||||||||
 Keywords Keywords | mitochondrial creatine kinase / TRANSFERASE | ||||||||||||
| Function / homology |  Function and homology information Function and homology informationcreatine kinase / Creatine metabolism / phosphocreatine biosynthetic process / creatine kinase activity / mitochondrial inner membrane / mitochondrion / ATP binding Similarity search - Function | ||||||||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | ||||||||||||
| Method | single particle reconstruction / cryo EM / Resolution: 2.2 Å | ||||||||||||
 Authors Authors | Demir M / Koepping L / Zhao J / Sergienko E | ||||||||||||
| Funding support |  United States, 3 items United States, 3 items
| ||||||||||||
 Citation Citation |  Journal: Structure / Year: 2025 Journal: Structure / Year: 2025Title: Structural basis for substrate binding, catalysis, and inhibition of cancer target mitochondrial creatine kinase by a covalent inhibitor. Authors: Merve Demir / Laura Koepping / Ya Li / Lynn Fujimoto / Andrey Bobkov / Jianhua Zhao / Taro Hitosugi / Eduard Sergienko /  Abstract: Mitochondrial creatine kinases (MtCKs) are key players in maintaining energy homeostasis in cells that work with cytosolic creatine kinases for energy transport from mitochondria to cytoplasm. The ...Mitochondrial creatine kinases (MtCKs) are key players in maintaining energy homeostasis in cells that work with cytosolic creatine kinases for energy transport from mitochondria to cytoplasm. The inhibition of breast cancer growth by cyclocreatine targeting CKs indicates dependence of cancer cells on the "energy shuttle" for cell growth and survival. Hence, understanding key mechanistic features of creatine kinases and their inhibition plays an important role in the development of cancer therapeutics. Herein, we present mutational and structural investigations on understudied ubiquitous MtCK that showed closure of the loop comprising His61 is specific to and relies on creatine binding and mechanism of phosphoryl transfer depends on electrostatics of active site. We demonstrate that previously identified pan-CK covalent inhibitor CKi inhibit breast cancer cell proliferation; however, our biochemical and structural data indicated that inhibition by CKi is highly dependent on covalent link formation and conformational changes upon creatine binding are not observed. | ||||||||||||
| History |
|
- Structure visualization
Structure visualization
| Supplemental images |
|---|
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_44068.map.gz emd_44068.map.gz | 32.3 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-44068-v30.xml emd-44068-v30.xml emd-44068.xml emd-44068.xml | 19.1 KB 19.1 KB | Display Display |  EMDB header EMDB header |
| Images |  emd_44068.png emd_44068.png | 54.4 KB | ||
| Masks |  emd_44068_msk_1.map emd_44068_msk_1.map | 64 MB |  Mask map Mask map | |
| Filedesc metadata |  emd-44068.cif.gz emd-44068.cif.gz | 6.5 KB | ||
| Others |  emd_44068_half_map_1.map.gz emd_44068_half_map_1.map.gz emd_44068_half_map_2.map.gz emd_44068_half_map_2.map.gz | 59.1 MB 59.1 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-44068 http://ftp.pdbj.org/pub/emdb/structures/EMD-44068 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44068 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44068 | HTTPS FTP |
-Related structure data
| Related structure data |  9b14MC  9b04C  9b05C  9b0tC  9b0uC  9b16C M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| Related items in Molecule of the Month |
- Map
Map
| File |  Download / File: emd_44068.map.gz / Format: CCP4 / Size: 64 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_44068.map.gz / Format: CCP4 / Size: 64 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 1.064 Å | ||||||||||||||||||||||||||||||||||||
| Density |
| ||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
|
-Supplemental data
-Mask #1
| File |  emd_44068_msk_1.map emd_44068_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Half map: #2
| File | emd_44068_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
-Half map: #1
| File | emd_44068_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Projections & Slices |
| ||||||||||||
| Density Histograms |
- Sample components
Sample components
-Entire : Octameric u-type mitochondrial creatine kinase
| Entire | Name: Octameric u-type mitochondrial creatine kinase |
|---|---|
| Components |
|
-Supramolecule #1: Octameric u-type mitochondrial creatine kinase
| Supramolecule | Name: Octameric u-type mitochondrial creatine kinase / type: complex / ID: 1 / Parent: 0 / Macromolecule list: #1 |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
-Macromolecule #1: Creatine kinase U-type, mitochondrial
| Macromolecule | Name: Creatine kinase U-type, mitochondrial / type: protein_or_peptide / ID: 1 / Number of copies: 8 / Enantiomer: LEVO / EC number: creatine kinase |
|---|---|
| Source (natural) | Organism:  Homo sapiens (human) Homo sapiens (human) |
| Molecular weight | Theoretical: 47.598719 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: MGSSHHHHHH SSGLVPRGSH MENLYFQGAA SERRRLYPPS AEYPDLRKHN NCMASHLTPA VYARLCDKTT PTGWTLDQCI QTGVDNPGH PFIKTVGMVA GDEETYEVFA DLFDPVIQER HNGYDPRTMK HTTDLDASKI RSGYFDERYV LSSRVRTGRS I RGLSLPPA ...String: MGSSHHHHHH SSGLVPRGSH MENLYFQGAA SERRRLYPPS AEYPDLRKHN NCMASHLTPA VYARLCDKTT PTGWTLDQCI QTGVDNPGH PFIKTVGMVA GDEETYEVFA DLFDPVIQER HNGYDPRTMK HTTDLDASKI RSGYFDERYV LSSRVRTGRS I RGLSLPPA CTRAERREVE RVVVDALSGL KGDLAGRYYR LSEMTEAEQQ QLIDDHFLFD KPVSPLLTAA GMARDWPDAR GI WHNNEKS FLIWVNEEDH TRVISMEKGG NMKRVFERFC RGLKEVERLI QERGWEFMWN ERLGYILTCP SNLGTGLRAG VHI KLPLLS KDSRFPKILE NLRLQKRGTG GVDTAATGGV FDISNLDRLG KSEVELVQLV IDGVNYLIDC ERRLERGQDI RIPT PVIHT KHGSSDYKDD DDK UniProtKB: Creatine kinase U-type, mitochondrial |
-Macromolecule #2: N-[(E)-AMINO(IMINO)METHYL]-N-METHYLGLYCINE
| Macromolecule | Name: N-[(E)-AMINO(IMINO)METHYL]-N-METHYLGLYCINE / type: ligand / ID: 2 / Number of copies: 8 / Formula: CRN |
|---|---|
| Molecular weight | Theoretical: 131.133 Da |
| Chemical component information | 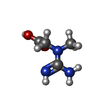 ChemComp-CRN: |
-Macromolecule #3: ADENOSINE-5'-DIPHOSPHATE
| Macromolecule | Name: ADENOSINE-5'-DIPHOSPHATE / type: ligand / ID: 3 / Number of copies: 8 / Formula: ADP |
|---|---|
| Molecular weight | Theoretical: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-Macromolecule #4: MAGNESIUM ION
| Macromolecule | Name: MAGNESIUM ION / type: ligand / ID: 4 / Number of copies: 8 / Formula: MG |
|---|---|
| Molecular weight | Theoretical: 24.305 Da |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Concentration | 1.0 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Buffer | pH: 8 Component:
Details: 10 mM creatine, 1 mM ADP and 50 mM sodium nitrate | ||||||||||||
| Grid | Model: Quantifoil R2/2 / Material: COPPER / Mesh: 300 / Support film - Material: CARBON / Support film - topology: HOLEY / Pretreatment - Type: GLOW DISCHARGE / Pretreatment - Time: 25 sec. | ||||||||||||
| Vitrification | Cryogen name: ETHANE / Chamber humidity: 100 % / Chamber temperature: 277.15 K / Instrument: FEI VITROBOT MARK IV |
- Electron microscopy
Electron microscopy
| Microscope | FEI TITAN KRIOS |
|---|---|
| Image recording | Film or detector model: GATAN K3 (6k x 4k) / Average electron dose: 34.0 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Nominal defocus max: 2.1 µm / Nominal defocus min: 0.9 µm |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
 Movie
Movie Controller
Controller









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)













































