+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of HMPV (MPV-2cREKR) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Prefusion / HMPV / Cryo-EM / VIRUS / VIRAL PROTEIN | |||||||||
| 生物種 |  Human metapneumovirus (ウイルス) Human metapneumovirus (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.14 Å | |||||||||
 データ登録者 データ登録者 | Yu X / Langedijk JPM | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Efficacious human metapneumovirus vaccine based on AI-guided engineering of a closed prefusion trimer. 著者: Mark J G Bakkers / Tina Ritschel / Machteld Tiemessen / Jacobus Dijkman / Angelo A Zuffianò / Xiaodi Yu / Daan van Overveld / Lam Le / Richard Voorzaat / Marlies M van Haaren / Martijn de ...著者: Mark J G Bakkers / Tina Ritschel / Machteld Tiemessen / Jacobus Dijkman / Angelo A Zuffianò / Xiaodi Yu / Daan van Overveld / Lam Le / Richard Voorzaat / Marlies M van Haaren / Martijn de Man / Sem Tamara / Leslie van der Fits / Roland Zahn / Jarek Juraszek / Johannes P M Langedijk /   要旨: The prefusion conformation of human metapneumovirus fusion protein (hMPV Pre-F) is critical for eliciting the most potent neutralizing antibodies and is the preferred immunogen for an efficacious ...The prefusion conformation of human metapneumovirus fusion protein (hMPV Pre-F) is critical for eliciting the most potent neutralizing antibodies and is the preferred immunogen for an efficacious vaccine against hMPV respiratory infections. Here we show that an additional cleavage event in the F protein allows closure and correct folding of the trimer. We therefore engineered the F protein to undergo double cleavage, which enabled screening for Pre-F stabilizing substitutions at the natively folded protomer interfaces. To identify these substitutions, we developed an AI convolutional classifier that successfully predicts complex polar interactions often overlooked by physics-based methods and visual inspection. The combination of additional processing, stabilization of interface regions and stabilization of the membrane-proximal stem, resulted in a Pre-F protein vaccine candidate without the need for a heterologous trimerization domain that exhibited high expression yields and thermostability. Cryo-EM analysis shows the complete ectodomain structure, including the stem, and a specific interaction of the newly identified cleaved C-terminus with the adjacent protomer. Importantly, the protein induces high and cross-neutralizing antibody responses resulting in near complete protection against hMPV challenge in cotton rats, making the highly stable, double-cleaved hMPV Pre-F trimer an attractive vaccine candidate. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_43516.map.gz emd_43516.map.gz | 59.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-43516-v30.xml emd-43516-v30.xml emd-43516.xml emd-43516.xml | 17.2 KB 17.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_43516.png emd_43516.png | 53.5 KB | ||
| Filedesc metadata |  emd-43516.cif.gz emd-43516.cif.gz | 6.4 KB | ||
| その他 |  emd_43516_half_map_1.map.gz emd_43516_half_map_1.map.gz emd_43516_half_map_2.map.gz emd_43516_half_map_2.map.gz | 59.4 MB 59.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-43516 http://ftp.pdbj.org/pub/emdb/structures/EMD-43516 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43516 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43516 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_43516_validation.pdf.gz emd_43516_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_43516_full_validation.pdf.gz emd_43516_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_43516_validation.xml.gz emd_43516_validation.xml.gz | 12.3 KB | 表示 | |
| CIF形式データ |  emd_43516_validation.cif.gz emd_43516_validation.cif.gz | 14.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43516 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43516 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43516 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43516 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_43516.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_43516.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.948 Å | ||||||||||||||||||||||||||||||||||||
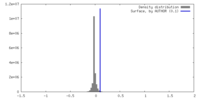
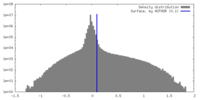
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_43516_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
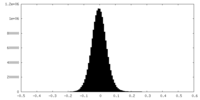
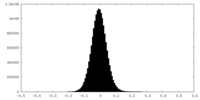
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_43516_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
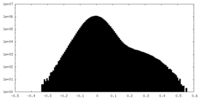
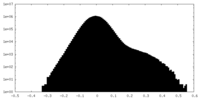
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Prefusion of HMPV (MPV-2cREKR)
| 全体 | 名称: Prefusion of HMPV (MPV-2cREKR) |
|---|---|
| 要素 |
|
-超分子 #1: Prefusion of HMPV (MPV-2cREKR)
| 超分子 | 名称: Prefusion of HMPV (MPV-2cREKR) / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Human metapneumovirus (ウイルス) Human metapneumovirus (ウイルス) |
-分子 #1: Prefusion of HMPV (MPV-2c)
| 分子 | 名称: Prefusion of HMPV (MPV-2c) / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Human metapneumovirus (ウイルス) Human metapneumovirus (ウイルス) |
| 分子量 | 理論値: 56.553586 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MSWKVVIIFS LLITPQHGLK ESYLEESCST ITEGYLSVLR TGWYTNVFTL EVGDVENLTC SDGPSLIKTE LDLTKSALRE LKTVSADQL RRRRELPRFM NYTLNNAKKT NVTLSKKRKR RFVLGAIALG RATAAAVTAG VAIAKTIRLE SEVTAIKNAL K TTNEAVST ...文字列: MSWKVVIIFS LLITPQHGLK ESYLEESCST ITEGYLSVLR TGWYTNVFTL EVGDVENLTC SDGPSLIKTE LDLTKSALRE LKTVSADQL RRRRELPRFM NYTLNNAKKT NVTLSKKRKR RFVLGAIALG RATAAAVTAG VAIAKTIRLE SEVTAIKNAL K TTNEAVST LGNGVRVLAT AVRELKDFVS KNLTRAINKN KCDIDDLKMA VSFSQFNRRF LNVVRQFSEN AGITPAISLD LM TDAELAR AISNMPTSAG QIKLMLENRA MVRRKGFGIL IGVYGSSVIY MVQLPIFGVI DTPCWIVKAA PSCSEKKGNY ACL LREDQG WYCQNAGSTV YYPNEKDCET RGDHVFCDTA AGINVAEQSK ECNINISTTN YPCKVSTGRN PISMVALSPL GALV ACYKG VSCSIGSNRV GIIKQLNKGC SYITNQDADT VTIDNTVYQL SKVEGEQHVI KGRPVSSSFD PIKFPPDQFN VALDQ VFEN IENSQAWVRK FDEILSSIEK GNTGGSEPEA |
-分子 #4: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 4 / コピー数: 3 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)