+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of heparosan synthase 2 from Pasteurella multocida with polysaccharide in the GlcNAc-T active site | |||||||||
 マップデータ マップデータ | Phenix auto-sharpened map, scaled 0.9578358 from original | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | polysaccharide synthase / complex / TRANSFERASE | |||||||||
| 機能・相同性 | Glycosyltransferase 2-like / Glycosyl transferase family 2 / hexosyltransferase activity / biosynthetic process / Nucleotide-diphospho-sugar transferases / nucleotide binding / metal ion binding / Heparosan synthase B 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Pasteurella multocida (バクテリア) Pasteurella multocida (バクテリア) | |||||||||
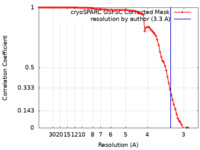
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Krahn JM / Pedersen LC / Liu J / Stancanelli E / Borgnia M / Vivarette E | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: ACS Catal / 年: 2024 ジャーナル: ACS Catal / 年: 2024タイトル: Structural and Functional Analysis of Heparosan Synthase 2 from (PmHS2) to Improve the Synthesis of Heparin. 著者: Eduardo Stancanelli / Juno A Krahn / Elizabeth Viverette / Robert Dutcher / Vijayakanth Pagadala / Mario J Borgnia / Jian Liu / Lars C Pedersen /  要旨: Heparin is a widely used drug to treat thrombotic disorders in hospitals. Heparosan synthase 2 from (PmHS2) is a key enzyme used for the chemoenzymatic synthesis of heparin oligosaccharides. It has ...Heparin is a widely used drug to treat thrombotic disorders in hospitals. Heparosan synthase 2 from (PmHS2) is a key enzyme used for the chemoenzymatic synthesis of heparin oligosaccharides. It has both activities: glucosaminyl transferase activity and glucuronyl transferase activity; however, the mechanism to carry out the glyco-oligomerization is unknown. Here, we report crystal structures of PmHS2 constructs with bound uridine diphosphate (UDP) and a cryo-EM structure of PmHS2 in complex with UDP and a heptasaccharide (NS 7-mer) substrate. Using a LC-MC analytical method, we discovered the enzyme displays both a two-step concerted oligomerization mode and a distributive oligomerization mode depending on the non-reducing end of the starting oligosaccharide primer. Removal of 7 amino acid residues from the C-terminus results in an enzymatically active monomer instead of dimer and loses the concerted oligomerization mode of activity. In addition, the monomer construct can transfer N-acetyl glucosamine at a substrate concentration that is ∼7-fold higher than wildtype enzyme. It was also determined that an F529A mutant can transfer an N-sulfo glucosamine (GlcNS) saccharide from a previously inactive UDP-GlcNS donor. Performing the glyco-transfer reaction at a high substrate concentration and the capability of using unnatural donors are desirable to simplify the chemoenzymatic synthesis to prepare heparin-based therapeutics. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_43269.map.gz emd_43269.map.gz | 60.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-43269-v30.xml emd-43269-v30.xml emd-43269.xml emd-43269.xml | 17.8 KB 17.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_43269_fsc.xml emd_43269_fsc.xml | 8.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_43269.png emd_43269.png | 127 KB | ||
| Filedesc metadata |  emd-43269.cif.gz emd-43269.cif.gz | 6.6 KB | ||
| その他 |  emd_43269_half_map_1.map.gz emd_43269_half_map_1.map.gz emd_43269_half_map_2.map.gz emd_43269_half_map_2.map.gz | 58.9 MB 58.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-43269 http://ftp.pdbj.org/pub/emdb/structures/EMD-43269 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43269 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43269 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_43269_validation.pdf.gz emd_43269_validation.pdf.gz | 956.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_43269_full_validation.pdf.gz emd_43269_full_validation.pdf.gz | 956.2 KB | 表示 | |
| XML形式データ |  emd_43269_validation.xml.gz emd_43269_validation.xml.gz | 16.4 KB | 表示 | |
| CIF形式データ |  emd_43269_validation.cif.gz emd_43269_validation.cif.gz | 21.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43269 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43269 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43269 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-43269 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8viwMC  8vh7C  8vh8C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_43269.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_43269.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Phenix auto-sharpened map, scaled 0.9578358 from original | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.2835 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half map A, scaled 0.9578358 from original
| ファイル | emd_43269_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A, scaled 0.9578358 from original | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map A, scaled 0.9578358 from original
| ファイル | emd_43269_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A, scaled 0.9578358 from original | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : pmHS2 with 7-mer polysaccharide
| 全体 | 名称: pmHS2 with 7-mer polysaccharide |
|---|---|
| 要素 |
|
-超分子 #1: pmHS2 with 7-mer polysaccharide
| 超分子 | 名称: pmHS2 with 7-mer polysaccharide / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Pasteurella multocida (バクテリア) Pasteurella multocida (バクテリア) |
-分子 #1: Heparosan synthase B
| 分子 | 名称: Heparosan synthase B / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pasteurella multocida (バクテリア) Pasteurella multocida (バクテリア) |
| 分子量 | 理論値: 64.548277 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSAAADKQTT SITDLYNEVA KSDLGLVKET NSANPLVSII MTSHNTAQFI EASINSLLLQ TYKNIEIIIV DDDSSDNTFE IASRIANTT SKVRVFRLNS NLGTYFAKNT GILKSKGDII FFQDSDDVCH HERIERCVNI LLANKETIAV RCAYSRLAPE T QHIIKVNN ...文字列: GSAAADKQTT SITDLYNEVA KSDLGLVKET NSANPLVSII MTSHNTAQFI EASINSLLLQ TYKNIEIIIV DDDSSDNTFE IASRIANTT SKVRVFRLNS NLGTYFAKNT GILKSKGDII FFQDSDDVCH HERIERCVNI LLANKETIAV RCAYSRLAPE T QHIIKVNN MDYRLGFITL GMHRKVFQEI GFFNCTTKGS DDEFFHRIAK YYGKEKIKNL LLPLYYNTMR ENSLFTDMVE WI DNHNIIQ KMSDTRQHYA TLFQAMHNET ASHDFKNLFQ FPRIYDALPV PQEMSKLSNP KIPVYINICS IPSRIAQLRR IIG ILKNQC DHFHIYLDGY VEIPDFIKNL GNKATVVHCK DKDNSIRDNG KFILLEELIE KNQDGYYITC DDDIIYPSDY INTM IKKLN EYDDKAVIGL HGILFPSRMT KYFSADRLVY SFYKPLEKDK AVNVLGTGTV SFRVSLFNQF SLSDFTHSGM ADIYF SLLC KKNNILQICI SRPANWLTED NRDSETLYHQ YRDNDEQQTQ LIMENGPWGY SSIYPLVKNH PKFTDLIPCL PFYFL UniProtKB: Heparosan synthase B |
-分子 #3: MANGANESE (II) ION
| 分子 | 名称: MANGANESE (II) ION / タイプ: ligand / ID: 3 / コピー数: 8 / 式: MN |
|---|---|
| 分子量 | 理論値: 54.938 Da |
-分子 #4: URIDINE-5'-DIPHOSPHATE
| 分子 | 名称: URIDINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 8 / 式: UDP |
|---|---|
| 分子量 | 理論値: 404.161 Da |
| Chemical component information |  ChemComp-UDP: |
-分子 #5: water
| 分子 | 名称: water / タイプ: ligand / ID: 5 / コピー数: 4 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | 2D array |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 25 mM Tris pH 7.5, 87.5 mM NaCl, 1 mM MnCl2, 1 mM UDP and 1 mM NS-7mer (GlcA-GlcNS-GlcA-GlcNS-GlcA-GlcNS-GlcA-pNP) |
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: NITROGEN |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 4544 / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)