+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Computational Designed Nanocage O43_129 | |||||||||
 マップデータ マップデータ | Main Map, DeepEMhancer | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | De Novo / Nanocage / O43_129 / extendable nanomaterials / DE NOVO PROTEIN | |||||||||
| 生物種 | unidentified (未定義) / synthetic construct (人工物) | |||||||||
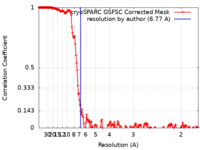
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.77 Å | |||||||||
 データ登録者 データ登録者 | Weidle C / Kibler RD | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2024 ジャーナル: Nature / 年: 2024タイトル: Blueprinting extendable nanomaterials with standardized protein blocks. 著者: Timothy F Huddy / Yang Hsia / Ryan D Kibler / Jinwei Xu / Neville Bethel / Deepesh Nagarajan / Rachel Redler / Philip J Y Leung / Connor Weidle / Alexis Courbet / Erin C Yang / Asim K Bera / ...著者: Timothy F Huddy / Yang Hsia / Ryan D Kibler / Jinwei Xu / Neville Bethel / Deepesh Nagarajan / Rachel Redler / Philip J Y Leung / Connor Weidle / Alexis Courbet / Erin C Yang / Asim K Bera / Nicolas Coudray / S John Calise / Fatima A Davila-Hernandez / Hannah L Han / Kenneth D Carr / Zhe Li / Ryan McHugh / Gabriella Reggiano / Alex Kang / Banumathi Sankaran / Miles S Dickinson / Brian Coventry / T J Brunette / Yulai Liu / Justas Dauparas / Andrew J Borst / Damian Ekiert / Justin M Kollman / Gira Bhabha / David Baker /   要旨: A wooden house frame consists of many different lumber pieces, but because of the regularity of these building blocks, the structure can be designed using straightforward geometrical principles. The ...A wooden house frame consists of many different lumber pieces, but because of the regularity of these building blocks, the structure can be designed using straightforward geometrical principles. The design of multicomponent protein assemblies, in comparison, has been much more complex, largely owing to the irregular shapes of protein structures. Here we describe extendable linear, curved and angled protein building blocks, as well as inter-block interactions, that conform to specified geometric standards; assemblies designed using these blocks inherit their extendability and regular interaction surfaces, enabling them to be expanded or contracted by varying the number of modules, and reinforced with secondary struts. Using X-ray crystallography and electron microscopy, we validate nanomaterial designs ranging from simple polygonal and circular oligomers that can be concentrically nested, up to large polyhedral nanocages and unbounded straight 'train track' assemblies with reconfigurable sizes and geometries that can be readily blueprinted. Because of the complexity of protein structures and sequence-structure relationships, it has not previously been possible to build up large protein assemblies by deliberate placement of protein backbones onto a blank three-dimensional canvas; the simplicity and geometric regularity of our design platform now enables construction of protein nanomaterials according to 'back of an envelope' architectural blueprints. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_42906.map.gz emd_42906.map.gz | 554.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-42906-v30.xml emd-42906-v30.xml emd-42906.xml emd-42906.xml | 23.1 KB 23.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_42906_fsc.xml emd_42906_fsc.xml | 18.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_42906.png emd_42906.png | 188.2 KB | ||
| Filedesc metadata |  emd-42906.cif.gz emd-42906.cif.gz | 6.7 KB | ||
| その他 |  emd_42906_additional_1.map.gz emd_42906_additional_1.map.gz emd_42906_half_map_1.map.gz emd_42906_half_map_1.map.gz emd_42906_half_map_2.map.gz emd_42906_half_map_2.map.gz | 300 MB 575.8 MB 575.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42906 http://ftp.pdbj.org/pub/emdb/structures/EMD-42906 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42906 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42906 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_42906_validation.pdf.gz emd_42906_validation.pdf.gz | 889.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_42906_full_validation.pdf.gz emd_42906_full_validation.pdf.gz | 889.3 KB | 表示 | |
| XML形式データ |  emd_42906_validation.xml.gz emd_42906_validation.xml.gz | 27.6 KB | 表示 | |
| CIF形式データ |  emd_42906_validation.cif.gz emd_42906_validation.cif.gz | 36.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42906 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42906 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42906 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42906 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_42906.map.gz / 形式: CCP4 / 大きさ: 620.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_42906.map.gz / 形式: CCP4 / 大きさ: 620.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Main Map, DeepEMhancer | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.885 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Homogeneous Refinement
| ファイル | emd_42906_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Homogeneous Refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half Map A
| ファイル | emd_42906_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half Map B
| ファイル | emd_42906_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half Map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Nanocage O43_129
| 全体 | 名称: Nanocage O43_129 |
|---|---|
| 要素 |
|
-超分子 #1: Nanocage O43_129
| 超分子 | 名称: Nanocage O43_129 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Chain A and Chain B are expressed separately in E coli. Complex is formed by mixing the cell lysis of both expressions, forming the nanocage. Nanocage was purified through a His tag on chain A. |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) |
| 分子量 | 理論値: 1.60 MDa |
-分子 #1: O43_129 component B
| 分子 | 名称: O43_129 component B / タイプ: protein_or_peptide / ID: 1 / コピー数: 24 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 34.478918 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGDDLLLKLL ELLVEQARVS AEFARRQGDE KMLEEVARKA EEVARKAESI ARKARKEGNL ELALKALEIL VRAAHVLAEI ARERGNEEL QKKAHKLAKE ALRQVIEIAI RAIQEGNLEL AIIALHISVR IAEVLLETRP DDREEIREQQ AIFELLIAAL E AAIRLEKL ...文字列: MGDDLLLKLL ELLVEQARVS AEFARRQGDE KMLEEVARKA EEVARKAESI ARKARKEGNL ELALKALEIL VRAAHVLAEI ARERGNEEL QKKAHKLAKE ALRQVIEIAI RAIQEGNLEL AIIALHISVR IAEVLLETRP DDREEIREQQ AIFELLIAAL E AAIRLEKL KEEGAPPEQI ERVAEHGLER LKEIAKEISK EVDSPESKRI AYKIVAAAAE FLLKILAEGG ATPEQLERVT EH ALEVLKE VAKELADSPE SGLAALAAIA SLAKLGLEQL KEIGAPPEQQ RRVTKAGIEA VREIYRYGRK LY |
-分子 #2: O43_129 component A
| 分子 | 名称: O43_129 component A / タイプ: protein_or_peptide / ID: 2 / コピー数: 24 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 33.976965 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGCDAIQAAA ALGEAGISSN EILELLAAAA ELGLDPDAIQ AAAQLGEAGI SSEEIKELLR AAHELGLDPD AIAAAADLGQ AGVSPVEIL ALLIAASVLG LDPDAIQAAA ALGEAGISAE EIIELLTAAR DLGLDPDAIQ AAAQLGEAGI SSEEIKELLR A AHELGLDP ...文字列: MGCDAIQAAA ALGEAGISSN EILELLAAAA ELGLDPDAIQ AAAQLGEAGI SSEEIKELLR AAHELGLDPD AIAAAADLGQ AGVSPVEIL ALLIAASVLG LDPDAIQAAA ALGEAGISAE EIIELLTAAR DLGLDPDAIQ AAAQLGEAGI SSEEIKELLR A AHELGLDP DCIAAAADLG QAGISSSEIT ALLLAAAAIE LAKRADDKDV REIVRDALEL ASRSTNDEVI RLALEAAVLA AR STDSDVL EIVKDALELA KQSTNEEVIK LALKAAVLAA KSTDEEVLEE VKEALRRAKE STDEEEIKEE LRKAVEEAEG GSW LEHHHH HH |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.8 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
詳細: 25mM Tris HCl, 300mM NaCl, pH 8 | |||||||||
| グリッド | モデル: Quantifoil / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 2 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 25 sec. / 前処理 - 雰囲気: AIR / 前処理 - 気圧: 0.039 kPa | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295.15 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 0.9 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: Other / Chain - Initial model type: in silico model / 詳細: De Novo Design |
|---|---|
| 詳細 | Model was initially fit in Chimera. Model was further refined with Namdinator, Isolde, and Coot |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 955.4 |
| 得られたモデル |  PDB-8v2d: |
 ムービー
ムービー コントローラー
コントローラー























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)