+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
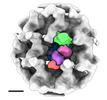
| タイトル | Intracellular Ebola nucleocapsid-like structure obtained from cells expressing NP, VP24 and VP35 at 17.6 angstrom | |||||||||
 マップデータ マップデータ | intracellular Ebola virus nucelocapsid from cells expressing NP, VP24 and VP35 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | VIRAL PROTEIN / nucleoprotein / nucleocapsid / Ebola virus / EBOV / filovirus / subtomogram averaging / cryo-ET / FIB / intracellular / in situ | |||||||||
| 生物種 |  | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 17.57 Å | |||||||||
 データ登録者 データ登録者 | Watanabe R / Zyla D / Saphire EO | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2024 ジャーナル: Cell / 年: 2024タイトル: Intracellular Ebola virus nucleocapsid assembly revealed by in situ cryo-electron tomography. 著者: Reika Watanabe / Dawid Zyla / Diptiben Parekh / Connor Hong / Ying Jones / Sharon L Schendel / William Wan / Guillaume Castillon / Erica Ollmann Saphire /  要旨: Filoviruses, including the Ebola and Marburg viruses, cause hemorrhagic fevers with up to 90% lethality. The viral nucleocapsid is assembled by polymerization of the nucleoprotein (NP) along the ...Filoviruses, including the Ebola and Marburg viruses, cause hemorrhagic fevers with up to 90% lethality. The viral nucleocapsid is assembled by polymerization of the nucleoprotein (NP) along the viral genome, together with the viral proteins VP24 and VP35. We employed cryo-electron tomography of cells transfected with viral proteins and infected with model Ebola virus to illuminate assembly intermediates, as well as a 9 Å map of the complete intracellular assembly. This structure reveals a previously unresolved third and outer layer of NP complexed with VP35. The intrinsically disordered region, together with the C-terminal domain of this outer layer of NP, provides the constant width between intracellular nucleocapsid bundles and likely functions as a flexible tether to the viral matrix protein in the virion. A comparison of intracellular nucleocapsids with prior in-virion nucleocapsid structures reveals that the nucleocapsid further condenses vertically in the virion. The interfaces responsible for nucleocapsid assembly are highly conserved and offer targets for broadly effective antivirals. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_42515.map.gz emd_42515.map.gz | 580.6 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-42515-v30.xml emd-42515-v30.xml emd-42515.xml emd-42515.xml | 14.5 KB 14.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_42515.png emd_42515.png | 94.7 KB | ||
| マスクデータ |  emd_42515_msk_1.map emd_42515_msk_1.map | 1.4 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-42515.cif.gz emd-42515.cif.gz | 4.4 KB | ||
| その他 |  emd_42515_half_map_1.map.gz emd_42515_half_map_1.map.gz emd_42515_half_map_2.map.gz emd_42515_half_map_2.map.gz | 1 MB 1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42515 http://ftp.pdbj.org/pub/emdb/structures/EMD-42515 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42515 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42515 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_42515_validation.pdf.gz emd_42515_validation.pdf.gz | 704.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_42515_full_validation.pdf.gz emd_42515_full_validation.pdf.gz | 704 KB | 表示 | |
| XML形式データ |  emd_42515_validation.xml.gz emd_42515_validation.xml.gz | 7.3 KB | 表示 | |
| CIF形式データ |  emd_42515_validation.cif.gz emd_42515_validation.cif.gz | 8.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42515 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42515 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42515 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-42515 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_42515.map.gz / 形式: CCP4 / 大きさ: 1.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_42515.map.gz / 形式: CCP4 / 大きさ: 1.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | intracellular Ebola virus nucelocapsid from cells expressing NP, VP24 and VP35 | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 5.37 Å | ||||||||||||||||||||||||||||||||||||
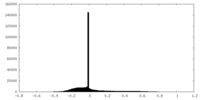
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_42515_msk_1.map emd_42515_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
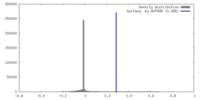
| 密度ヒストグラム |
-ハーフマップ: half map1
| ファイル | emd_42515_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
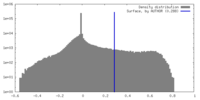
| 密度ヒストグラム |
-ハーフマップ: half map2
| ファイル | emd_42515_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
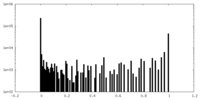
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Ebola virus - Mayinga, Zaire, 1976
| 全体 | 名称:  |
|---|---|
| 要素 |
|
-超分子 #1: Ebola virus - Mayinga, Zaire, 1976
| 超分子 | 名称: Ebola virus - Mayinga, Zaire, 1976 / タイプ: virus / ID: 1 / 親要素: 0 / NCBI-ID: 128952 / 生物種: Ebola virus - Mayinga, Zaire, 1976 / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: OTHER / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 7.2 |
|---|---|
| グリッド | モデル: Quantifoil / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE-PROPANE |
| 詳細 | FIB-milled-plunge-frozen cell expressing EBOV NP, VP24 and VP35 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 3.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 5.5 µm / 最小 デフォーカス(公称値): 4.5 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 17.57 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3.1) / 使用したサブトモグラム数: 8371 |
|---|---|
| 抽出 | トモグラム数: 10 / 使用した粒子像数: 39000 |
| 最終 3次元分類 | クラス数: 3 / 平均メンバー数/クラス: 13000 / ソフトウェア - 名称: RELION (ver. 3.1) |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION (ver. 3.1) |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)